В банке pdb найдем на странице SMILES нотацию для NAG.
Это удобно сделать на странице структуры 1lmp.
obgen nag.smi > nag.mol babel -imol nag.mol -opdb nag.pdb
Про использование prepare_ligand4.py узнать можно запустив скрипт с флагом -h prepare_ligand4.py -l nag.pdb
prepare_receptor4.py -r model.pdb
Для докинга необходимо указать область структуры белка, в которой будет происходить поиск места для связывания. Удобно его задать как куб с неким центором. Координаты центра определяем из полученной ранее модели комплекса.
Для этого выберем атом сахара, который находится в центре сайта связывания, и из текста pdb файла найдем его координаты. HETATM 1031 C2B NAG B 128 41.533 42.665 27.389 1.00157.52 CПостроим файл vina.cfg с примерно таким содержанием:
center_x=41.533 center_y=42.665 center_z=27.389 size_x = 25 size_y = 25 size_z = 25 num_modes = 20
vina --config vina.cfg --receptor model.pdbqt --ligand nag.pdbqt --out nag_prot.pdbqt --log nag_prot.log
| Расположение | Энергия (ккал/моль) | Геометрическая разница с лучшей моделью (rmsd l.b.) |
| 1 | -5.4 | 0.000 (best mode 0.000) |
| 7 | -5.0 | 1.876 (best mode 2.194) |
| 3 | -5.3 | 1.826 (best mode 2.978) |
В PyMol загрузим файлы nag_prot.pdbqt и model.pdbqt. Просмотрим их с анимацией.
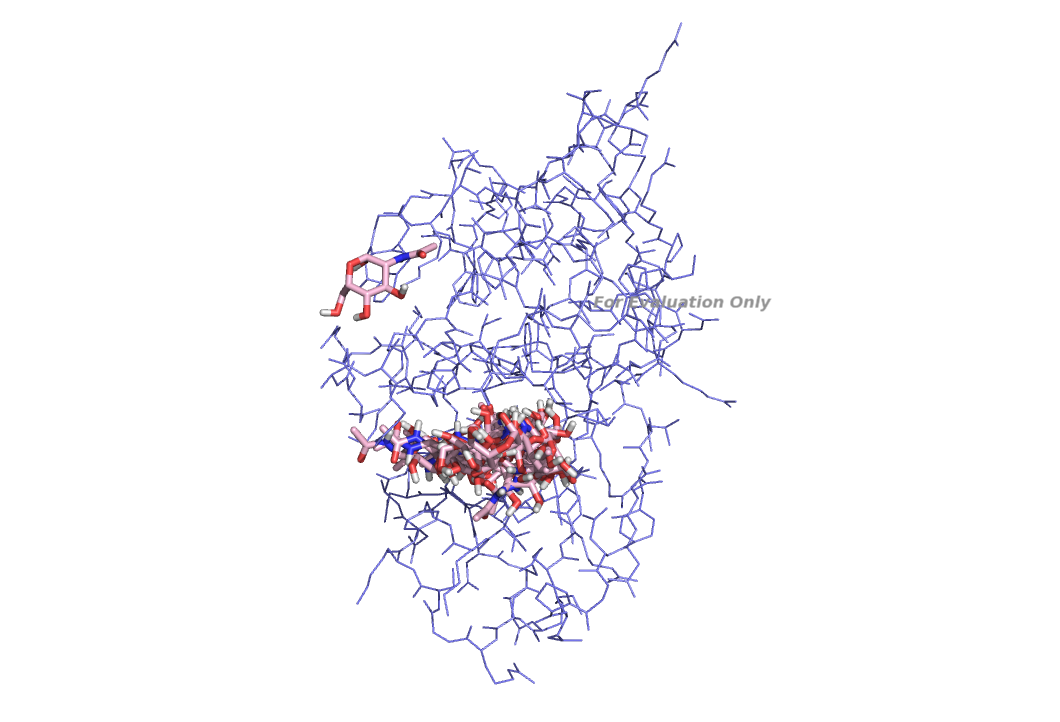 |  |
Видим, что состояния впринципе разные. Состояние лиганда сильно меняет свое положение. Так же есть одно выбивающиеся состояние - не в центре взаимодействия.
prepare_flexreceptor4.py -r model.pdbqt -s ASN59_PRO107_VAL109и проведём докинг:
vina --config vina.cfg --receptor model_rigid.pdbqt --flex model_flex.pdbqt --ligand nag.pdbqt --out vina_model_flex.pdbqt --log vina_model_flex.log
| Расположение | Энергия (ккал/моль) | Геометрическая разница с лучшей моделью (rmsd l.b.) |
| 1 | -4.9 | 0.000 (best mode 0.000) |
| 6 | -4.4 | 1.542 (best mode 2.079) |
| 14 | -3.5 | 1.789 (best mode 2.268) |
| 7 | -4.3 | 2.568 (best mode 5.444) |
| 3 | -4.8 | 2.138 (best mode 3.889) |
Время затраченное на этот докинг больше, по сравнению с прошлым, однако энергии полученных моделей меньше и геометрическая разница меньше.
В PyMol загрузим файлы vina_model_flex.pdbqt и model_rigid.pdbqt. Просмотрим полученный ролик.
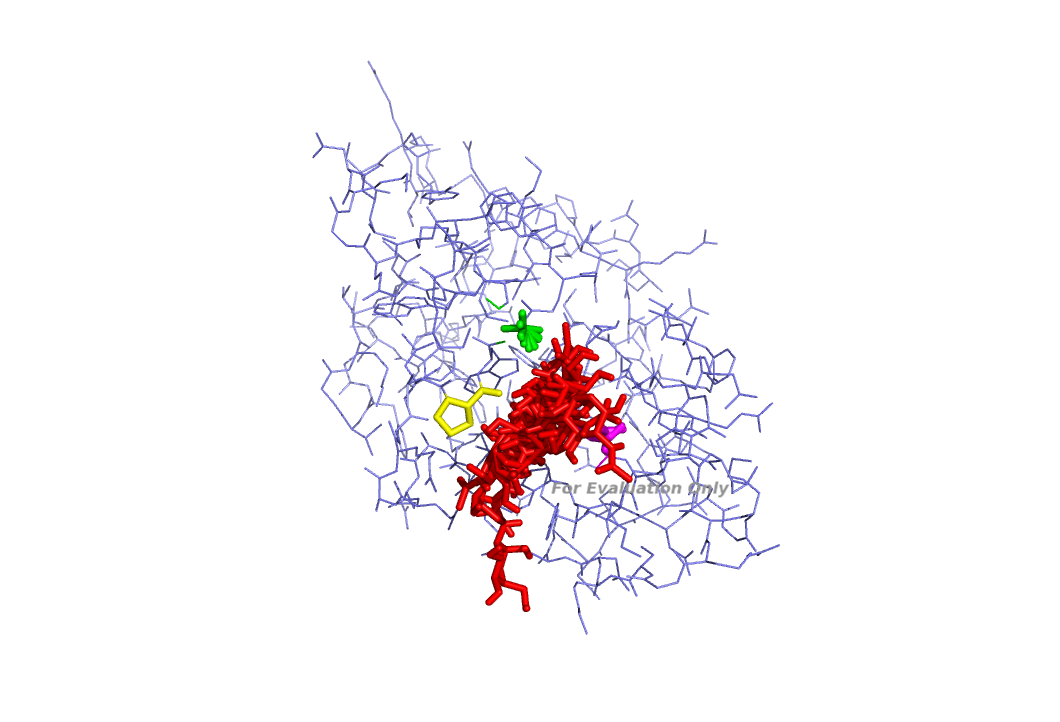 | 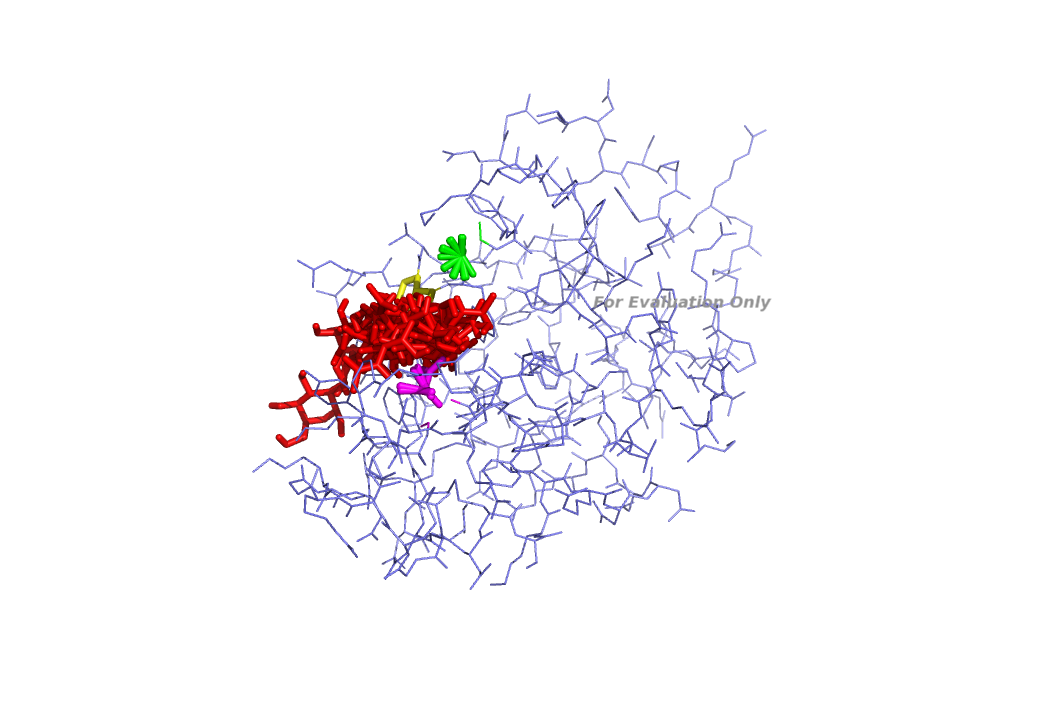 |
По полученным изображениям видно, что лиганд по прежнему меняет свое положение, так же меняют положение выбранные остатки, но это изменение не сильно касается PRO107 - у него стационарное положение. Вообще, изменение положение лиганда становится более обширным. Такой докинг является более правильным с биологической точки зрения, так как известно в биологии, что некоторые остатки в белке меняют свое положение при взаимодействии с лигандами.
Для каждого из этих лигандов проведем обыкновенный докинг и с подвижными радикалами:
| Смайл структуры: OH NH2 H |
pdbqt файлы для: OH NH2 H | |
| Файлы обычного докинга для OH: nag1_model.log nag1_model.pdbqt model.pdbqt |
Файлы обычного докинга для NH2: nag2_model.log nag2_model.pdbqt model.pdbqt |
Файлы обычного докинга для H: nag3_model.log nag3_model.pdbqt model.pdbqt |
| Файлы подвижного докинга для OH: vina_model1_flex.log vina_model1_flex.pdbqt model_rigid.pdbqt |
Файлы подвижного докинга для NH2: vina_model2_flex.log vina_model2_flex.pdbqt model_rigid.pdbqt |
Файлы подвижного докинга для H: vina_model3_flex.log vina_model3_flex.pdbqt model_rigid.pdbqt |
Запишем энергии 3ёх лучших расположений и геометрическую разницу между ними для каждого лиганда в 2х докингах и рассмотрим полученные модели.
Для OH:
| Вид докинга | Расположение | Энергия (ккал/моль) | Геометрическая разница с лучшей моделью (rmsd l.b.) |
| обычный | 1 | -4.7 | 0.000 (best mode 0.000) |
| обычный | 6 | -4.4 | 1.113 (best mode 1.659) |
| обычный | 3 | -4.5 | 1.618 (best mode 2.596) |
| подвижный | 1 | -4.3 | 0.000 (best mode 0.000) |
| подвижный | 5 | -3.9 | 1.017 (best mode 1.823) |
| подвижный | 11 | -3.4 | 1.750 (best mode 2.169) |
Полученные модели для обычного докинга:
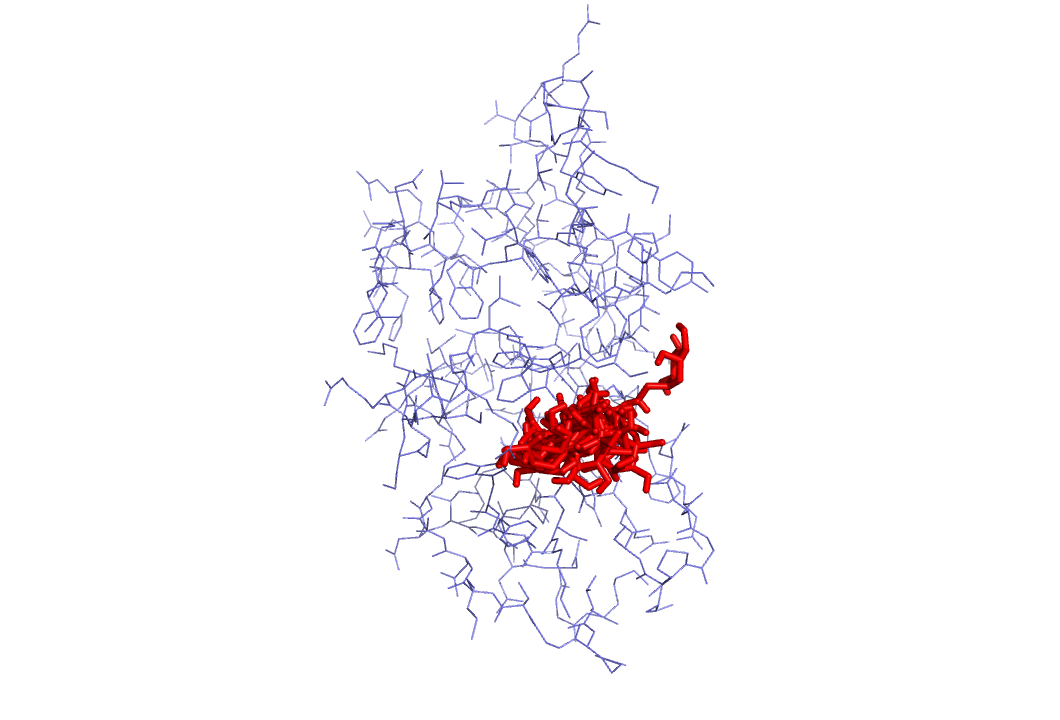 | 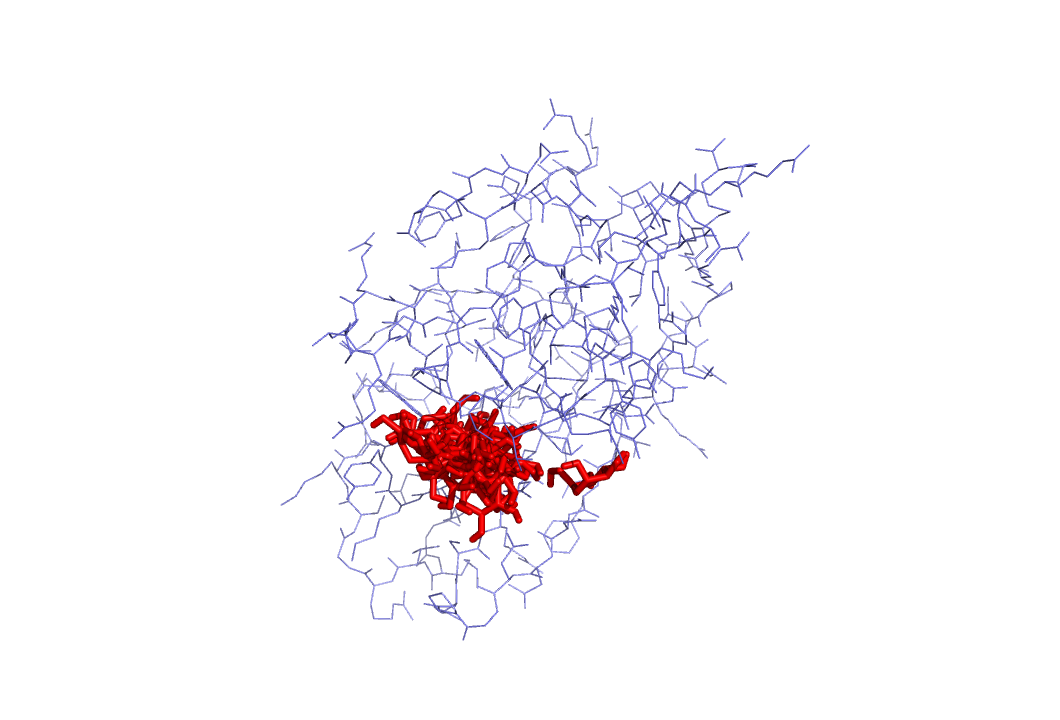 |
Полученные модели для подвижного докинга:
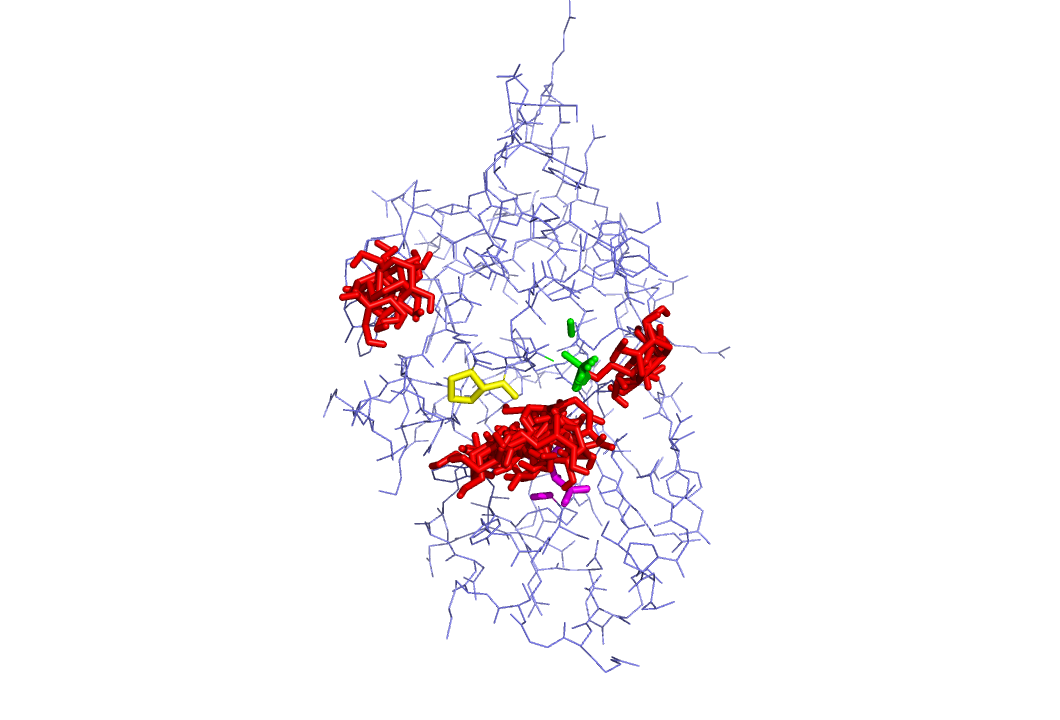 | 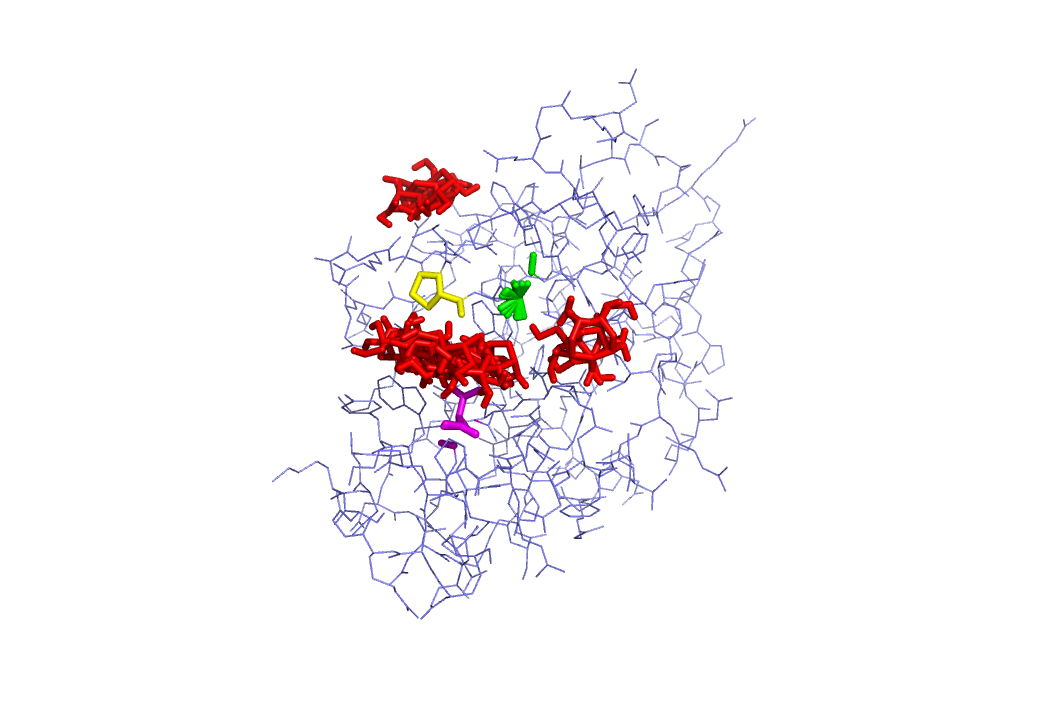 |
Для NH2:
| Вид докинга | Расположение | Энергия (ккал/моль) | Геометрическая разница с лучшей моделью (rmsd l.b.) |
| обычный | 1 | -4.7 | 0.000 (best mode 0.000) |
| обычный | 3 | -4.5 | 1.852 (best mode 2.605) |
| обычный | 4 | -4.4 | 1.773 (best mode 2.999) |
| подвижный | 1 | -4.3 | 0.000 (best mode 0.000) |
| подвижный | 6 | -4.0 | 1.343 (best mode 2.744) |
| подвижный | 18 | -3.0 | 1.994 (best mode 2.883) |
Полученные модели для обычного докинга:
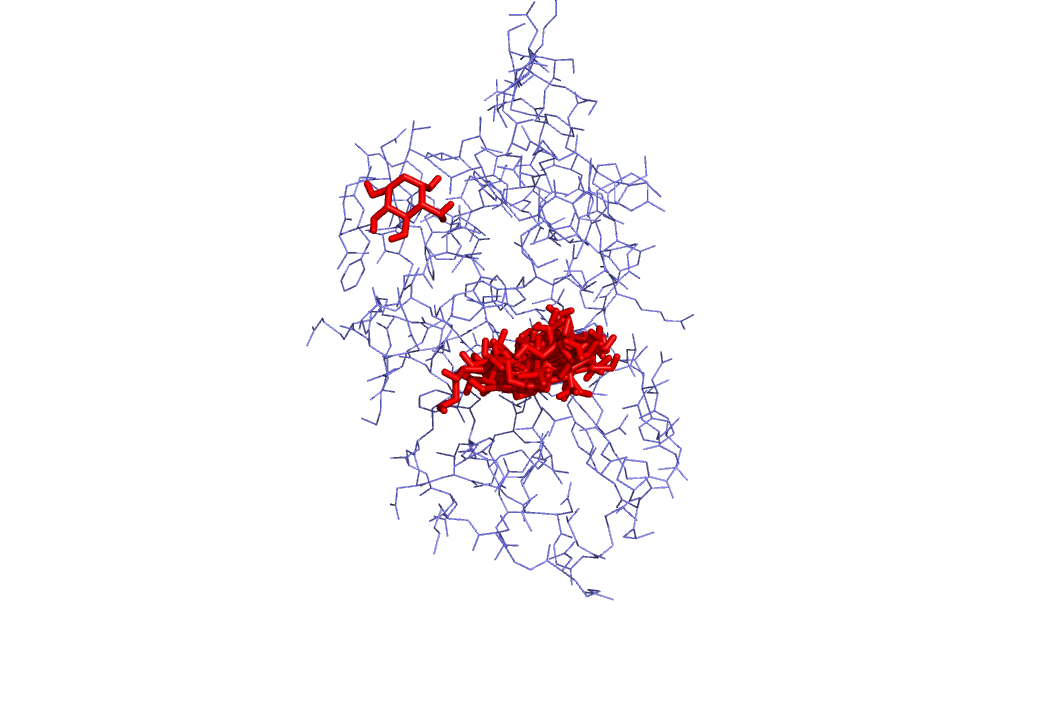 |  |
Полученные модели для подвижного докинга:
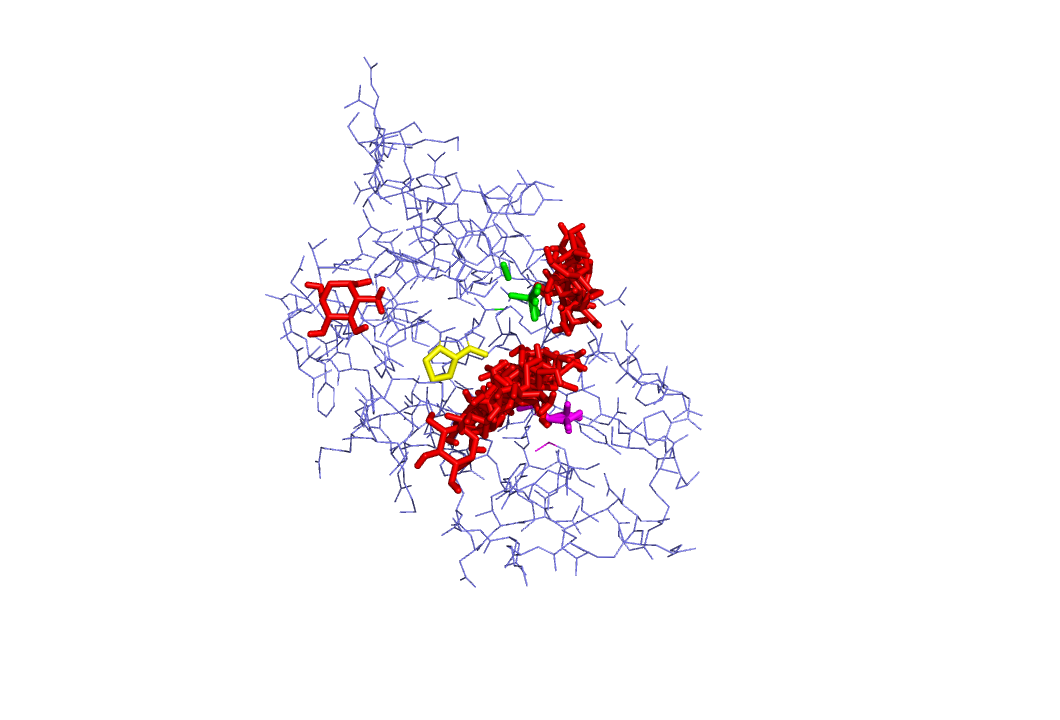 | 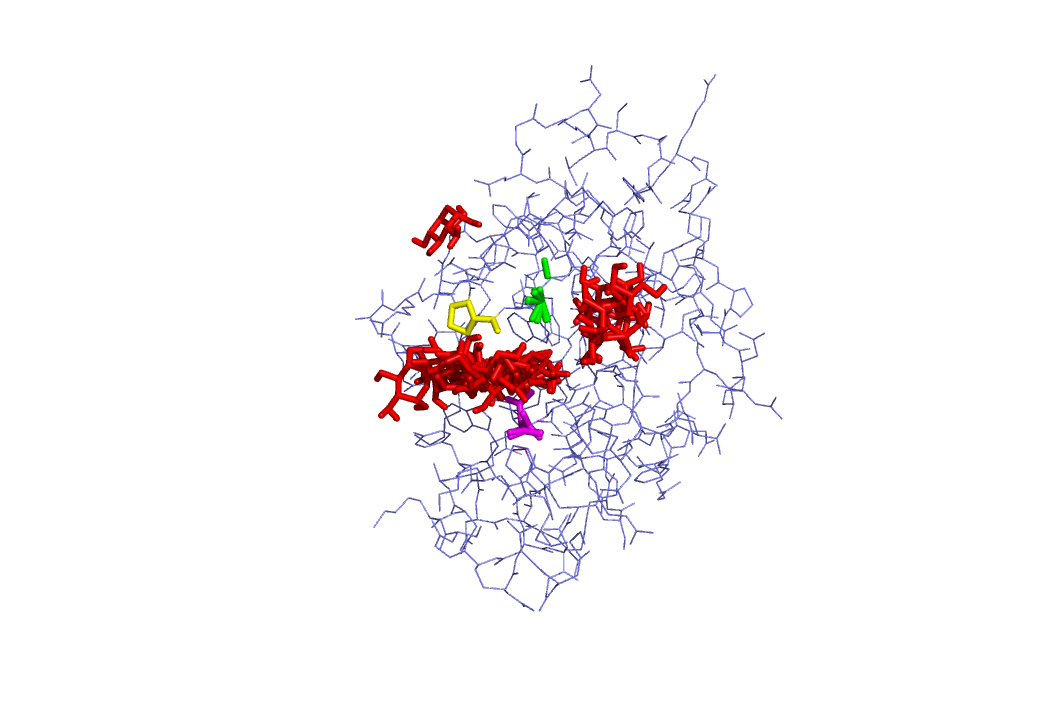 |
Аналогичная ситуация с прошлым разом. Однако стоит заметить, что вылезание лиганда из активного сайта происходит в меньшем количестве моделей. Возможно это связано с тем, что группа NH2 важна для взаимодейстивия сахара с белком.
Для H:
| Вид докинга | Расположение | Энергия (ккал/моль) | Геометрическая разница с лучшей моделью (rmsd l.b.) |
| обычный | 1 | -4.8 | 0.000 (best mode 0.000) |
| обычный | 7 | -4.3 | 1.574 (best mode 1.867) |
| обычный | 16 | -3.8 | 1.832 (best mode 2.117) |
| подвижный | 1 | -4.1 | 0.000 (best mode 0.000) |
| подвижный | 10 | -3.5 | 1.592 (best mode 2.127) |
| подвижный | 8 | -3.7 | 0.988 (best mode 2.403) |
Полученные модели для обычного докинга:
 |  |
Полученные модели для подвижного докинга:
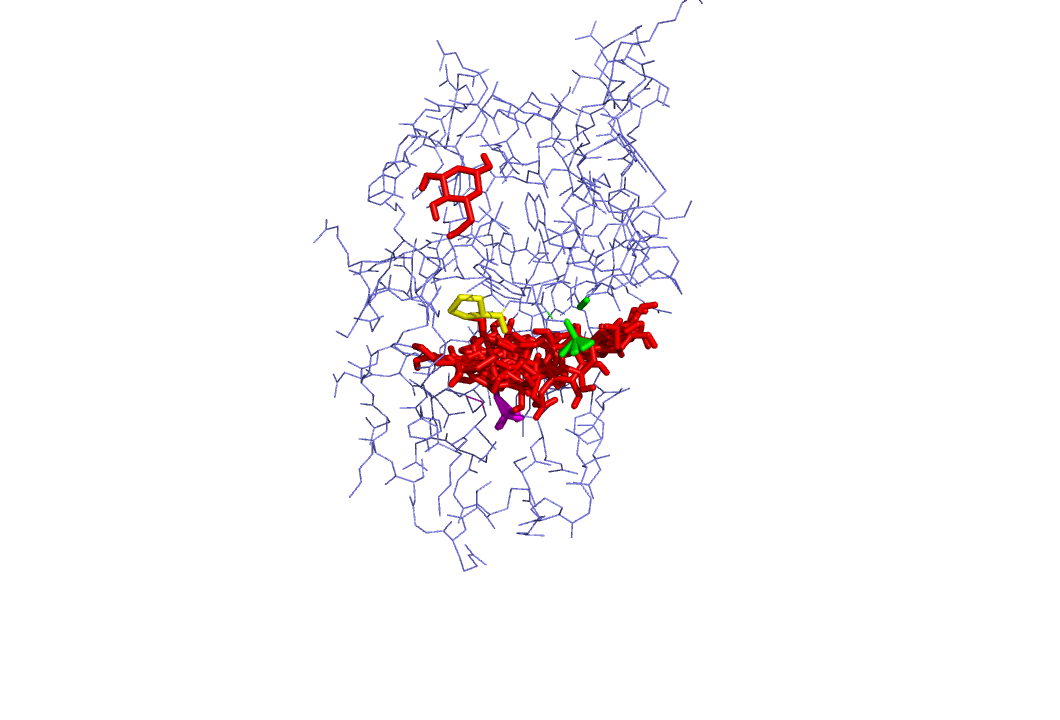 | 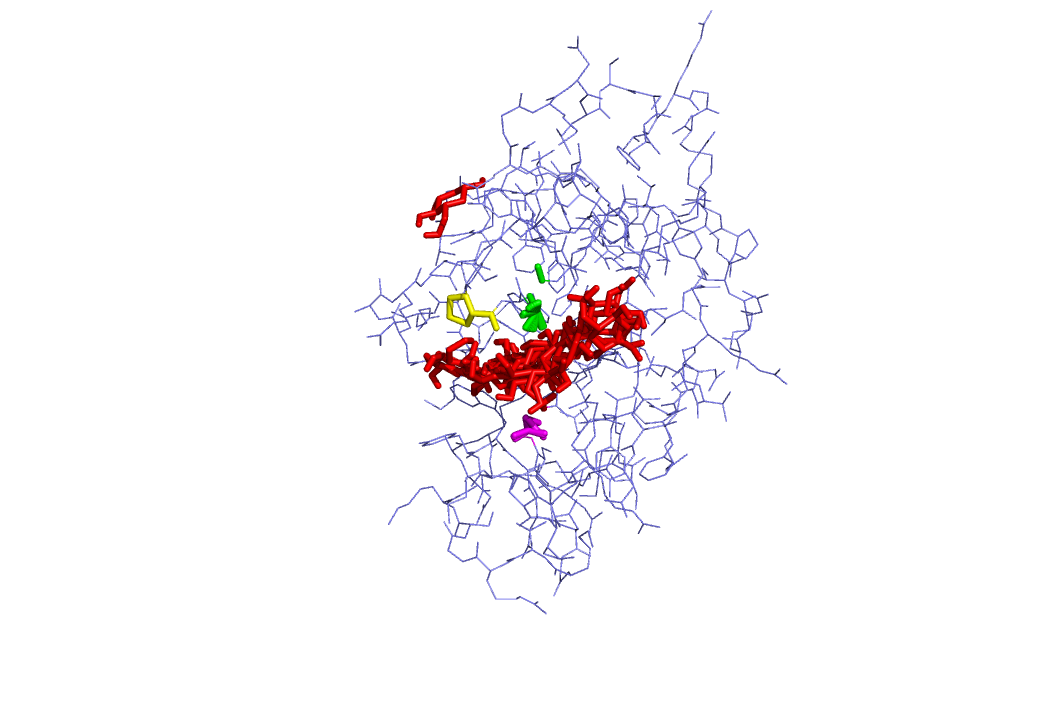 |
В случае водорода видим, что лиганд практически всегда остается в сайте связывания.