"Исследование структуры тРНК"
- Краткое описание структуры в файле 1g59.pdb
В файле приведены координаты атомов следующих молекул
глутамил-тРНК синтетазы и тРНК(глутаминовая кислота) из Escherichia coli. Научное название Thermus thermophilus.
Для исследования была выбрана цепь D, представляющая глутамил-тРНК со следующей последовательностью:
[501] 5'- G G C C C C A U C G U C U A G C G G U U A G G A C G C G G C C C U C U C A A G G C C G A A A C G G G G G U U C G A U U C C C C C U G G G G U C A C C A - 3' [576]
В последовательности на 3'-конце имеется триплет CCA, к которому присоединяется аминокислота глутаминовая кислота. Координаты его атомов с 3134 по 3196. - Исследование вторичной структуры
С помощью программы find_pair пакета 3DNA были определены возможные водородные связи
между азотистыми основаниями (1G59_Old.out). Используя полученные данные,
а также схему строения тРНК (статья из соровского журнала):
RMSD of the bases (----- for WC bp, + for isolated bp, x for helix change) Strand I Strand II Helix 1 (0.009) D:.501_:[..G]G-----C[..C]:.572_:D (0.010) | 2 (0.010) D:.502_:[..G]G-*---U[..U]:.571_:D (0.016) | 3 (0.010) D:.503_:[..C]C-----G[..G]:.570_:D (0.008) | 4 (0.009) D:.504_:[..C]C-----G[..G]:.569_:D (0.006) | 5 (0.009) D:.505_:[..C]C-----G[..G]:.568_:D (0.009) | 6 (0.008) D:.506_:[..C]C-----G[..G]:.567_:D (0.008) | 7 (0.009) D:.507_:[..A]Ax----U[..U]:.566_:D (0.015) | 8 (0.011) D:.549_:[..G]G-----C[..C]:.565_:D (0.012) | 9 (0.008) D:.550_:[..G]G-----C[..C]:.564_:D (0.010) | 10 (0.009) D:.551_:[..G]G-----C[..C]:.563_:D (0.014) | 11 (0.007) D:.552_:[..G]G-----C[..C]:.562_:D (0.011) | 12 (0.011) D:.553_:[..G]G----xC[..C]:.561_:D (0.009) | 13 (0.017) D:.554_:[..U]U-*--xA[..A]:.558_:D (0.008) | 14 (0.019) D:.555_:[..U]Ux**+xG[..G]:.517_:D (0.022) x 15 (0.011) D:.538_:[..A]A-*---C[..C]:.532_:D (0.013) | 16 (0.013) D:.539_:[..G]G-----C[..C]:.531_:D (0.008) | 17 (0.008) D:.540_:[..G]G-----C[..C]:.530_:D (0.009) | 18 (0.010) D:.541_:[..C]C-----G[..G]:.529_:D (0.008) | 19 (0.010) D:.542_:[..C]C-----G[..G]:.528_:D (0.006) | 20 (0.009) D:.543_:[..G]G-----C[..C]:.527_:D (0.012) | 21 (0.009) D:.544_:[..A]Ax*---G[..G]:.526_:D (0.009) | 22 (0.010) D:.510_:[..G]G-----C[..C]:.525_:D (0.012) | 23 (0.018) D:.511_:[..U]U-----A[..A]:.524_:D (0.009) | 24 (0.011) D:.512_:[..C]C-----G[..G]:.523_:D (0.008) | 25 (0.016) D:.513_:[..U]U-*--xG[..G]:.522_:D (0.007) | 26 (0.010) D:.514_:[..A]A-**-xU[..U]:.508_:D (0.015) | 27 (0.010) D:.515_:[..G]Gx**+xC[..C]:.548_:D (0.008) x 28 (0.012) D:.518_:[..G]G-----C[..C]:.556_:D (0.032) +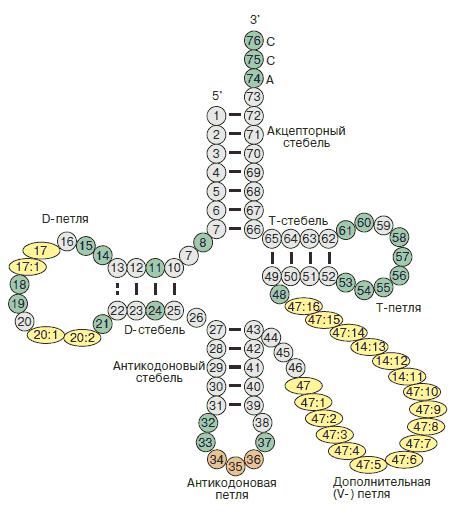
Делаем вывод, что акцепторный стебель состоит из участка 501-507 и комплементарного ему участка 566-572.
Т-стебель из 549-552 и 562-565
D-стебель из 510-513 и 522-525
антикодоновый стебель из 539-543 и 527-531
Структуру стеблевых дуплексов поддерживают 19 канонических и 1 (G502-U571) неканоническая пара оснований.Рис.1. Вторичная структура глутамил-тРНК из Escherichia coli
(ввиде остовной модели, где акцепторный стебель выделен красным, Т-стебель - зеленым, D-стебель - синим, антикодоновый - оранжевым.)
Скрипт для получения изображения: background white restrict none select *D wireframe off cpk off backbone 100 define stem_acceptor 501-507, 566-572 define stem_t 549-552, 562-565 define stem_d 510-513, 522-525 define stem_anticodon 539-543, 527-531 select stem_acceptor color red select stem_t color green select stem_d color blue select stem_anticodon color orange select not (stem_acceptor, stem_t, stem_d, stem_anticodon) color grey
Неканоническая пара G502 и U571.
Так же из этих данных узнаем, что в моей тРНК присутствует вариабельная петля (нуклеотиды с 544 до 548), хотя по внешнему виду она больше похожа на связку:
вариабельная петля черного цвета
Так же можно отметить отсутствие остатка тимидина в Т-петле и дигидроуридина в D-петле
*Дополнительное задание.
антикодон в антикодоновой петле.
Глутаминовую кислоту кодируют кодоны GAA и GAG. Соответственно антикодоны UUC и CUC. На 534-536 местах антикодоновой петли располагается антикодон CUC. Покажем эти нуклеотиды в шарнирной модели.
- Исследование третичной структуры
1.Исследование возможности стекинг-взаимодействия между основаниями конца акцепторно стебля и начала Т-стебля.
Стекинг-взаимодействиями называются взаимодействия между комплементарными парами оснований, лежащих друг за другом, образуя "стопки". Стекинг-взаимодействия имеют две основных составляющие: гидрофобные взаимодействия и связь пи-облаков.
С помощью программы analyze обнаружила 27 возможных стекинг-взаимодействия. Данные получаем из файла.
Рассмотрим водородные связи в интересующем нас участке:7 (0.009) D:.507_:[..A]Ax----U[..U]:.566_:D (0.015) | 8 (0.011) D:.549_:[..G]G-----C[..C]:.565_:D (0.012) |
Видим, что нас интересуют 2 взаимодействия A507-G549 и U566-D565. Найдем их в стекинг-взаимодействиях.step i1-i2 i1-j2 j1-i2 j1-j2 sum 7 AG/CU 0.00( 0.00) 0.00( 0.00) 0.14( 0.00) 1.67( 0.48) 1.81( 0.48)Используя программы stack2img и Ghost, получаю изображение:
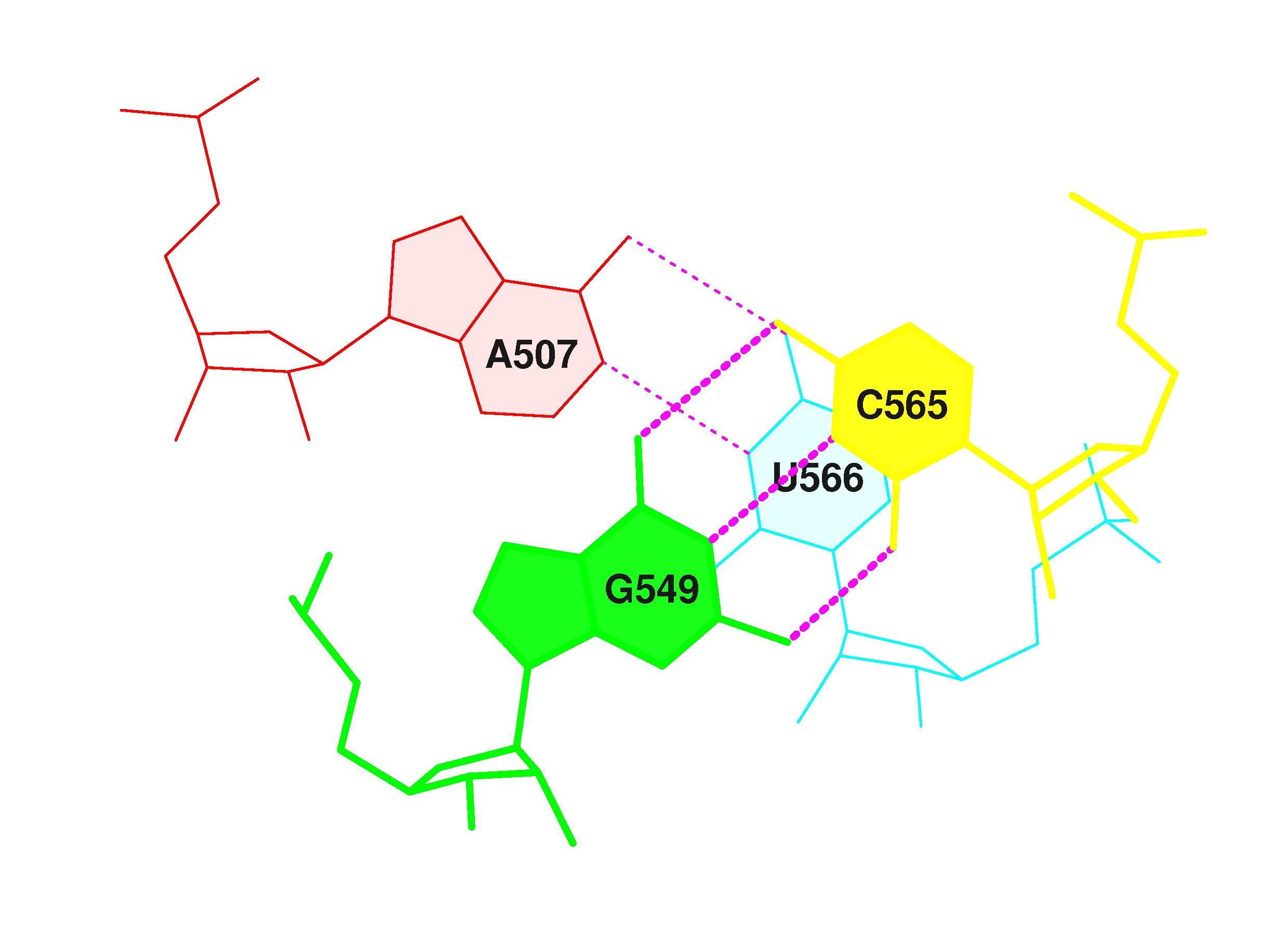
Из полученного изображения заметно, что нуклеотиды очень слабо перекрываются. Данные это подтверждают: площадь перекрывания равна 1.81 квадратный ангстрем. Площадь этого перекрывания существенно мала, поэтому можно предположить, что такое стекинг-взаимодействие маловероятно существует реально.
2. Рассмотрим возможность существования дополнительных водородных связей между основаниями D- и Т-петель. Между ними существует 2 водородные связи (U555-G517 неканоническое и G518-C556 каноническое взаимодействия):14 (0.019) D:.555_:[..U]Ux**+xG[..G]:.517_:D (0.022) x 28 (0.012) D:.518_:[..G]G-----C[..C]:.556_:D (0.032) +
Каноническое взаимодействие G517-U555:
Неканоническое взаимодействие G518-C556:
- Предсказание вторичной структуры тРНК
Предсказание вторичной структуры тРНК путем поиска инвертированных повторов.
Используем программу einverted из пакета EMBOSS, позволяющую найти инвертированные участки в нуклеотидных последовательностях. Программа базируется на "выравнивании" нуклеотидов относительно комплементраных, по параметрам выравниваются разные части тРНК. Наилучший результат, на мой взгляд, показан при штрафе за гэп равном 12, мин.весе выравнивания 2, весе совпадений 5 и весе несовпадения -5. Найдем возможные комплементарные участки в последовательности исследуемой тРНК, а затем сравним с их описанием, полученным с помощью find_pair. Результаты занесу в таблицу.Предсказание вторичной структуры тРНК по алгоритму Зукера.
Программа mfold из пакета EMBOSS использует алгоритм Зукера.Использую команду mfold SEQ='1G59.fasta.txt' P=10,полученный изображения(несколько вариантов):
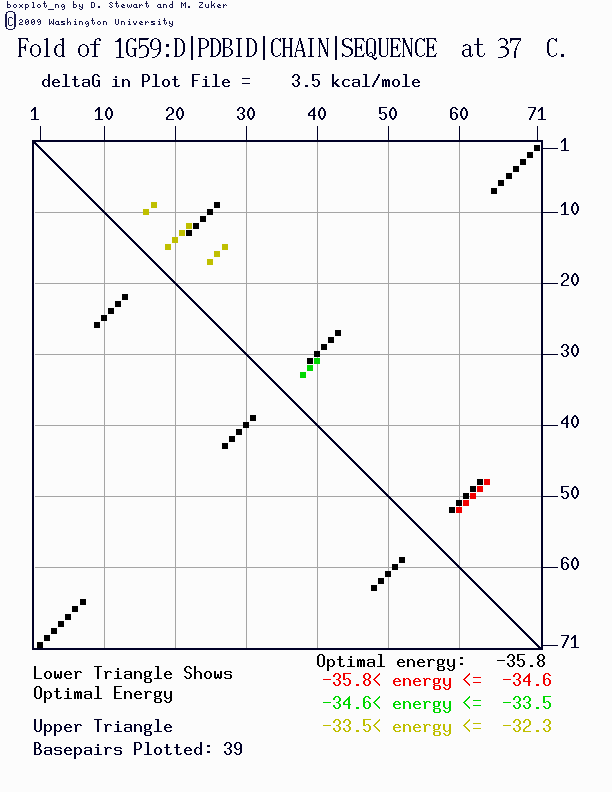
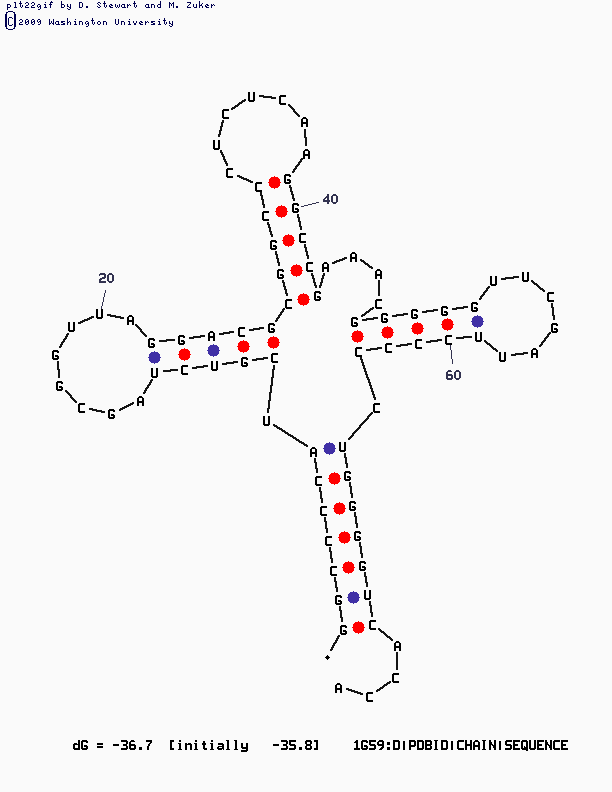
Реальная и предсказанная вторичная структура тРНК из 1g59.pdb
Очевидно, что точнее способы определения вторичной структуры тРНК с помощью программы find_pair и алгоритма Зукера. Алгоритм создает схему строения тРНК, что является более наглядным, чем список водородных связей. При помощи einverted можно предсказать часть вторичной структуры тРНК, все сильно меняется от разных параметров.Участок структуры) Позиции в структуре
(по результатам find_pair)Результаты предсказания
с помощью einvertedРезультаты предсказания
по алгоритму ЗукераАкцепторный стебель 5' 501-507 3'
5' 566-572 3'
Всего 7 парпредсказано 0 пар 7 пар D-стебель 5' 510-513 3'
5' 522-525 3'
Всего 4 парыпредсказано 0 пар 5 пар(лишняя C509-G526) T-стебель 5' 549-552 3'
5' 562-565 3'
Всего 4 парыпредсказано 0 пар 5 пар(лишняя G553-U560) Антикодоновый стебель 5' 539-543 3'
5' 527-531 3'
Всего 5 парпредсказано 0 пар 5 пар Общее число канонических пар нуклеотидов 19 17 18