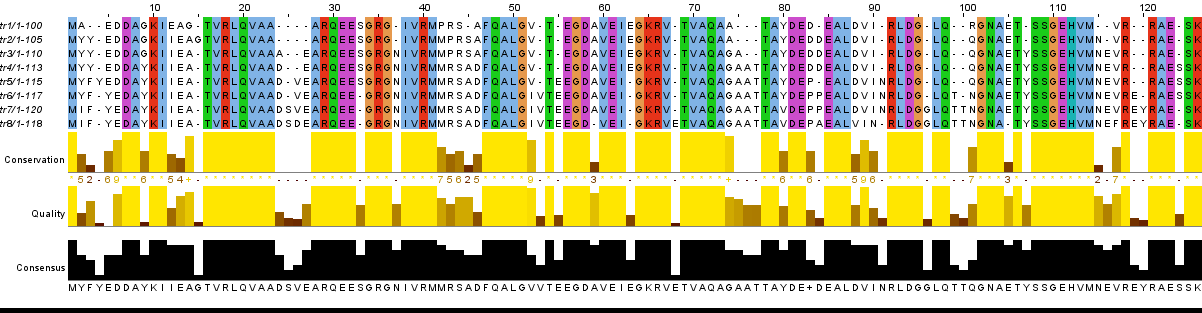
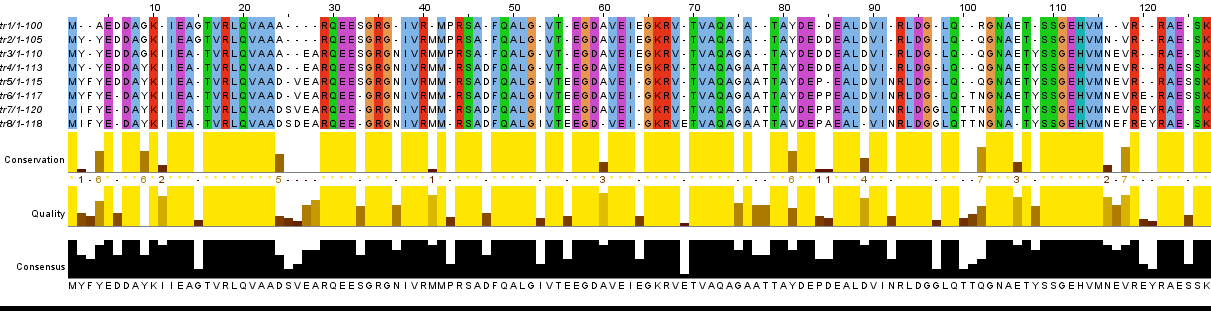
Ниже представлено выравнивание шести последовательностей белков семейства HSP70 (семейство белков теплого шока, взаимодействующих с синтезируемой на рибосомах полипептидной цепью, предотвращая преждевременное неправильное сворачивание незрелой полипептидной цепи, и участвующих в транспорте белка к органеллам) по алгоритму ТcoffeeWS из Eukaryota, Bacteria и Archea (точные наименования - в таблице 1). Окрашивание произведено по схеме ClustalX с условием Identity Threshold = 100%. Предложенная цветовая схема совершает окрашивание столбцов только со 100% идентичными аминокислотными остатками в каждой последовательности.
Таблица 1: выбранные для задания белки.
| Запись | Имя белка | Домен | Оргнанизм |
| A7IC65 | Chaperone protein DnaK | Bacteria | Xanthobacter autotrophicus |
| Q85FW4 | Chaperone protein dnaK | Eukaryota | Cyanidioschyzon merolae |
| B0R3H4 | Chaperone protein DnaK | Archaea | Halobacterium salinarum |
| Q1GKS3 | Chaperone protein DnaK | Bacteria | Ruegeria sp |
| Q9I8F9 | Heat shock 70 kDa protein 1 | Eukaryota | Oryzias latipes |
| A0B747 | Chaperone protein DnaK | Archaea | Methanosaeta thermophila |
Рис 1. Полное выравнивание

Данные в таблицах ниже полученны с помощью программы infoalign пакета EMBOSS
Таблица 2: Данные о выравнивании при параметре plurality 100.0
| Имя последовательности | Длина последовательности | Длина выравнивания | Количество гэпов | Длина гэпов | Идентичные | Сходные | Процент гэпов |
| A7IC65|DNAK_XANP2 | 631 | 684 | 15 | 53 | 496 | 42 | 27.49 |
| Q85FW4|DNAK_CYAM1 | 607 | 684 | 21 | 77 | 409 | 90 | 40.20 |
| B0R3H4|DNAK_HALS3 | 629 | 684 | 15 | 55 | 379 | 99 | 44.59 |
| Q1GKS3|DNAK_RUEST | 642 | 684 | 17 | 42 | 488 | 50 | 28.65 |
| Q9I8F9|HSP71_ORYLA | 639 | 684 | 8 | 45 | 358 | 94 | 47.66 |
| A0B747|DNAK_METTP | 615 | 684 | 14 | 69 | 404 | 93 | 40.94 |