Ниже представлена таблица комманд, использованных в этой части задания.
| Функция | Комманда | ||||||
| Выдает информацию о качестве прочтений (Quality Score) | fastqc chr51.fastq | ||||||
| Выдает файл, где отрезаны с концов прочтений нуклеотиды качеством < 20 и удалены чтения длиной < 50 | java -jar /usr/share/java/trimmomatic.jar SE -phred33 chr5.fastq chr51.fastq LEADING:20 TRAILING:20 MINLEN:50 |
| До - 8208 ридов | После - 8114 ридов |
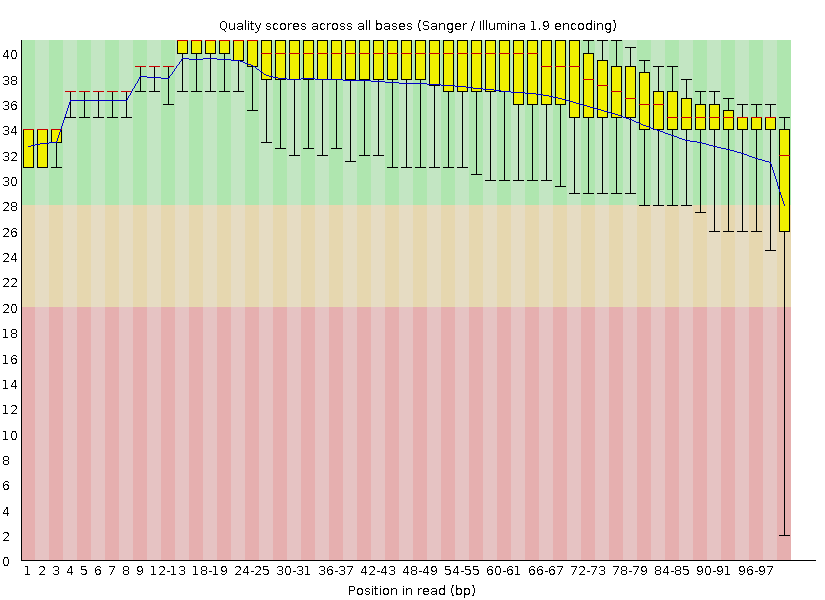 |
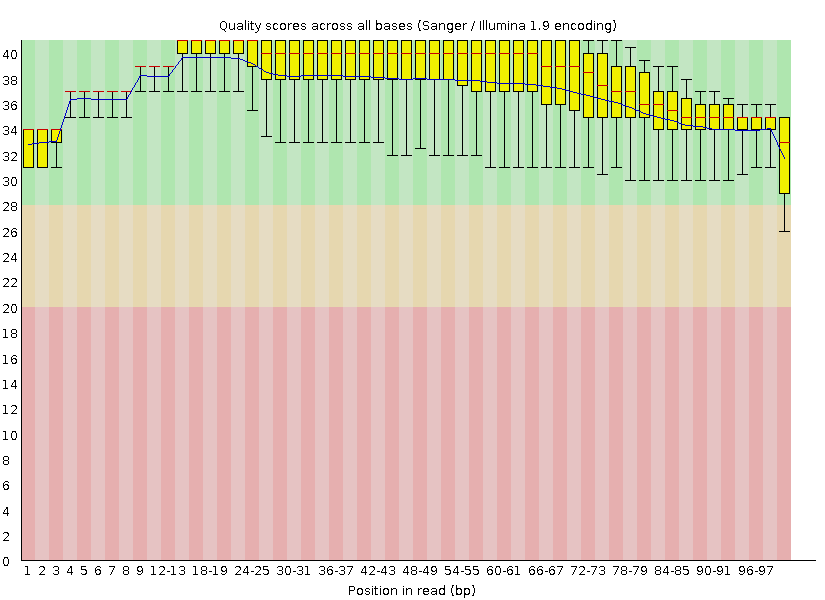 |
Чтобы объяснить, какие прочтения отсеялись и почему воспользуемся приведенными выше графиками, рассматривая каждый столбец (диаграмму размахов). Вследствие отбрасывания ридов с длиной меньше 50, а также отрезания концевых нуклеотидов с низким качеством прочтения уменьшился разброс по квартилям, сократился и сам интерквартильный размах. Также отбрасование потенциально вносящих шум слишком коротких ридов и "плохо" прочтенных (т.е. с низким качеством) нуклеотидов позволило уменьшить разброс качества прочтения концевых участков (укорочение т.н. "усов"), и обрезало последний столбец так, что после чистки весь он находится в зеленой зоне качества (Q > 28).
| Per sequence quality scores | Sequence Length Distribution | |
| До | 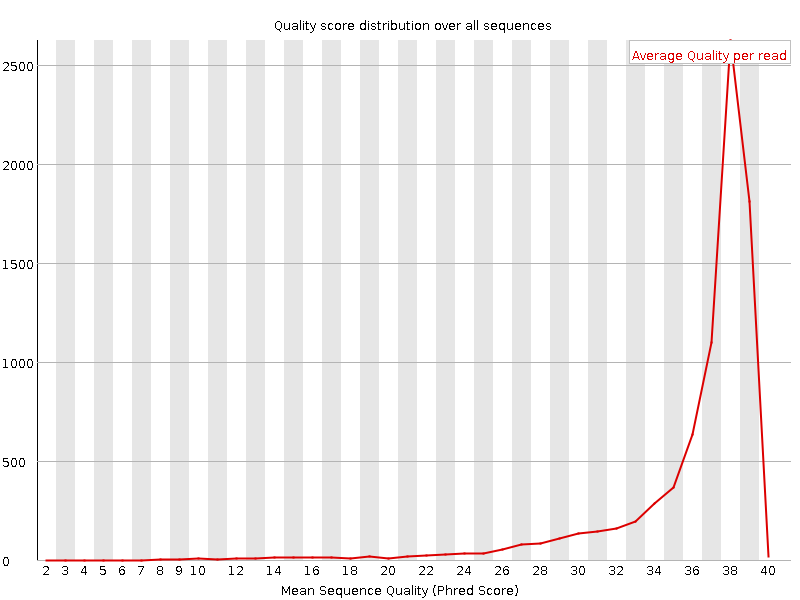 |
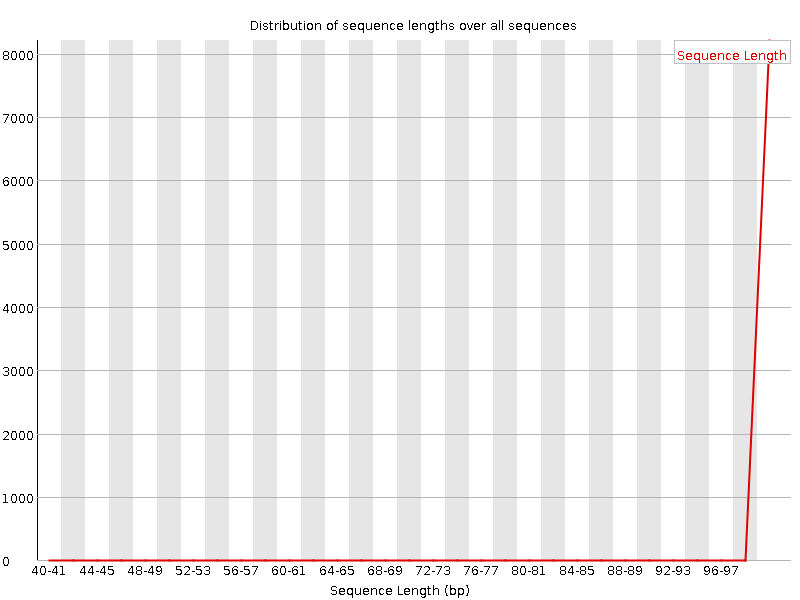 |
| После | 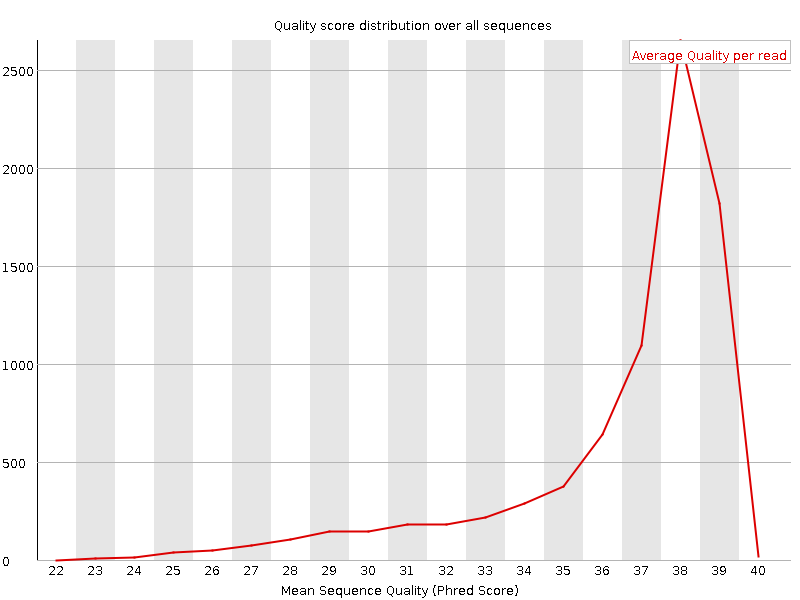 |
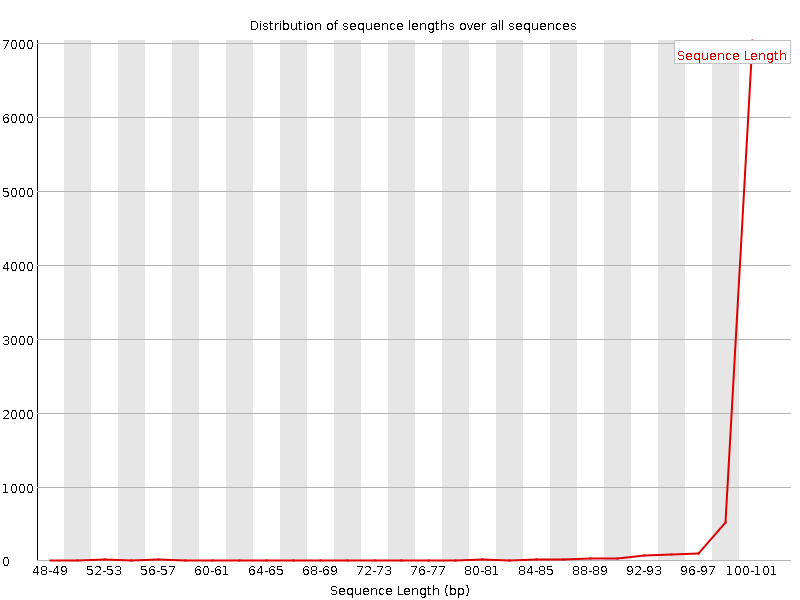 |
На изображениях в таблице выше также четко видны произощедшие с выборкой изменения. Разброс длины и качества заметно сократился (сразу ясно, при взгляде на оси абсцисс), что говорит об отсеве слишком коротких ридов, которые потенциально могут внести ошибки вследствие попадания в выборку димеров, а также о повышении среднего показателя качества по выборке.
Таблица комманд, использованных в этой части задания.
| Цель | Комманда |
| Экспорт Hisat2 | export PATH=${PATH}:/home/students/y06/anastaisha_w/hisat2-2.0.5 |
| Индексирование референсной последовательности | hisat2-build chr5.fasta chr5 |
| Выравнивание прочтений и референса в формате .sam | hisat2 --no-spliced-alignment --no-softclip -x chr5 -U chr51.fastq -S ali.sam |
| Перевод в бинарный формат | samtools view ali.sam -b -o ali.bam |
| Сортировка по координате начала в референсе | samtools sort ali.bam -T file.txt -o alisort.bam |
| Индексация отсортированного .bam файла | samtools index alisort.bam |
| Информация о количестве покрытий каждого нуклеотида | samtools depth alisort.bam >depth.tsv |
| Поиск количества покрытий по выбранному экзону | samtools depth alisort.bam -r chr5:35856951-35857161 >depth1.tsv |
После выполнения операции hisat2 --no-spliced-alignment --no-softclip -x chr5 -U chr51.fastq -S ali.sam (Выравнивание прочтений и референса в формате .sam) в соответствующий файл (здесь - ali.sam) попадает следующая информация: ID каждого чтения, координата в хромосоме и номер этой хромосомы, расстояние до генома, количество картирований и т.д.
По запрашиваемым параметрам: 8084 прочтения были выравнены единожды, 30 - ни разу. Ни одно прочтение не было выравнено более одного раза.
Выбранный нуклеотид - 35857090
Ген - IL7R (Homo sapiens interleukin 7 receptor (IL7R), transcript variant 2, non-coding RNA.)
Расположение - chr5:35,856,951-35,879,705
Экзон - chr5:35856951-35857161
Среднее покрытие экзона: 76,58768
Покрытие неравномерное.
Таблица комманд, использованных в этой части задания.
| Цель | Комманда |
| Создание файла с полиморфизмами в формате .bcf | samtools mpileup -uf chr5.fasta alisort.bam -o snp.bcf |
| Создание файла со списком отличий между референсом и чтениями в формате .vcf | bcftools call -cv snp.bcf -o snp.vcf |
| Создание файла для работы annovar | perl /nfs/srv/databases/annovar/convert2annovar.pl -format vcf4 snp1.vcf> snp1.annovar |
| Аннотация полученных snp по базе данных refgene | perl /nfs/srv/databases/annovar/annotate_variation.pl -out res.ref -build hg19 snp1.annovar /nfs/srv/databases/annovar/humandb/ |
| Аннотация полученных snp по базе данных dbsnp | perl /nfs/srv/databases/annovar/annotate_variation.pl -filter -out res.snp -build hg19 -dbtype snp138 snp1.annovar /nfs/srv/databases/annovar/humandb/ |
| Аннотация полученных snp по базе данных 1000 genomes | perl /nfs/srv/databases/annovar/annotate_variation.pl -filter -dbtype 1000g2014oct_all -out res.gen -buildver hg19 snp1.annovar /nfs/srv/databases/annovar/humandb/ |
| Аннотация полученных snp по базе данных Gwas | perl /nfs/srv/databases/annovar/annotate_variation.pl -regionanno -dbtype gwasCatalog -out res.gwas -build hg19 snp1.annovar /nfs/srv/databases/annovar/humandb/ |
| Аннотация полученных snp по базе данных Clinvar | perl /nfs/srv/databases/annovar/annotate_variation.pl -filter -out res.clin -dbtype clinvar_20150629 -buildver hg19 snp1.annovar /nfs/srv/databases/annovar/humandb/ |
Информация о трех полиморфизмах.
| Координата полиморфизма | Тип полиморфизма | Референс | Прочтение | Глубина покрытия | Качество чтений |
| 35860780 | Замена нуклеотида | G | T | 8 | 21,0411 |
| 35861152 | Замена нуклеотида | C | G | 30 | 176,009 |
| 35921069 | Замена нуклеотида | T | C | 28 | 221,999 |
Всего было обнаружено 32 полиморфизма, из которых 4 являются инделями и 28 - snp.
У всех трех найденных полиморфизмов плохое покрытие. Качество последнего - хорошее (как у большинства полиморфизмов в данном случае), качество второго - приемлемое.
| Координата | SNP | Quality и DP | refgene | dbsnp | 1000 genomes | Gwas | Clinvar |
| 35857177 | G C | 221.999 113 | intronic IL7R hom | rs1353252 | 0.647364 | ||
| 35857235 | C G | 221.999 83 | intronic IL7R hom | rs1494561 | 0.647564 | ||
| 35857262 | A G | 221.999 58 | intronic IL7R hom | rs1353250 | 0.647564 | ||
| 35860780 | G T | 21.0411 8 | intronic IL7R het | ||||
| 35861068 | T C | 221.999 44 | exonic IL7R hom | rs1494558 | 0.59984 | Severe_combined_immunodeficiency\x2c_autosomal_recessive\x2c_T_cell-negative\x2c_B_cell-positive\x2c_NK_cell-positive|not_specified | |
| 35861152 | C G | 176.009 30 | intronic IL7R het | rs11567705 | 0.233826 | ||
| 35861268 | T G | 38.7651 2 | intronic IL7R hom | rs969129 | 0.667332 | ||
| 35867343 | T C | 184.009 32 | intronic IL7R het | rs73750058 | 0.00838658 | ||
| 35870814 | A C | 32.7667 2 | intronic IL7R hom | rs10063445 | |||
| 35871010 | C T | 126.008 47 | intronic IL7R het | rs6893892 | 0.0247604 | ||
| 35871190 | G A | 221.999 98 | exonic IL7R hom | rs1494555 | 0.666933 | Severe_combined_immunodeficiency\x2c_autosomal_recessive\x2c_T_cell-negative\x2c_B_cell-positive\x2c_NK_cell-positive|not_specified | |
| 35871463 | T C | 111.008 20 | intronic IL7R het | rs9282751 | 0.0247604 | ||
| 35873899 | C A | 3.0136 3 | intronic IL7R het | ||||
| 35874575 | C T | 225.009 164 | exonic IL7R het | rs6897932 | 0.172524 | Multiple sclerosis,Type 1 diabetes | not_specified |
| 35875593 | A T | 221.999 41 | intronic IL7R hom | rs987106 | 0.44988 | ||
| 35910419 | C T | 221.999 53 | intronic CAPSL hom | rs1445899 | 0.525359 | ||
| 35910529 | C T | 221.999 92 | exonic CAPSL hom | rs1445898 | 0.525359 | Type 1 diabetes | |
| 35921069 | T C | 221.999 28 | intronic CAPSL hom | rs6859892 | 0.859625 | ||
| 35937050 | T C | 11.3429 1 | intronic CAPSL hom | rs6890660 | 0.926917 | ||
| 74633975 | C T | 8.64911 1 | intronic HMGCR hom | ||||
| 74639269 | C T | 22.7872 2 | intronic HMGCR hom | rs149363137 | 0.0321486 | ||
| 74639546 | T C | 179.014 19 | intronic HMGCR hom | rs80116386 | 0.047524 | ||
| 74642848 | A T | 26.0177 6 | intronic HMGCR het | rs17244834 | 0.432308 | ||
| 74647886 | T C | 11.3429 1 | intronic HMGCR hom | ||||
| 74651084 | A G | 221.999 90 | intronic HMGCR hom | rs3846662 | 0.625 | LDL cholesterol,Cholesterol, total | |
| 74652326 | T G | 83.0076 10 | intronic HMGCR het | rs17244883 | 0.0247604 | ||
| 74655726 | C T | 58.0073 5 | intronic HMGCR het | rs3846663 | 0.405751 | cholesterol,Quantitative traits | |
| 74656539 | T C | 225.009 58 | UTR3 HMGCR (NM_000859:c.*372T>C, NM_001130996:c.*372T>C) het | rs12916 | 0.416134 | Cholesterol, total,LDL cholesterol |
Ответы на вопросы.
Rs имеют 24 snp
Частота: от 0,008 до 0,92
SNP попали в гены IL7R, CAPSL, HMGCR, по большей части в интроны (23 из 28, +1 на 3' некодирующем участке, отмечен как UTR3)
Сведения о клинической аннотации представлены в столбцах Gwas и Clinvar.
По базе данных GWAS 5 замен ассоциированы со следующими признаками: Multiple sclerosis, Type 1 diabetes, повышение колличественных показателей LDL cholesterol,Cholesterol total
Согласно данным по snp, полученным с помощью Clinvar, snp с координатами 35861068 и 35871190 патогенны, а именно ассоциированы с тяжелым комбинированным иммунодефицитом
Назад
© Петрова Юлия 2016