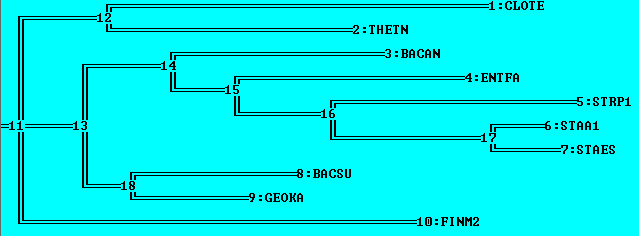
Филогенетическое дерево
- Задание 1
Отобранные бактерии:
| Название | Мнемоника |
| Bacillus anthracis | BACAN |
| Bacillus subtilis | BACSU |
| Clostridium tetani | CLOTE |
| Enterococcus faecalis | ENTFA |
| Geobacillus kaustophilus | GEOKA |
| Finegoldia magna | FINM2 |
| Staphylococcus aureus | STAA1 |
| Staphylococcus epidermidis | STAES |
| Streptococcus pyogenes | STRP1 |
| Thermoanaerobacter tengcongensis | THETN |
Скобочная форма дерева:
(((ENTFA,STRP1),((STAA1,STAES),((BACAN,BACSU),GEOKA))),((CLOTE,FINM2),THETN));
Изображение дерева:
Нетривиальные ветви дерева:
1) {ENTFA,STRP1} vs {STAA1,STAES,BACAN,BACSU,GEOKA,CLOTE,FINM2,THETN}
2) {STAA1,STAES} vs {ENTFA,STRP1,BACAN,BACSU,GEOKA,CLOTE,FINM2,THETN}
3) {BACAN,BACSU} vs {ENTFA,STRP1,STAA1,STAES,GEOKA,CLOTE,FINM2,THETN}
4) {CLOTE,FINM2} vs {ENTFA,STRP1,STAA1,STAES,BACAN,BACSU,GEOKA,THETN}
5) {BACAN,BACSU,GEOKA} vs {ENTFA,STRP1,STAA1,STAES,CLOTE,FINM2,THETN}
6) {CLOTE,FINM2,THETN} vs {ENTFA,STRP1,STAA1,STAES,BACAN,BACSU,GEOKA}
7) {STAA1,STAES,BACAN,BACSU,GEOKA} vs {ENTFA,STRP1,CLOTE,FINM2,THETN}
Филогенетическое дерево, занятие 2.
Задание 1
Таксономия:
BACAN: Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus cereus group
BACSU: Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus subtilis group
GEOKA: Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Geobacillus
STAA1: Bacteria; Firmicutes; Bacilli; Bacillales; Staphylococcaceae; Staphylococcus
STAES: Bacteria; Firmicutes; Bacilli; Bacillales; Staphylococcaceae; Staphylococcus
ENTFA: Bacteria; Firmicutes; Bacilli; Lactobacillales; Enterococcaceae; Enterococcus
STRP1: Bacteria; Firmicutes; Bacilli; Lactobacillales; Streptococcaceae; Streptococcus
THETN: Bacteria; Firmicutes; Clostridia; Thermoanaerobacterales; Thermoanaerobacteraceae; Caldanaerobacter; Caldanaerobacter subterraneus
CLOTE: Bacteria; Firmicutes; Clostridia; Clostridiales; Clostridiaceae; Clostridium
FINM2: Bacteria; Firmicutes; Clostridia; Clostridiales; Clostridiales incertae sedis; Clostridiales Family XI. Incertae Sedis; Finegoldia
Ветви:
Классы:
Ветвь (((ENTFA,STRP1),((STAA1,STAES),((BACAN,BACSU),GEOKA))) - Bacilli
Ветвь ((CLOTE,FINM2),THETN) - Clostridia
Поряки:
Ветвь (ENTFA,STRP1) - Lactobacillales
ветвь (((BACAN,BACSU),GEOKA),(STAA1,STAES)) - Bacillales
ветвь (CLOTE,FINM2) - Clostridiales
Семейства:
Ветвь (STAA1,STAES) - Staphylococcaceae, Staphylococcus (Род)
ветвь ((BACAN,BACSU),GEOKA)) - Bacillaceae
Роды:
Ветвь (BACAN,BACSU) - Bacillus
Задание 2
Выбранная функция - энолаза (ENO).
Команда для скачивания entret-файлов со Swiss-prot: entret sw:(мнемоника функции белка)_(мнемоника организма)
Команда для создания fasta-файла с последовательностями белков: seqret "*.entret" all.fasta
Задание 3
Команда для выравнивания последовательностей белков: muscle -in all.fasta -out allign.fasta
Задание 5
Команда для получения филогенетического дерева: fprotpars -in allign.fasta
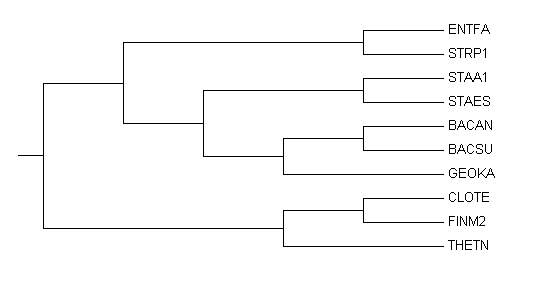
Правильное дерево:
 |
Полученное дерево:
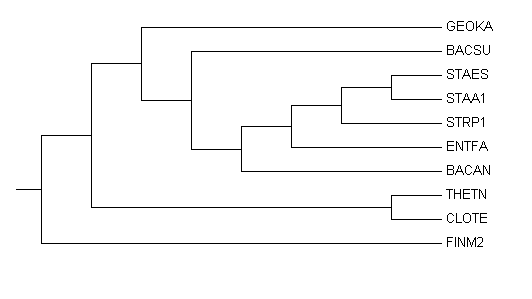 |
Ветви, которые есть в обоихдеревьях:
{STAA1,STAES} vs {ENTFA,STRP1,BACAN,BACSU,GEOKA,CLOTE,FINM2,THETN}
{CLOTE,FINM2,THETN} vs {ENTFA,STRP1,STAA1,STAES,BACAN,BACSU,GEOKA}
Ветви, которые есть только в правильном дереве:
{ENTFA,STRP1} vs {STAA1,STAES,BACAN,BACSU,GEOKA,CLOTE,FINM2,THETN}
{BACAN,BACSU} vs {ENTFA,STRP1,STAA1,STAES,GEOKA,CLOTE,FINM2,THETN}
{CLOTE,FINM2} vs {ENTFA,STRP1,STAA1,STAES,BACAN,BACSU,GEOKA,THETN}
{BACAN,BACSU,GEOKA} vs {ENTFA,STRP1,STAA1,STAES,CLOTE,FINM2,THETN}
{STAA1,STAES,BACAN,BACSU,GEOKA} vs {ENTFA,STRP1,CLOTE,FINM2,THETN}
Ветви, которые есть только в полученном дереве:
{STAA1,STAES,STRP1} vs {ENTFA,BACAN,BACSU,GEOKA,CLOTE,FINM2,THETN}
{STAA1,STAES,STRP1,ENTFA} vs {BACAN,BACSU,GEOKA,CLOTE,FINM2,THETN}
{STAA1,STAES,STRP1,ENTFA,BACAN} vs {BACSU,GEOKA,CLOTE,FINM2,THETN}
{STAA1,STAES,STRP1,ENTFA,BACAN,BACSU} vs {GEOKA,CLOTE,FINM2,THETN}
Задание 6
Матрица расстояний:
FINM2 CLOTE THETN BACAN BACSU GEOKA ENTFA STRP1 STAA1 STAES
FINM2 0.000000 0.422314 0.346572 0.362652 0.328530 0.304786 0.402775 0.469770 0.412631 0.417858
CLOTE 0.422314 0.000000 0.295729 0.410973 0.356587 0.319608 0.423546 0.491095 0.462743 0.464736
THETN 0.346572 0.295729 0.000000 0.348618 0.299499 0.259085 0.349405 0.419228 0.416549 0.431330
BACAN 0.362652 0.410973 0.348618 0.000000 0.234548 0.218080 0.243710 0.298437 0.275402 0.283263
BACSU 0.328530 0.356587 0.299499 0.234548 0.000000 0.135023 0.286696 0.349118 0.306673 0.311242
GEOKA 0.304786 0.319608 0.259085 0.218080 0.135023 0.000000 0.267808 0.339292 0.300606 0.299508
ENTFA 0.402775 0.423546 0.349405 0.243710 0.286696 0.267808 0.000000 0.218899 0.308318 0.323543
STRP1 0.469770 0.491095 0.419228 0.298437 0.349118 0.339292 0.218899 0.000000 0.211608 0.236847
STAA1 0.412631 0.462743 0.416549 0.275402 0.306673 0.300606 0.308318 0.211608 0.000000 0.058540
STAES 0.417858 0.464736 0.431330 0.283263 0.311242 0.299508 0.323543 0.236847 0.058540 0.000000
Ультраметричность: d(A,B) <= max(d(A,C), d(B,C))
"из трех расстояний между тремя объектами два всегда равны между собой и не меньше третьего".
Отклонения от ультраметричности:
d(BACAN,THETN)=0.348618
d(BACAN,CLOTE)=0.410973
d(CLOTE,THETN)=0.295729
Удоволетворяет ультраметричности:
d(STAA1,FINM2)=0.412631
d(STAES,FINM2)=0.417858
d(STAA1,STAES)=0.058540
Аддитивность: если есть 4 последовательности: A, B, C, D, - то из трех сумм d(A,B) + d(C,D); d(A,C) + d(B,D);
d(A,D) + d(B,C) две равны между собой и больше третьей.
GEOKA, ENTFA, STRP1, STAA1:
d(GEOKA, ENTFA)+d(STRP1, STAA1)=0.267808+0.211608=0.479416
d(GEOKA, STRP1)+d(ENTFA, STAA1)=0.339292+0.308318=0.64761
d(GEOKA, STAA1)+d(ENTFA, STRP1)=0.300606+0.218899=0.519505
Этот пример не удоволетворяет аддитивности.
Задание 7
Правильное дерево:
 |
Дерево, полученное с помощью программы fprotdist:
 |
Дерево, полученное с использованием алгоритма UPGMA:
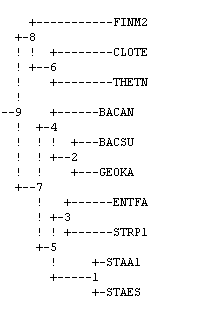 |
Дерево, полученное с использованием алгоритма Neighbor-Joining:
 |
Дерево, полученное с помощью алгоритма UPGMA ближе к правильному дереву, а дерево Neighbor-Joining - ближе к полученному с помощью программы
fprotdist. Однако все 4 дерева получились различные.
Занятие 3.
Задание 1
- Укоренить дерево, построенное методом максимльной экономии, нельзя, потому что этот алгоритм не выдает длин ветвей.
- Укоренять дерево, постороенное методом UPGMA не имеет смысла, так как UPGMA строит уже укорененные деревья.
Дерево, полученное методом neighbour-joining:
Правильное дерево:

(((ENTFA,STRP1),((STAA1,STAES),((BACAN,BACSU),GEOKA))),((CLOTE,FINM2),THETN));
|
Укорененное дерево:

(((BACAN,(ENTFA,(STRP1,(STAA1,STAES)))),(BACSU,GEOKA)),(FINM2,(CLOTE,THETN)));
|
Дерево было укоренено в ветвь {CLOTE,THETN,FINM2} vs {ENTFA,STRP1,STAA1,STAES,BACAN,BACSU,GEOKA}, то есть корень совпадает с корнем
правильного дерева.
Однако правильное и укорененное деревья отличаются по структуре ветвей:
В обоих деревьях совпадает только ветвь (STAA1,STAES). Все остальные ветви различны.
Задание 2
Правильное дерево:
 |
Укорененное дерево c ECOLI:
 |
1) Укорененное дерево с ECOLI полностью совпадает с деревом, построенным с помощью Neighbor-joining.
Задание 3
Правильное дерево:
 |
Дерево, полученное с помощью программы fconsense:
 |
1) Дерево, построенное программой fconsense сильно отличается от всех деревьев, в том числе и от правильного.
2) Реконструкция филогении не улучшилась. Возможно, такой результат получен из-за работы с данным
конкретным семейством белков.
Занятие 4.
Задание 1
Правильное дерево:
 |
Дерево, построенное программой fdnaml:
 |
Количественная оценка:
Число общих ветвей у деревьев, построенных приведенными ниже программами с эталонным деревом.
1) Метод NJ программы fneighbor:
Число общих ветвей - 2:
{STAA1,STAES} vs {ENTFA,STRP1,BACAN,BACSU,GEOKA,CLOTE,FINM2,THETN}
{CLOTE,FINM2,THETN} vs {ENTFA,STRP1,BACAN,BACSU,GEOKA,STAA1,STAES}
2) Метод UPGMA программы fneighbor:
Число общих ветвей - 4:
{STAA1,STAES} vs {ENTFA,STRP1,BACAN,BACSU,GEOKA,CLOTE,FINM2,THETN}
{CLOTE,FINM2,THETN} vs {ENTFA,STRP1,BACAN,BACSU,GEOKA,STAA1,STAES}
{BACAN,BACSU,GEOKA} vs {ENTFA,STRP1,CLOTE,FINM2,THETN,STAA1,STAES}
{ENTFA,STRP1} vs {BACAN,BACSU,GEOKA,CLOTE,FINM2,THETN,STAA1,STAES}
3) fprotpars без bootstrap'а:
Число общих ветвей - 2:
{STAA1,STAES} vs {ENTFA,STRP1,BACAN,BACSU,GEOKA,CLOTE,FINM2,THETN}
{CLOTE,FINM2,THETN} vs {ENTFA,STRP1,STAA1,STAES,BACAN,BACSU,GEOKA}
4) fprotpars c bootstrap'ом:
Число общих ветвей - 1:
{STAA1,STAES} vs {ENTFA,STRP1,BACAN,BACSU,GEOKA,CLOTE,FINM2,THETN}
5) fdnaml (дерево, построенное по нуклеотидным последовательностям):
Число общих ветвей - 4:
{CLOTE,FINM2} vs {ENTFA,THETN,STRP1,BACAN,BACSU,GEOKA,STAA1,STAES}
{CLOTE,FINM2,THETN} vs {ENTFA,STRP1,BACAN,BACSU,GEOKA,STAA1,STAES}
{STAA1,STAES} vs {ENTFA,STRP1,BACAN,BACSU,GEOKA,CLOTE,FINM2,THETN}
{ENTFA,STRP1} vs {BACAN,BACSU,GEOKA,CLOTE,FINM2,THETN,STAA1,STAES}
Таким образом дерево, полученное методом UPGMA программы fneighbor оказалось наиболее близким к эталонному.
Доделки (практикум 13)
1) Отобранные бактерии:
| Мнемоника | AC | FT | Команда |
| BACAN | AE016879 | направление соответствует выбранному для записи | seqret "BACAN.enret[9335:10841]" stdout >> rna.fasta |
| CLOTE | AE015927 | направление соответствует направлению, выбранному для записи | seqret "CLOTE.enret[8715:10223]" stdout >> rna.fasta |
| ENTFA | AE016830 | направление соответствует направлению, выбранному для записи | seqret "ENTFA.entret[248466:249987]" stdout >> rna.fasta |
| GEOKA | BA000043 | направление соответствует направлению, выбранному для записи | seqret "GEOKA.entret[10421:11973]" stdout >> rna.fasta |
| FINM2 | AP008971 | направление соответствует направлению, выбранному для записи | seqret "FINM2.entret[197837:199361]" stdout >> rna.fasta |
| STAA1 | AP009324 | направление соответствует направлению, выбранному для записи | seqret "STAA1.entret[531922:533476]" stdout >> rna.fasta |
| STAES | AE015929 | РНК лежит на комплементарной цепи. | seqret STAES.entret -sask stdout >>rna.fasta |
| STRP1 | AE004092 | направление соответствует направлению, выбранному для записи | seqret "STRP1.entret[17170:18504]" stdout >> rna.fasta |
| THETN | AE008691 | направление соответствует направлению, выбранному для записи | seqret "THETN.entret[53858:55384]" stdout >> rna.fasta |
2) Команда, с помощью которой получено выравнивание последовательностей 16S рРНК бактерий:
muscle -in rna.fasta -out rna_a.fasta
3) Для построения деревьев я использовала две команды: fdnaml, fdnapars. На вход подавался
файл с последовательностями rna_a.fasta. Все файлы лежат в дирректории H:\Term4\Practice4.
4)Сравним эталонное дерево с деревом, полученным программой fdnapars из рРНК-последовательностей и с каждым из деревьев, полученных по белковым выравниваниям
в практикуме 2.
а)Скобочная формула эталонного дерева
(((ENTFA,STRP1),((STAA1,STAES),((BACAN,BACSU),GEOKA))),((CLOTE,FINM2),THETN))
Скобочная формула РНК-дерева:
(STRP1,(ENTFA,(BACAN,((STAES,STAA1),(GEOKA,(THETN,FINM2)))),CLOTE)
У этих деревьев имеется единаственная общая ветвь:
(STAES,STAA1)
b)РНК-дерево и дерево, построенное по Neighbor-Joining:
Скобочная формула РНК-дерева:
(STRP1,(ENTFA,(BACAN,((STAES,STAA1),(GEOKA,(THETN,FINM2)))),CLOTE)
Скобочная формула дерева NJ:
(((((STAES,STAA1),STRP1),ENTFA),BACAN),(THETN,CLOTE),(GEOKA,BACSU),FINM2)
Общая ветвь та же:
(STAES,STAA1)
с)РНК-дерево и дерево, построенное программой UPGMA:
Скобочная формула РНК-дерева:
(STRP1,(ENTFA,(BACAN,((STAES,STAA1),(GEOKA,(THETN,FINM2)))),CLOTE)
Скобочная формула дерева UPGMA:
((FINM2,(CLOTE,THETN)),((BACAN,(BACSU,GEOKA)),((ENTFA,STRP1),(STAA1,STAES))));
Общая ветвь та же:
(STAES,STAA1)
Однако это дерево наиболее близко по структуре к эталонному.
d) Дерево, построенное с помощью программы fprotdist наиболее близко к РНК-дереву.
Задание 2
1) Чтобы найти гомологи белка CLPX_BACSU, были использованы следующие команды:
formatdb -i proteo.fasta -p T
blastall -p blastp -d proteo.fasta -i CLPX_BACSU.fasta -o CLPX_BACSU_out.fasta -e 0.001
2) Отобранные гомологи: CLPX_LISMO, HSLU_LISMO, CLPX_THETN, HSLU_THETN, CLPX_CLOTE, CLPX_ENTFA,
HSLU_ENTFA, CLPX_LACLM, HSLU_STAES, CLPC_BACSU, CLPE_BACSU, CLPY_BACSU, CLPC_STAES.
3) Дерево, полученное с помощью программы fprotpars:

Ортологи - белки а) из разных организмов; б) разделение их общего предка на линии,
ведущие к ним, произошло в результате видообразования.
Примеры ортологов:
HSLU_LISMO - HSLU_STAES
CLPX_CLOTE - CLPX_THETN
CLPC_BACSU - CLPC_STAES
Парологи - два гомологичных белка из одного организма.
Примеры парологов:
СLPX_BACSU - CLPY_BACSU
CLPX_BACSU - CLPC_BACSU
CLPX_THETN - HSLU_THETN
© Karavaeva Julia 2009