Практикум 8
Поиск гена, кодирующего δ-субъединицу АТФ-синтазы
Идентификатор белка: XM_001416157.1
Последовательность белка: fasta
Идентификатор нуклеотидной записи: NC_009356.1
Последовательность гена с его окрестностью: fasta
Координаты белок-кодирующей части гена: 613721..615313
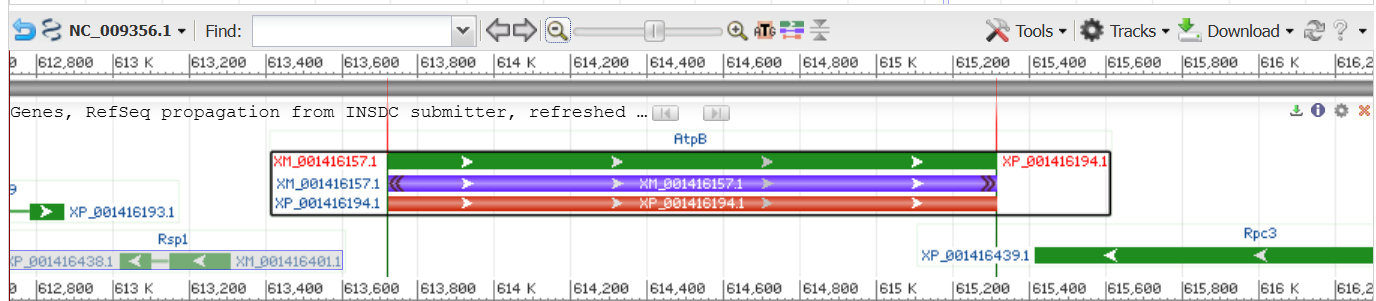
Поиск гомологов
По последовательности гена, полученного в предыдущем задании, был произведен поиск гомологичных генов с помощью алгоритма BLAST. Использовалась база данных refseq_genomes, поиск производился по таксону Пауки (Araneae), точнее - по 4 аннотированным геномам пауков.
Были использованы алгоритмы blastn и tblastn. Blastn используется для выравнивания нуклеотидных последовательностей на нуклеотидную базу данных. Алгоритм не очень хорош, если нас интересуют белки, поскольку он не учитывает вырожденность генетического кода, то есть чувствителен к т. н. "молчащим" нуклеотидным заменам, не влияющим на аминокислоту и, соответственно, на белок. В связи с этим число находок нулкеотидного бласта для белок-кодирующих последовательностей как правило меньше, чем число находок аналогичных "бластов" по белковым последовательностям, таких как, например, tblastn. Tblastn сперва транслирует нуклеотидную базу данных в как-бы белковую, затем производя по уже белковой базе данных поиск гомологов нашего белка. Tblastn будет более точен в поиске гомологов, поскольку "не замечает" однонуклеотидных замен, не влияющих на белок, а также использует матрицу замен.
В алгоритме blastn длина слова - 11, число находок - 19.
В алгоритме tblastn длина слова - 5, число находок - 28.
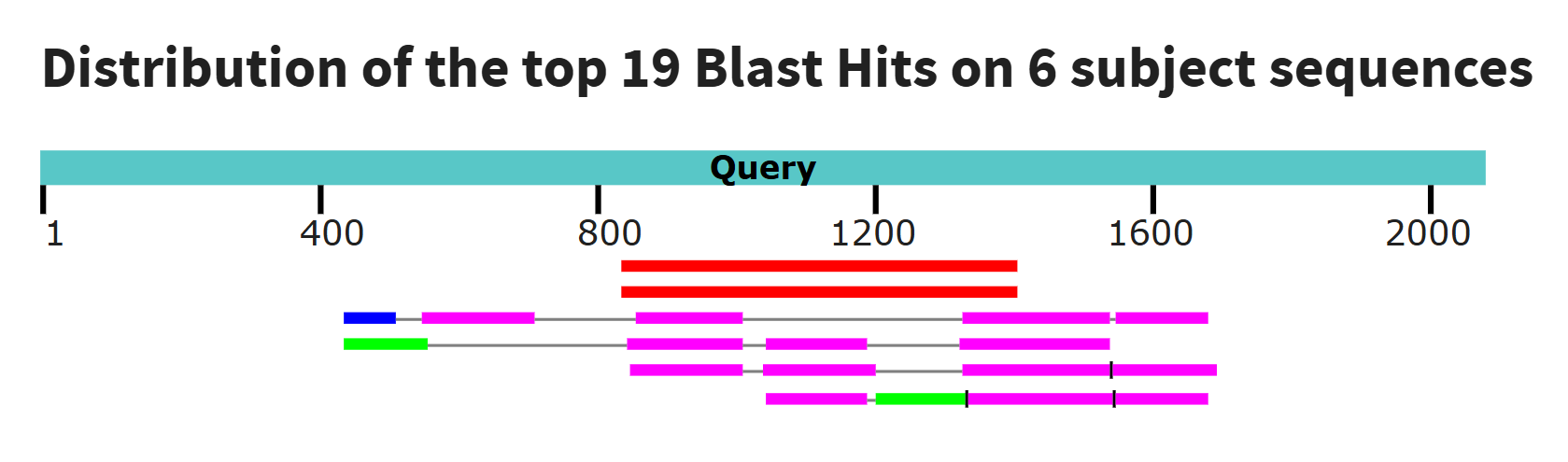
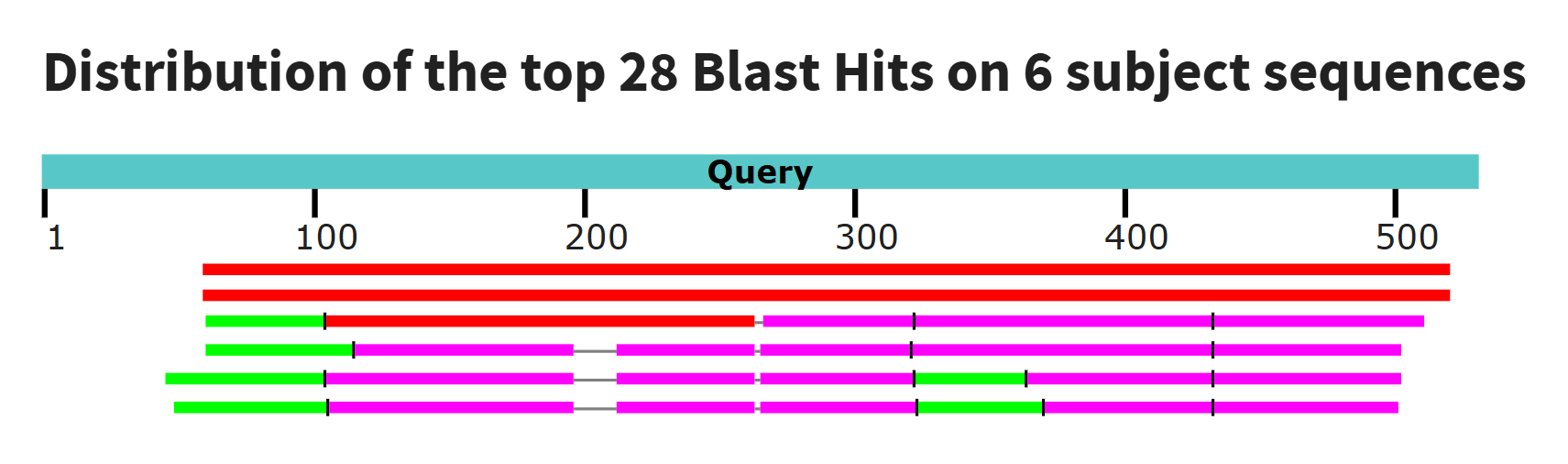
Поиск гомологов эукариотической рРНК по далекому гомологу
Сначала я создала локальную нуклеотидную базу данных с геномом моего эукариота Ostreococcus lucimarinus с помощью команды:
Ранее были сохранены нуклеотидные последовательности двух рРНК (16S и 23S) Escherichia coli. Для каждой из них я провела локальный поиск BLAST.
Для последовательности 16S-рРНК было найдено 16 находок, для последовательности 23S-рРНК найдена 21 находка, все аннотированны в RefSeq.

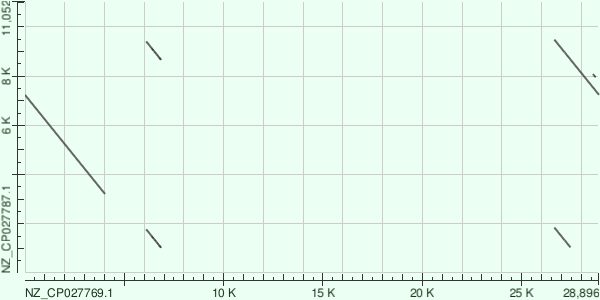
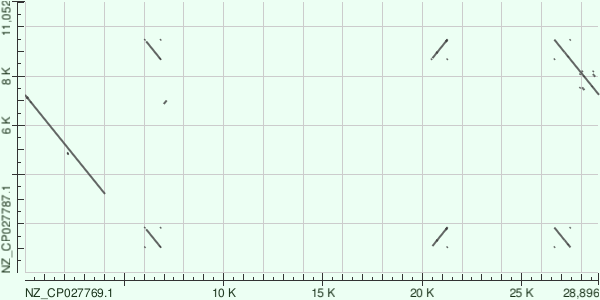
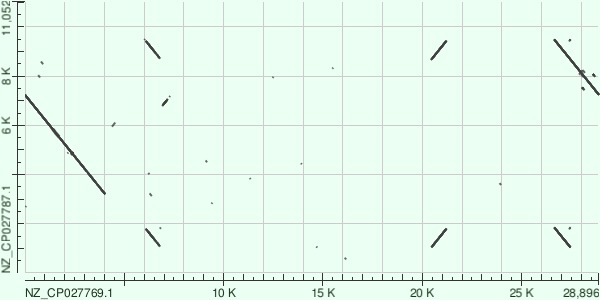
Карты локального сходства
Для сравнения результатов megablast, blastn и tblastx я выбрала плазмиды бактерий Tetragenococcus halophilus (NZ_CP027769.1) и Tetragenococcus koreensis (NZ_CP027787.1). Заслуживаем внимания, что megablast не смог распознать существенную дупликацию.