Целью этого задания было получение последовательности ДНК, основываясь на
двух последовательностях, сгенерированных после секвенирования по Сэнгеру.
Первая последовательность была прямой, вторая - обратной.
Чтобы выполнить это задание, в первую очередь необходимо было проанализировать хроматограммы обеих последовательностей и определённым образом изменить их.
Подробнее об этом ниже:
Прямая хроматограмма хорошо читалась со 44 по 706 нуклеотид; обратная (к которой предварительно был применён reverse+complement) читалась со 36 по 751 нуклеотид )5', 3'-концы).
В среднем сигнал превосходил шум в 10 раз, но встречались и участки, где сигнал превосходил шум в 3-4 раза.
Средняя сила сигнала и шума вдоль оси X в обеих хроматограммах, за исключением концов, равномерна.
В прямой цепи наиболее сильный сигнал наблюдается у нуклеотидов A и G (сильнее остальных сигналов в 1.5 раза)(см. Рис. 1 и 2).В обратной цепи сигналы были сильнее у T и C (интенсивнее остальных сигналов примерно в 2 - 2.5 раза)(см. Рис. 3 и 4).
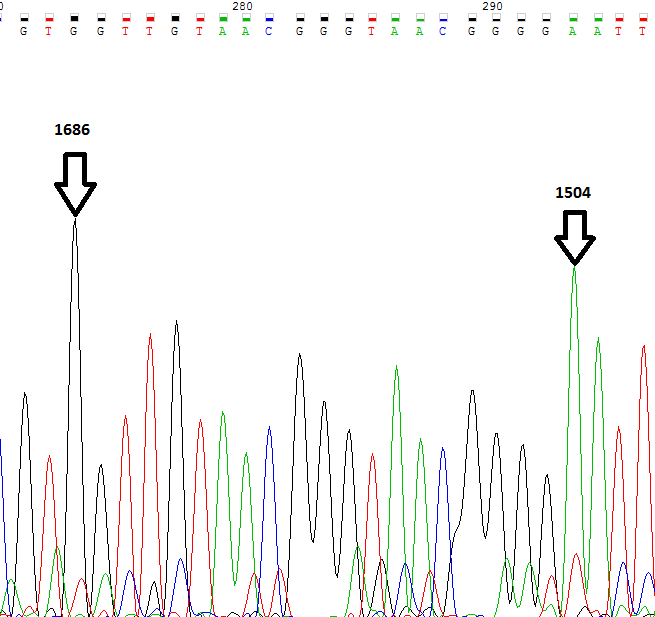 Рис.1. |
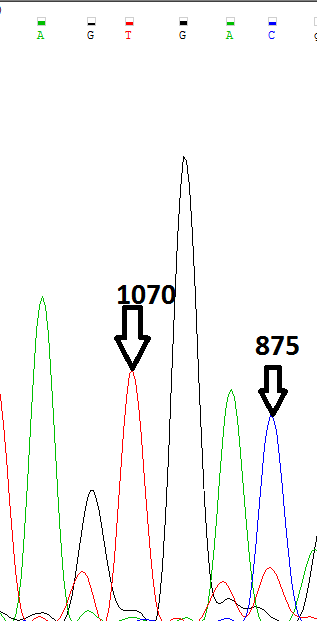 Рис.2. |
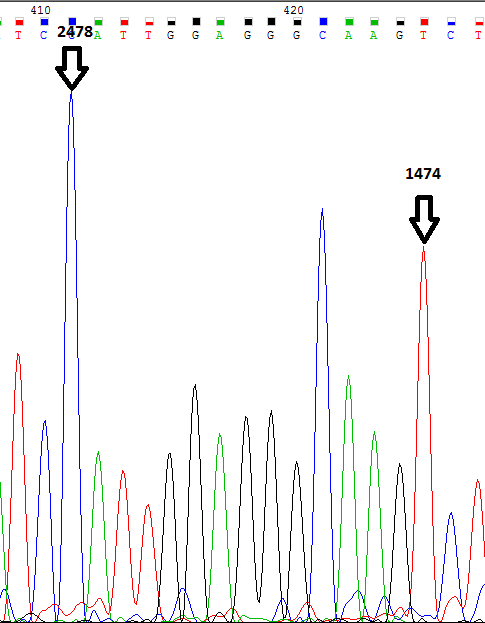 Рис.3. |
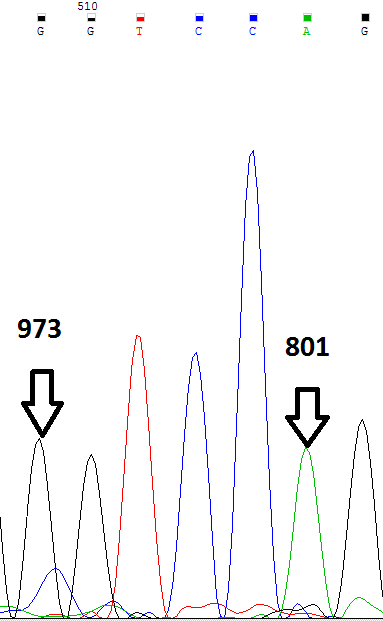 Рис.4 |
Проанализировав хроматограммы, я отредактировала неоднозначные места в base calling каждой хроматограммы, основываясь на данных из другой хроматограммы. Эти исправления проделаны маленькими буквами. Приведу несколько примеров таких исправлений:
Таблица 2. Прямая цепь (левая картинка - обратная)
Как правило, непрочтитанные места в одной хроматограмме читались в другой (см. Рис. 5 и 6). | |
 Рис.5. |
 Рис.6. |
Аналогично | |
|
|
|
Тоже самое | |
 Рис.9. Рис.9.
|
 Рис.10. Рис.10.
|
Тоже самое | |

|

|
Ориентируясь по обратной цепи, в прямой не хватает "С" | |

|

|
Таблица 3. Обратная цепь
Указанная буква определена по прямой цепи< | |
 Рис.11 |
 Рис.12 |
Указанная буква определена по прямой цепи | |
|
|
|
К сожалению здесь мой зоркий глаз упустил букву в "Chromas", поэтому прикрепляю выравнивание. | |
 Рис.15 Рис.15
|
|
Таким образом, обе последовательности были выравнены в JalView (см. здесь); были получены исправленные хроматограммы (прямая и обратная) и на основании полученного выравнивания была построена гипотетическая "чистая" последовательность, в которой N = A, T, G, C. Никаких резких ухудшений качества найдено не было. Передние и задние концы нечитаемы (из-за наличия коротких фрагментов, на которых отжигается праймер), однако сравнение хромотограмм прямой и обратной цепи позволило внести ясность в определение последовательности. В целом качество хроматограмм хорошее. Пики в основном четкие, расположены на приблизительно одинаковом расстоянии друг от друга. Средняя сила сигналов и шума равномерна по всей длине последовательностей за исключением некоторых мест. В среднем шум в 5 раз менее интенсивен, чем сигналы нуклеотидов, хотя в единичных случаях он достигает половины интенсивности сигнала или даже совпадает с ним по силе. Очевидные пики, соответствующие отдельным нуклеотидам, иногда отличаются по силе. Для гуанина (G, черный) и аденина (A, зеленый) такие отличия могут быть в 4-5 раз, для цитозина (С, синий) и тимина (T, красный) не более чем в 2 раза.
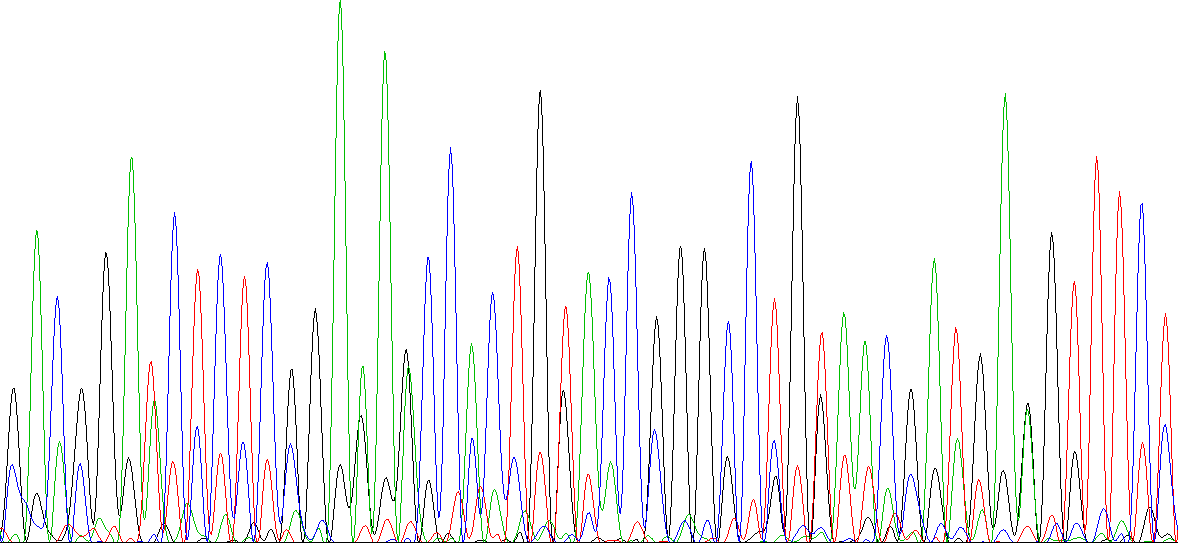
Далее приведён пример "плохой" хроматограммы WSWS2931.ab1(см. Рис. 15):
Пример нечитаемой хроматограммы из файла WS2943.ab1(см. Рис. 16):
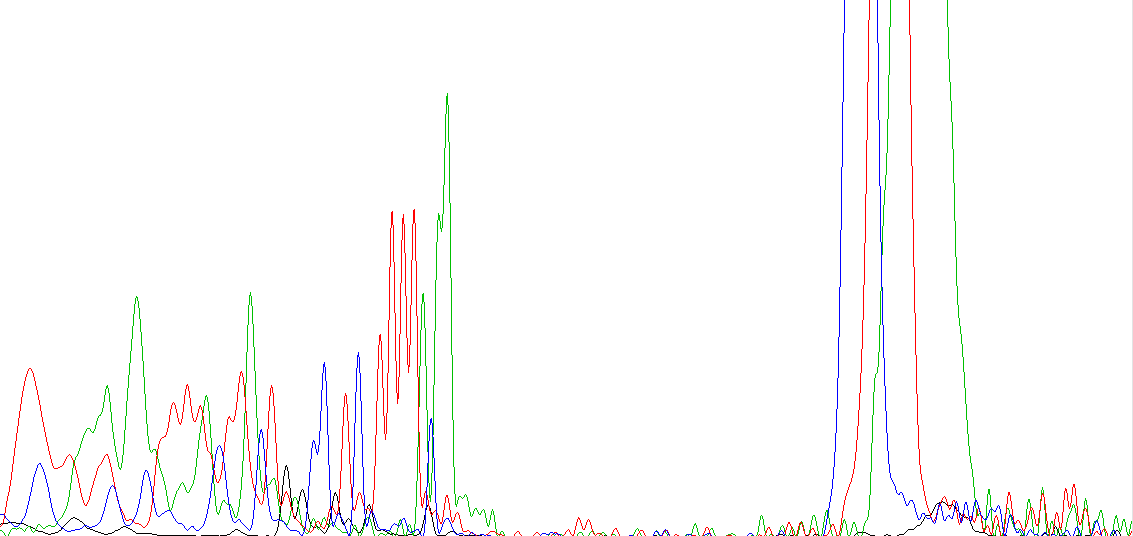
Рис.16
Первую из них читать невозможно, так как уровень шума часто совпадает с уровнями сигналов и иногда имеет ту же интенсивность, что и обычный чистый сигнал. Таким образом, нельзя определить, является ли самый высокий пик сигналом правильного нуклеотида или шумом. Во второй хроматограмме заметен резкий скачок интенсивности сигналов. Неясно, чем мог быть вызван такой переход и как он повлиял на правильность полученных данных. При этом пики сигналов часто сливаются в один удлиненный пик, что также вносит нечеткость в определение последовательности. Возможно, наиболее большие "пики" - это следы краски, кляксы. Если посмотреть обратную цепь, результат лучше не станет. Кроме того, не исключено, что в препарате присутствует не одна ДНК.
***
В качестве дополнения ко всему вышесказанному, я провела поиск гомологичной последовательности blast'ом (мне стало интересно, с чем я работаю). Выдача была следующей:

Как можно видеть, перед нами ген 18S (входит в малую субъединицу) рибосомальной РНК червя Saccocirrus tridentiger, его таксономия такая:
>Eukaryota
>Metazoa
>Lophotrochozoa
>Annelida
>Polychaeta
>Polychaeta incertae sedis
>Saccocirridae
>Pharyngocirrus
Вообще существует 2 "расы" этих червей - "papillocercus" и "krusadensis, как правило, исследуются особенности обеих рас. Наш образец, видимо, "krusadensis" [1].
[4] - NCBI // URL: https://www.ncbi.nlm.nih.gov/pubmed?LinkName=nuccore_pubmed&from_uid=669173618
© Yuliia Preobrazhenskaya, 2016