| Нитроредуктаза | |
 | |
| Информация о белке | |
|---|---|
| Uniprot ID |
M4KDD6_LACPN
|
| Uniprot AC | M4KDD6 |
| Refseq ID | WP_003641675.1 |
| PDB ID | Точного соответствия Uniprot Retrieve/ID mapping не находит, ближайший аналог: 4QLX |
| Молекулярная масса | 24270Da |
| Длина | 217 |
| Рекомендуемое название | Nitroreductase (нитроредуктаза) |
UniProt представляет собой базу данных последовательностей белков. Информация из базы данных доступна для всех пользователей. Она охватывает различные аспекты анализа белковых последовательностей. Многие из последовательностей стали известны в результате реализации проектов секвенирования геномов последних лет. Кроме того, база данных UniProt содержит большое количество информации о биологических функциях белков, полученной из научной литературы.
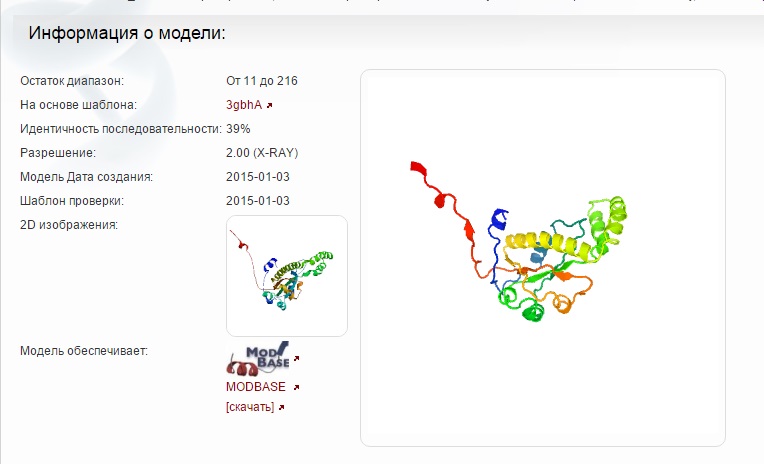
Существование данного белка не доказано и, соответственно, структура на PDB есть только для ближайшего аналога - 4QLX. Его структура представлена двумя одинаковыми цепями - А и В. Для белка M4KDD6_LACPN же в записях имеется пометка только о визуализации предполагаемой структуры на сайте ProteinModelPortal.Структура кристалла, соответственно, не известна. Видимо, изучением данного фермента не занимались (всего лишь 1 запись, аннотированная экспертом).
Содержание
- 1 Работа с белком в Uniprot
- 1.1 Описание кластеров Uniref
- 1.2 Результаты сеансов поиска в Uniprot. Гемоглобин, актин и трипсин
- 2 История изменений записи
- 3 Нестандартные аминокислотные остатки
- 4 Модифицированные остатки
- 5 Альтернативный сплайсинг
- 6 Сравнение аннотаций записей белка в Uniprot и Refseq Proteins
- 7 Ссылки
UniProt представляет собой базу данных последовательностей белков. Информация из базы данных доступна для всех пользователей. Она охватывает различные аспекты анализа белковых последовательностей. Многие из последовательностей стали известны в результате реализации проектов секвенирования геномов последних лет. Кроме того, база данных UniProt содержит большое количество информации о биологических функциях белков, полученной из научной литературы.
Единая база данных UniProt была создана путем объединения баз данных Swiss-Prot, TrEMBL и PIR — PSD.
Swiss-Prot
База данных Swiss-Prot была создана в 1986 году Амосом Байрошем во время работы над своим PhD-проектом и развита в дальнейшем в Швейцарском Институте Биоинформатики (SIB), а позже доработана Рольфом Апвейлером в Европейском Институте Биоинформатики (EBI)[7][8][9]. Основная функция базы данных Swiss-Prot направлена на обеспечение надежности информации о белковых последовательностях, обусловленной высоким, детальным уровнем аннотации, выполненной вручную. Она включает описание функции белка, его доменной структуры, пост-трансляционных модификаций, различных вариантов последовательности и т. д., причём с минимальным уровнем избыточности и высоким уровнем интеграции с другими базами данных.
TrEMBL
База данных «Библиотека данных Нуклеотидных последовательностей» (TrEMBL) была разработана в 1996 году как аннотированное компьютерное приложение к Swiss-Prot[4][6][7]. Решение о создании TrEMBL было принято в ответ на увеличение потока данных в результате появления геномных проектов, а затратный по времени и трудоемкий процесс ручной аннотации в UniProtKB / Swiss-Prot превышал возможности Swiss-Prot для того, чтобы включить все доступные белковые последовательности[4][6]. TrEMBL предоставляет возможность автоматизированной аннотации для трансляции имеющихся нуклеотидных последовательностей и превращения их в белковые последовательности вне Swiss-Prot.
PIR-PSD
PIR, организованный Национальным фондом медико-биологических исследований (NBRF) в Медицинском центре Джорджтаунского университета в Вашингтоне, округ Колумбия, США, является наследником старейшей базы данных последовательностей белков, а именно, созданным Margaret Dayhoff «Атласом последовательностей белка и структуры», впервые опубликованным в 1965 году[10]. PIR поддерживает несколько белковых баз данных, а именно: главную базу белковых последовательностей (PIR-PSD), базу данных, связанную с классификацией белков по структуре и функциям (iProClass), а также другие базы данных белковых последовательностей и курируемых семейств.
После объединения каждый член консорциума активно участвует в обеспечении единой базы данных последовательностями белков и их аннотации.
*Далее можно посмотреть кластер (100%), называемый Enone reductase CLA-ER, в который входят 86 белков, из которых представлены 10
| Uniprot ID |
Организм |
Функция |
Существует ли | Биохимический путь |
|
Lactobacillus plantarum subsp. plantarum ATCC 14917 = JCM 1149 = CGMCC 1.2437 |
Оксидоредуктазная активность |
Существование предсказано |
Нитроредуктазы катализируют процесс взаимодействия в микробной клетке белков группы ферридоксинов с нитросоединениями. |
|
| Оксидоредуктазная активность |
Существование предсказано |
Нитроредуктазы катализируют процесс взаимодействия в микробной клетке белков группы ферридоксинов с нитросоединениями. |
||
| Оксидоредуктазная активность |
Существует |
Нитроредуктазы катализируют процесс взаимодействия в микробной клетке белков группы ферридоксинов с нитросоединениями. | ||
| Оксидоредуктазная активность |
Существование предсказано |
Нитроредуктазы катализируют процесс взаимодействия в микробной клетке белков группы ферридоксинов с нитросоединениями. |
||
| Оксидоредуктазная активность |
Существование предсказано |
Нитроредуктазы катализируют процесс взаимодействия в микробной клетке белков группы ферридоксинов с нитросоединениями. | ||
|
Lactobacillus plantarum (strain ATCC BAA-793 / NCIMB 8826 / WCFS1) |
НАДН-дегидрогеназа |
Существование предсказано |
Нитроредуктазы катализируют процесс взаимодействия в микробной клетке белков группы ферридоксинов с нитросоединениями. |
|
| Оксидоредуктазная активность |
Существование предсказано |
Нитроредуктазы катализируют процесс взаимодействия в микробной клетке белков группы ферридоксинов с нитросоединениями. |
||
|
Оксидоредуктазная активность |
Существование предсказано |
Нитроредуктазы катализируют процесс взаимодействия в микробной клетке белков группы ферридоксинов с нитросоединениями. |
*Далее можно посмотреть кластер (90%), называемый Enone reductase CLA-ER, в который входят 20 белков, из которых представлен 1, так как 10 есть в предыдущей
| Uniprot ID |
Организм |
Функция |
Существует ли | Биохимический путь |
| Оксидоредуктазная активность |
Существование предсказано |
Нитроредуктазы катализируют процесс взаимодействия в микробной клетке белков группы ферридоксинов с нитросоединениями. |
*Далее можно посмотреть кластер (50%), называемый Enone reductase CLA-ER, в который входят 86 белков, из которых представлены 2, так как 8 (первых) есть в таблице по Uniref100
| Uniprot ID |
Организм |
Функция |
Существует ли | Биохимический путь |
| Оксидоредуктазная активность |
Существование предсказано |
Нитроредуктазы катализируют процесс взаимодействия в микробной клетке белков группы ферридоксинов с нитросоединениями. | ||
| Оксидоредуктазная активность |
Существование предсказано |
Нитроредуктазы катализируют процесс взаимодействия в микробной клетке белков группы ферридоксинов с нитросоединениями. |
В поле "DE" можно найти всего 4 названия. Проведем поиск по каждому из них без уточнения таксона
При запросе "Enone reductase" обнаруживается 8 белков разных организмов (7 в таблице, еще один - данный).Четыре из них описаны (имеют звездочку в таблице), также описаны их гены и в названии отображен субстрат для реакции (например, 2-methylene-furan-3-one reductase) и отсутствует слово "putative" (предполагаемая функция). Для "reviwed" такжеописаны константы Михаэлиса, оптимумы температуры и рН, каталитическая активность, сайты связывания лигандов или кофакторов, имеются кристаллы. Кроме того, идентификаторы Uniprot ID короче у описанных белков.
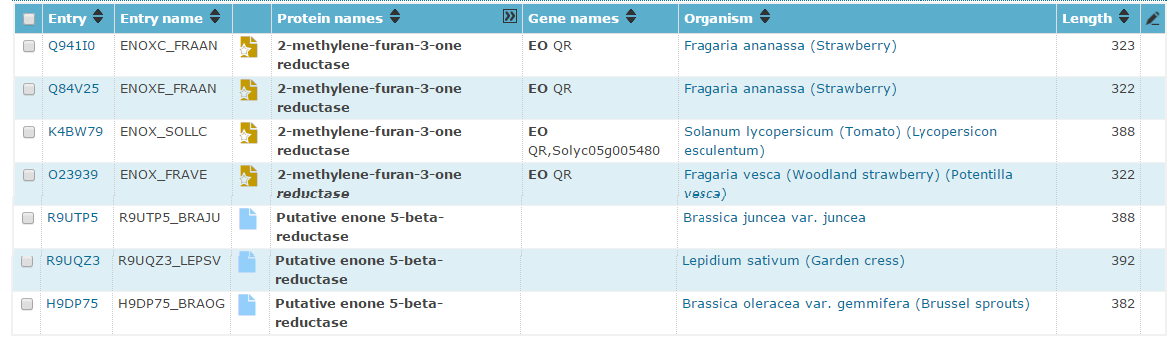
При запросе "NAD(P)H-dependent quinone reductase" обнаруживается 75 белков разных организмов. Ни один из данных белков не описан in vivo и in vitro, поэтому нет информации о функциях и кинетике. Кроме того, идентификаторы Uniprot ID длинее у неописанных белков. Также для некоторых неописанных белков здесь представлены только фрагменты (очень маленькая длина).
При запросе "NADH dehydrogenase" обнаруживается 164004 белков разных организмов. 6145 из них описано (имеют звездочку в таблице) и относятся к абсолютно разным организмам.
При запросе "nitroreductase" обнаруживается 27389 белков разных организмов. 72 из них описаны (имеют звездочку в таблице), также описаны их гены и в названии отображен субстрат для реакции и отсутствует слово "putative" (предполагаемая функция). Для "reviwed" такжеописаны константы Михаэлиса, оптимумы температуры и рН, каталитическая активность, сайты связывания лигандов или кофакторов, имеются кристаллы. Кроме того, идентификаторы Uniprot ID короче у описанных белков.
Теперь учтем таксон - для начала, семейство. В данном случае это Lactobacillaceae
При запросе "NAD(P)H-dependent quinone reductase" обнаруживается 33 белков разных организмов. Ни один из них не описан.
При запросе "Enone reductase" обнаруживается 1 белок, не являющийся описанным.
При запросе "NADH dehydrogenase" обнаруживается 350 белков разных организмов. Ни один из них не описан.
При запросе "nitroreductase" обнаруживается 804 белков разных организмов. Ни один из данных белков не описан in vivo и in vitro, поэтому нет информации о функциях и кинетике. Последовательности загружены автоматичски с помощью TrEMBL. Длина нитроредуктаз данного семейства варьирует в пределе 138-365 аминокислотных остатков.
Рассмотрим заданные белки типа Firmicutes
При запросе "NADH dehydrogenase" обнаруживается 7695 белков разных организмов. 151 из них описан (имеют звездочку в таблице), также описаны их гены и в названии отображен субстрат для реакции ) и отсутствует слово "putative" (предполагаемая функция). Для "reviwed" такжеописаны константы Михаэлиса, оптимумы температуры и рН, каталитическая активность, сайты связывания лигандов или кофакторов, имеются кристаллы.
При запросе "nitroreductase" обнаруживается 7691 белков разных организмов. 20 из них описаны (имеют звездочку в таблице), также описаны их гены и в названии отображен субстрат для реакции и отсутствует слово "putative" (предполагаемая функция). Для "reviwed" такжеописаны константы Михаэлиса, оптимумы температуры и рН, каталитическая активность, сайты связывания лигандов или кофакторов, имеются кристаллы.
При запросе "NAD(P)H-dependent quinone reductase" обнаруживается 69 белков разных организмов. Ни один из них не описан.
При запросе "Enone reductase" обнаруживается 1 белок, не являющийся описанным [1].
Поиск заданных белков по 3 вариантам
Гемоглобин
При запросе "Hemoglobin" без ограничения на организмы обнаруживается 8374 белков разных организмов, а также различных субъединиц гемоглобина. 944 из них описаны (имеют звездочку в таблице), также описаны их гены и в названии отображен субстрат для реакции и отсутствует слово "putative" (предполагаемая функция). Для "reviwed" такжеописаны константы Михаэлиса, оптимумы температуры и рН, каталитическая активность, сайты связывания лигандов или кофакторов, имеются кристаллы в форме, связанной с лигадами, причем как естественными, так и приводящими к патологии
При запросе "Hemoglobin" таксона "Ciliophora" обнаруживается 10 белков разных инфузорий, представляющих собой гемоглобин-подобные белки. 3 из них описаны (имеют звездочку в таблице), также описаны их гены и в названии отображен субстрат для реакции и отсутствует слово "putative" (предполагаемая функция).
При запросе "Hemoglobin" таксона "Fungi" обнаруживается 44 белка разных дрожжей, представляющих собой гемоглобин-подобные белки. 4 из них описаны (имеют звездочку в таблице), также описаны их гены и в названии отображен субстрат для реакции и отсутствует слово "putative" (предполагаемая функция).
Актин
При запросе "Actin" без ограничения на организмы обнаруживается 37639 белков разных организмов, а также различных белков одного семейства одного организма. 775 из них описаны (имеют звездочку в таблице), также описаны их гены и в названии отображен субстрат для реакции и отсутствует слово "putative" (предполагаемая функция). Для "reviwed" такжеописаны константы Михаэлиса, оптимумы температуры и рН, каталитическая активность, сайты связывания лигандов или кофакторов, имеются кристаллы в форме, связанной с лигадами, причем как естественными, так и приводящими к патологии
При запросе "Actin" таксона "Fungi" обнаруживается 17025 белков разных грибов, а также различных белков одного семейства одного организма. 83 из них описаны (имеют звездочку в таблице), также описаны их гены и в названии отображен субстрат для реакции и отсутствует слово "putative" (предполагаемая функция).
При запросе "Actin" таксона "Fungi" обнаруживается 289 белков разных инфузорий, а также различных белков одного семейства одного организма. 10 из них описаны (имеют звездочку в таблице), также описаны их гены и в названии отображен субстрат для реакции и отсутствует слово "putative" (предполагаемая функция) [2].
Трипсин и его ингибиторы
При вводе в поиск "tripsin" мы получаем 11,159 результатов, причем 301 белок описан. Стоит заметить, что помимо разных изоформ трипсина одного организма и аналогичных белков других организмов, в табоице присутствует множество белков, название которых содержит "trypsin". Если добавить в запрос слово "inhibitor", получаем 2,558 белков, описано из которых лишь 203 (сюда также включены белки с названием "ингибитор сериновой протеаз", что логично, так как по классификации трипсин - именно такой фермент).
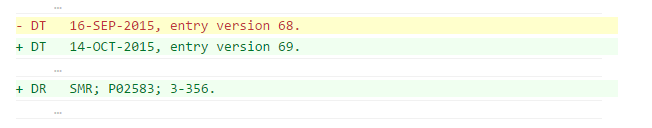
Версия 1 датирована 1986-07-21.
Последнее изменение (69 версия) 2015-12-09.
Добавлена одна ссылка на базу данных "DR"
Для того, чтобы определить, как прописаны в Uniprot нестандарные аминокислоты, модифицированные остатки и альтернативный спайсинг, введем в поиске в разделе Help ключевое слово. Например, "Non-standard residue" - появляется ссылка на страницу, в которой с примерами указано, в какой части txt-файла необходимо искать данную нестандартную аминокислоту. Нестандартные аминокислоты обозначаются меткой NON_STD в таблице свойств (feature table, FT строки).
Например, в записи P24183 есть следующая строка:

Она указывает, что 196-я аминокислота белка - селеноцистеин.

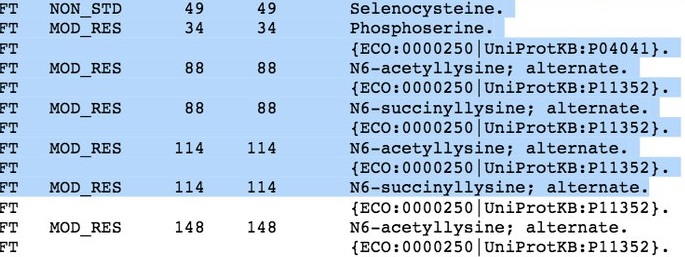
В случаях, когда селеноцистеин одновременно является частью активного сайта белка, он аннотируется дважды: как нестандартный остаток и как активный сайт. Например, в записи P07203 это описано так:

В записи P07203 можно найти модифицированные остатки, идущие сразу после нестандартной аминокислоты с номером 49. То есть, так же как и нестандартные аминокислоты, модифицированные остатки расположены в таблице свойств (FT).
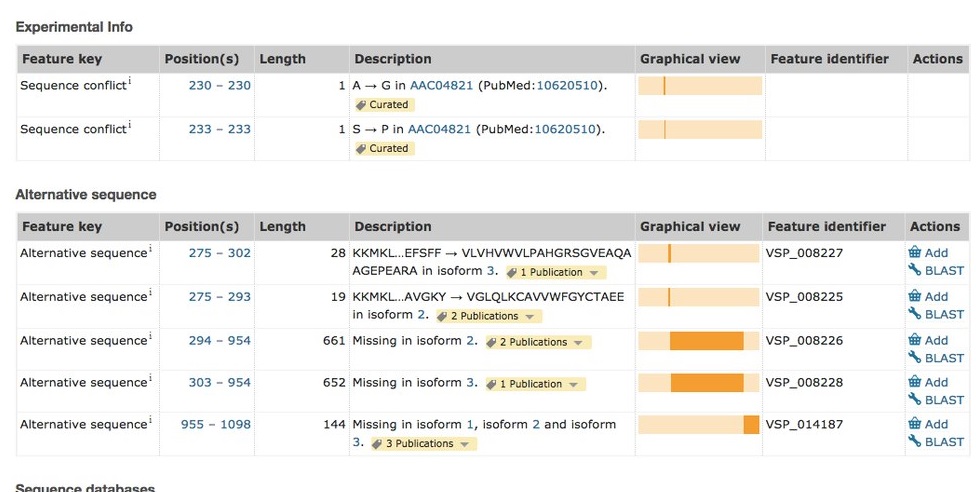
Точно так же, как в предыдущих заданиях, делаем запрос в разделе "help" и попадаем на страницу с аннотацией. В одном из примеров генов (однако попадаем мы на страницу одной из изоформ, признанной канонической и указывающейся в версиях для скачивания), продукты которого получаются через альтернативный сплайсинг, можно увидеть, что в таблицах представлены все возможные здесь белки (изоформы) и участки сплайсинга:
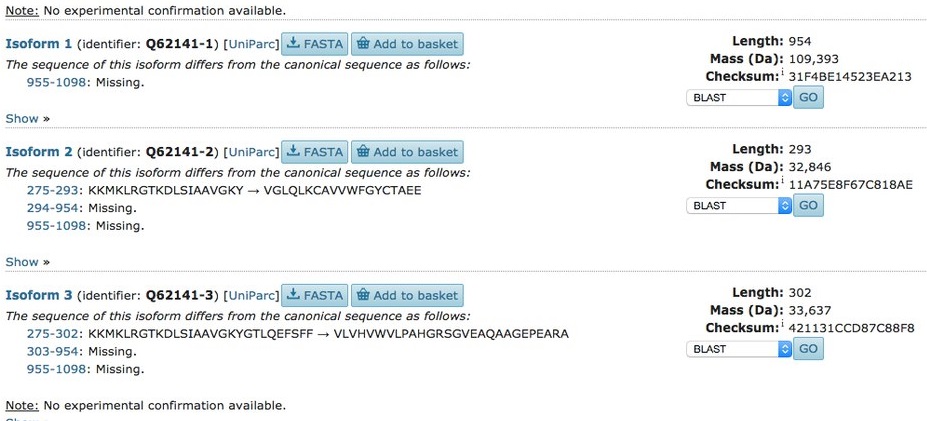
В текстовом файле в разделе "СС" можно обнаружить строку про альтернативные продукты, получающиеся с этого же гена:

Форматы записей Uniprot и RefSeq сильно оличаются, тем не менее, большая часть информации о белке присутствует в обоих файлах. Файлы записей белка в формате Uniprot и RefSeq Proteins.
Использовался один из белков кластера, существование этого белка доказано. Однако о моем белке информации в обеих базах довольно мало. Таким образом сравнение выполнено на основании информации о белке WP_003641675.1 и в пунктах, помеченных "*", сравнение выполнялось по страницам описанных белков.
1.В Refseq есть строчка с сылкой на таксономию NCBI
/db_xref="taxon:1590"
2.В Refseq более подробно расписано про сайты связывания (в данном случае сюда добавляются сайты, в который мономеры связаны в димер) и их обозначены координаты.
3.В Refseq более подробно расписана функция белка
4.*В записи Uniprot указано больше ссылок на другие базы данных (RefSeq содержит только ссылки на InterPro и GeneID).
5.*В записи Uniprot приведены альтернативные имена белка
6.*В записи Uniprot указано семейство, к которому принадлежит белок, его обычное местоположение в клетке, путь биосинтеза в котором он участвует
[1] - Uniprot // URL: http://www.uniprot.org/uniprot/M4KDD6
[2] - NCBI Taxonomy
© Yuliia Preobrazhenskaya, 2015-2016