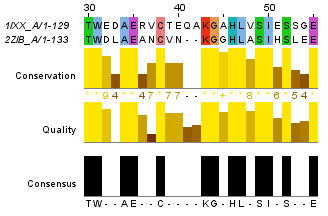
Рисунок 1. Часть глобального выравнивания, с 30 по 55 остатки
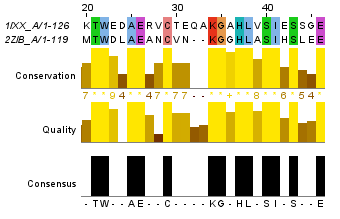
Рисунок 2. Часть локального выравнивания, с 20 по 46 остатки
Для выполнения практикума используется множественное выравнивание и PDB-файл с совмещением 2 структур. Это структуры двух последних белков, при удалении других белков и пустых колонок получается файл с парным выравниванием. Графическая интерпретация исходного множественного и парного выравниваний сохранена в проекте Jalview.
 |
 |
Рисунок 1. Часть глобального выравнивания, с 30 по 55 остатки |
Рисунок 2. Часть локального выравнивания, с 20 по 46 остатки |
Для построения глобального выравнивания, часть которого изображена на рисунке 1, я использовал программу needle из пакета EMBOSS. Исходными файлами для работы этой программы являются 2 файла в формате fasta с аминокислотными последовательностями. По умолчанию выходной файл выглядит так. Для сохранения файла в формате fasta используется опция -aform fasta (такой выходной файл).
Аналогично можно сделать локальное выравнивание, с помощью программы water. Здесь можно скачать файл с выравниваем по умолчанию, а здесь - в формате fasta.
На рисунках 1 и 2 приведены одинаковые фрагменты последовательностей, различаются только номера остатков. Есть скачать проект с этими двумя выравниваниями, то можно легко проверить полное совпадение выравниваний. Так как локальное выравнивание не обязано распространяться на всю последовательность, концевые фрагменты отрезаны.
Теперь проведем аналгоичные действия с заведомо негомологичными последовательностями. Для глобального выравнивания: файл с описанием выравнивания, в формате fasta. Те же файлы для локального выравнивания: текстовый и fasta.
Наиболее значимые характеристики всех этих выравниваний представлены в таблице 1. В колонке SeqLen - длина последовательности, AlignLen - длина выравнивания без учета гепов на конце. Количество открытий гепов указано в колонке Gaps, а их общая длина - GapLen. В колонке Ident таблицы 1 указывается количество полностью совпадающих аминокислотных остатков, Similar - сходные остатки.
Таблица 1. Сведения о полученных выравниваниях
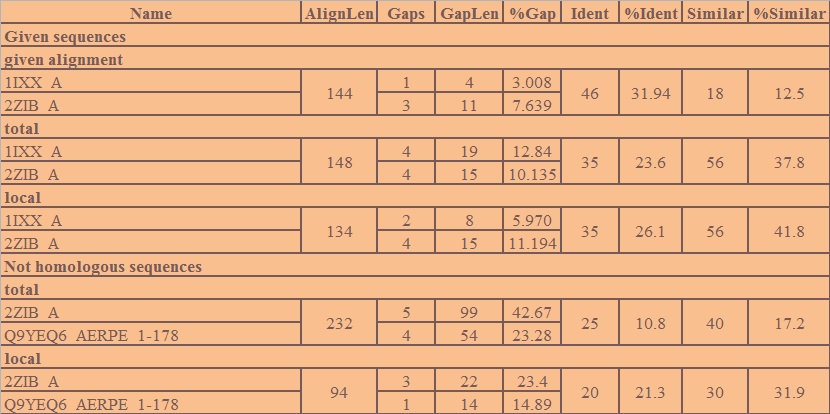
Для гомологичных последовательностей лучшим должно являеться глобальное выравнивание (однако из таблицы видим, что лучшим является локальное), для заведомо негомологичных - локальное.
Для сравнения исходного выравнивания и результата работы программы needle я в Jalview построил их выравнивание. Не изменяя выравниваний, я добавил несколько гепов во второе парное выравнивание. При этом совпала большая часть парных выравниваний. Участок с 35 по 48 с покраской BLOSUM62 изображен на рисунке 5. Это отличие заключается только в том, чему ближе аспарагин: глутаминовой кислоте (E) или аланину (A). Это не имеет значения, так как аспарагин с аланином объединяет размер радикала и отсутствие заряда. С другой стороны, глутаминовая кислота может быть легко изменена в аспарагин, если в данном месте белка заряд не имеет большого значения. Поэтому качество обоих выравниваний совпадает. Полностью результаты можно посмотреть, если скачать исходный проект. |
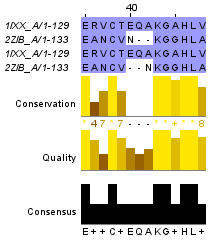 |
Рисунок 5. Участок различия между данным и полученным глобальным выравниваниями, с 35 по 48 остатки |
Для проверки правильности выравнивания (то есть вероятности гомологии) можно использовать также пространственную структуру белков. Гомологичные белки обладают сходными функциями и, значит, сходным строением. С помощью программы SupCheck можно получить скрипт, который последовательно выделяет колонки выравнивания при запуске в RasMol.
Для выравниваний, полученных изначально и с помощью needle, я проанализировал соответствие пространственных структур. В проекте Jalview в строках Correct alignment совмещенные остатки отмечены буквой "S". Колонки, которые я считаю выравненными достаточно достоверно, отмечены знаком "+".
Рассмотрим возникшие ошибки. Ошибки первого рода: аминокислотные остатки отмечены "+", но не совмещены. Ошибки второго рода: остатки совпадают в структуре, но в разных колонках или не отмечены "+". Для исходного выравнивания найдено 16 ошибок первого рода и 26 ошибок второго рода. Для глобального выравнивания (needle) 18 - первого рода и 29 - второго.
Последнее обновление: 29.02.2016