ДНК - мобильная молекула. Возможно вращение вокруг некоторых связей сахарофосфатного скелета, и из-за теплового движения может происходить сгибание, растягивание и распаривание (плавление) цепей.
Известны три формы ДНК: А-, В- и Z-
Для построения трехмерных моделей этих структур используются программы пакета 3DNA.
Таким образом были построены с помощью программы "fiber" А-, В- и Z-формы ДНК, состоящие из пятикратных повторов последовательности GATС
Полученные структуры представлены на рисунках ниже.
 |
 |
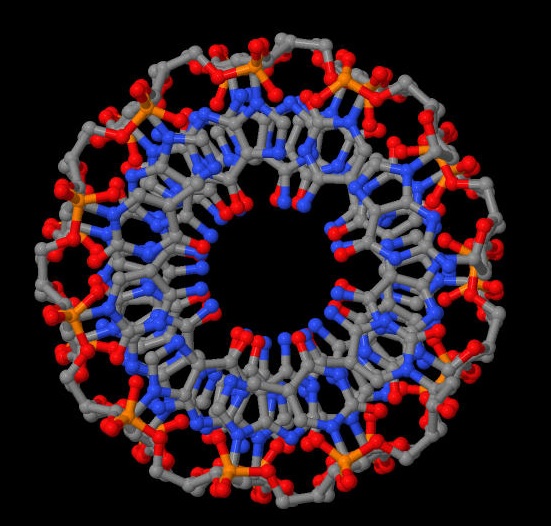
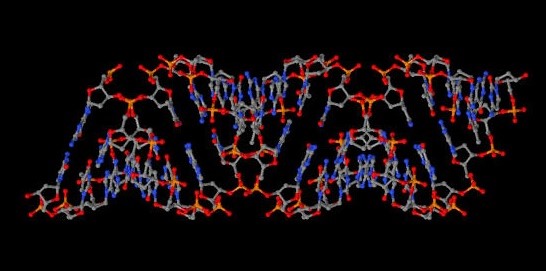
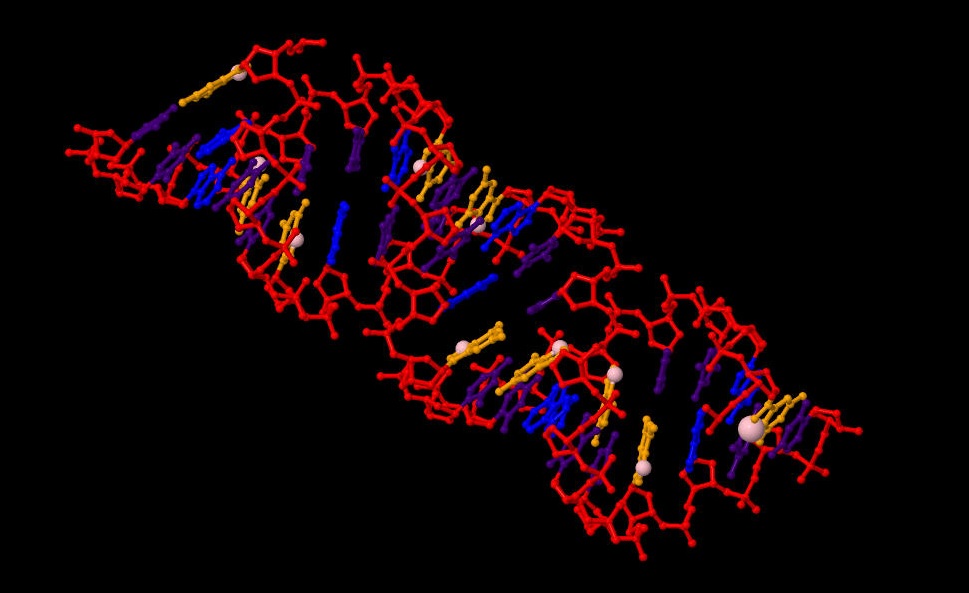
| A-форма. Образуется в средах с низким содержанием воды. |
 |
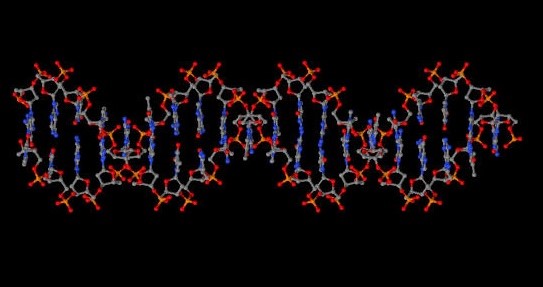 |
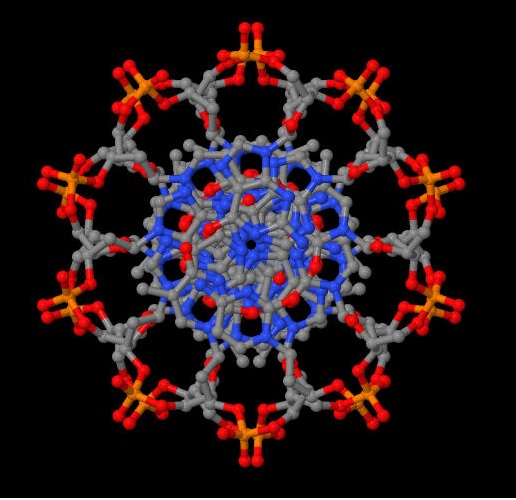
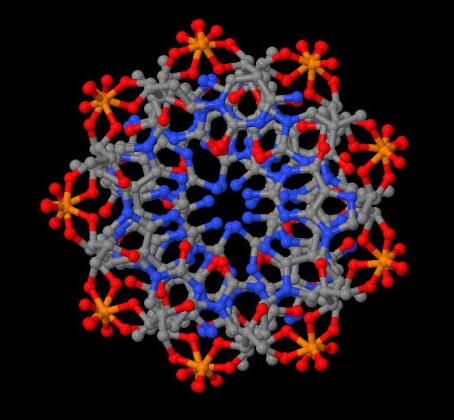
| B-форма. Её образование наиболее вероятно для случайной последовательности молекулы ДНК при физиологических условиях. |
 |
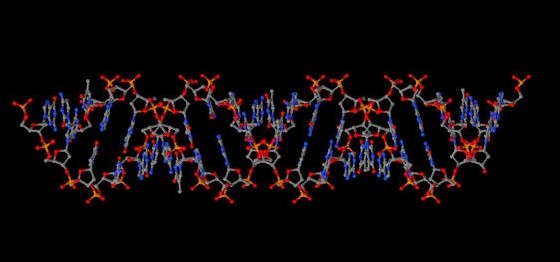 |
| Z-форма. Короткие фрагменты Z-ДНК могут быть и у прокариот, и у эукариот. Эти фрагменты Z-ДНК могут играть роль (пока неизвестно какую) в регуляции экспрессии некоторых генов или в генетической рекомбинации. |
Теперь с этими структурами можно работать в программме Jmol. Можно выделять отдельные нуклеотиды, атомы, сахарофосфатный остов и т. д.
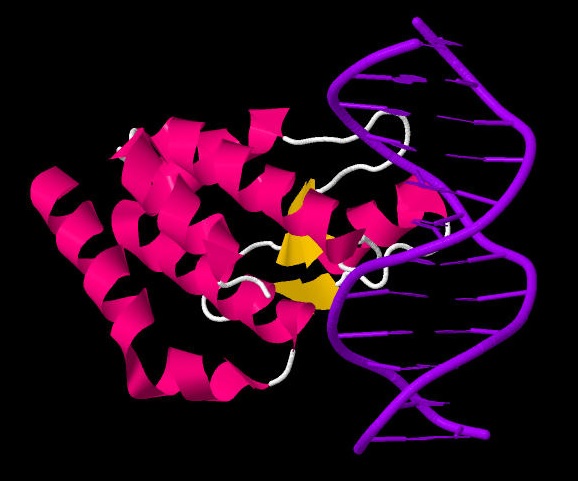
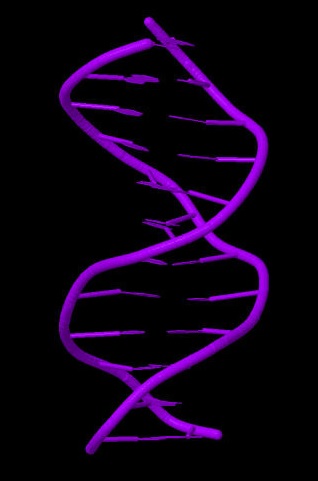
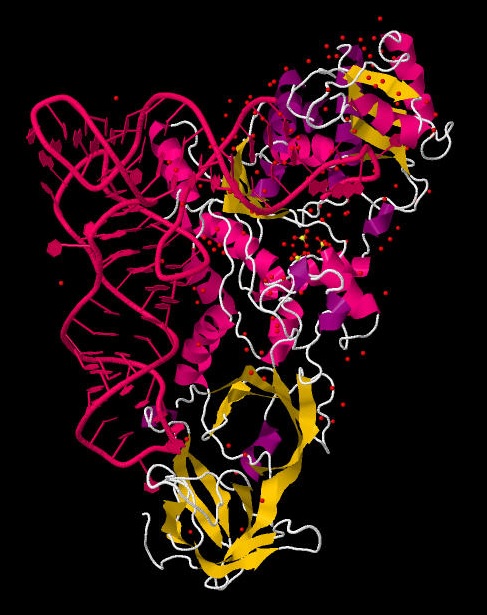
Jmol также позволяет работать с трехмерными структурами ДНК- и РНК-белковых комплексов. Изучим комплексы 1LRR и 1QTQ на предмет разрыва цепей нуклеиновых кислот. Такие разрывы могут присутствовать в реальной структуре или являются результатом ошибки рентгеноструктурного анализа и выбора множества атомов.
 |
 |
 |
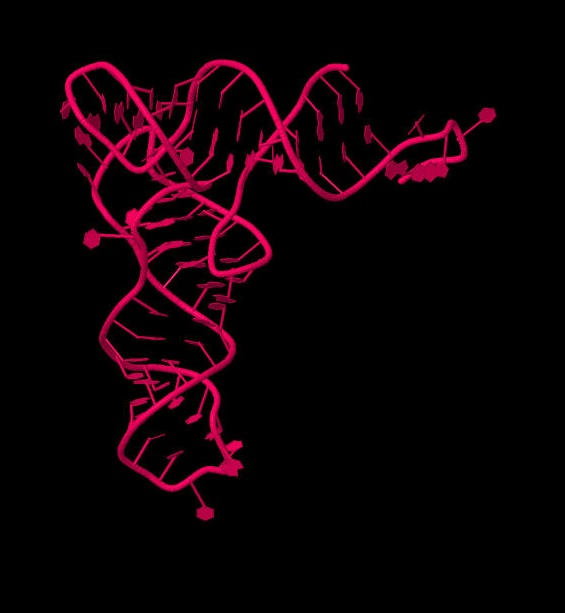 |
При рассмотрении нуклеиновой кислоты отдельно от всего комплекса видно, что разрывов в цепях нет.
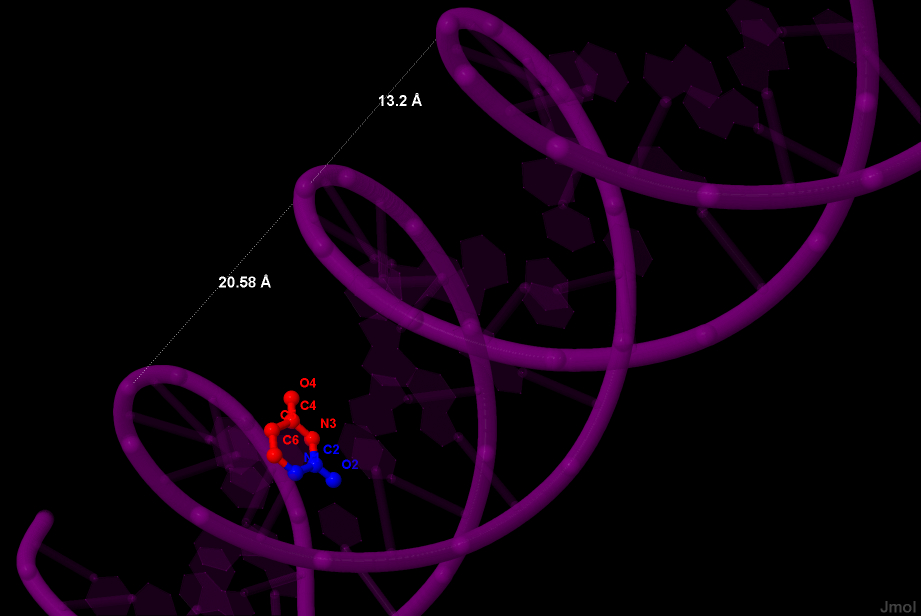
В спиралях ДНК различают так называемые малые и большие бороздки. Пуриновые и пиримидиновые обоих тяжей уложены очень близко друг к другу внутри двойной спирали, часть их атомов обращена на большую бороздку, а часть на малую. Рассмотрим ориентацию атомов тимина (7 остатка цепи A) в В-спирали ДНК.
В сторону малой бороздки обращены атомы C2, N1, O2. В сторону большой - C4, C5, C6, N3, O4.
 |
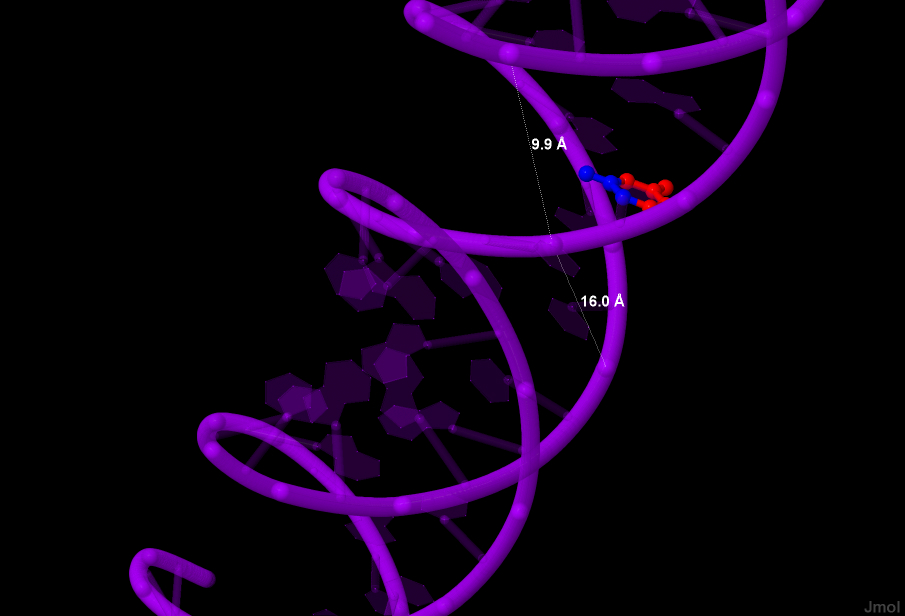 |
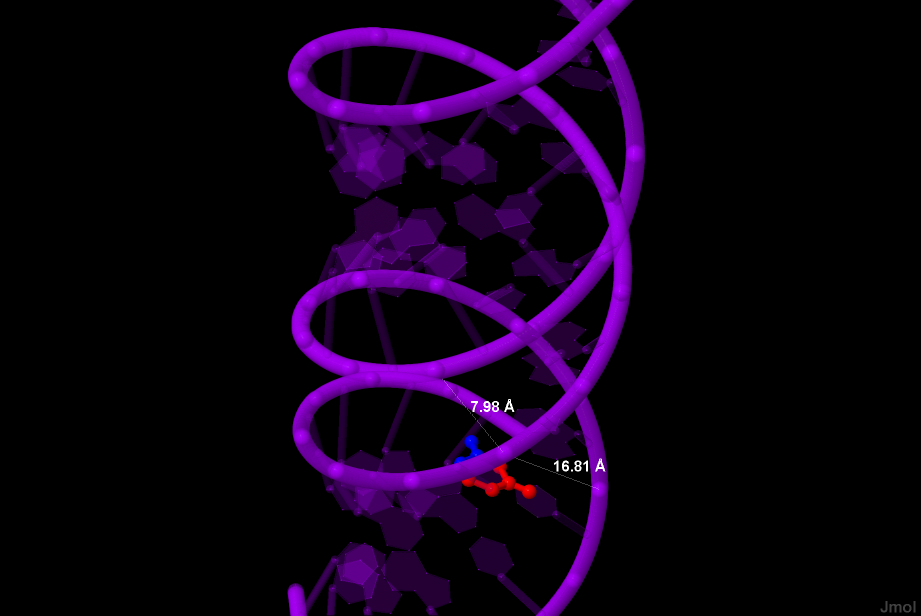
| Рис. 6. A-форма. Синим цветом выделены атомы, обращенные к малой бороздке (C4, C5, O4), красным - к большой (C2, N1, N3, O2, C6). А также измерена ширина малой б. = 7.98Å и ширина большой б. = 16.81Å | Рис. 7. Z-форма.Синим цветом выделены атомы, обращенные к малой бороздке (C2, N1, O2), красным - к большой (N3, C6, C4, C5, O4). А также измерена ширина малой б. = 9.9Å и ширина большой б. = 16Å |
| А-форма | В-форма | Z-форма | |
| Тип спирали (парвая или левая) | правая | правая | левая |
| Диаметр (Å) | ~26 | ~20 | ~18 |
| Шаг спирали (Å) | 28.03 | 33.75 | 43.5 |
| Число оснований на виток | 11 | 10 | 12 |
| Наклон оснований к оси спирали | 20° | 6° | 7° |
| Ширина большой бороздки (Å) | 16.81 | 20.58 | 16 |
| Ширина малой бороздки (Å) | 7.98 | 13.2 | 9.9 |
| Конформация сахара | С-3'эндо- | С-2'эндо- | С-2'эндо- для пиримидинов С-3'эндо- для пуринов |
| Конформация гликозидной связи | анти- | анти- | анти- для пиримидинов син- для пуринов |
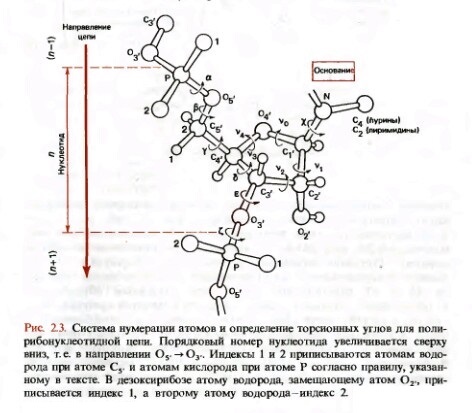
 |
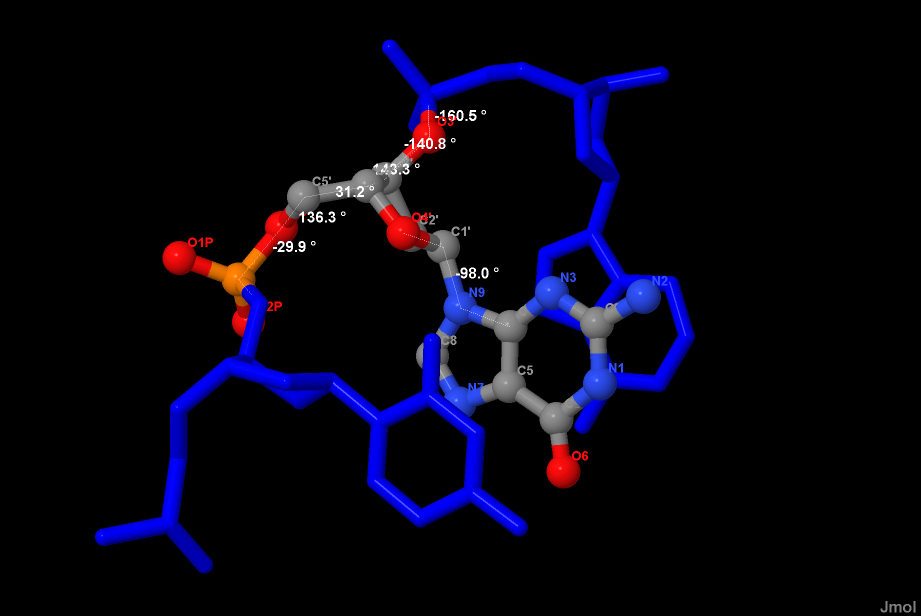 |
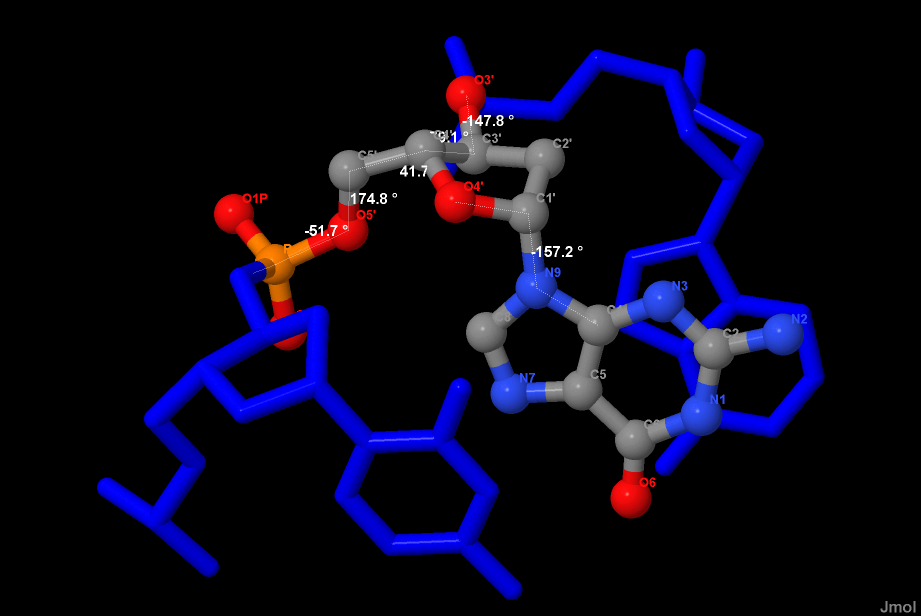
| Рис. 9. Торсионные углы в нулкотие А5 А-формы ДНК | Рис. 10.Торсионные углы в нулкотие А5 B-формы ДНК |
Значения измеренных мною углов представлены в таблице 2
| Форма | α | β | γ | δ | ε | ζ | χ |
| A | -51.7 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| B | -29.9 | 136.3 | 31.2 | 143.3 | -140.8 | -160.5 | -98 |
Данные из презентации: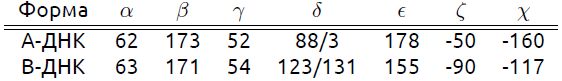
Видно, что они в той или иной степени различны.
Использованные в предудцщей части работы pdb-фалйы можно исследовать с помощбю программ пакета 3DNA. Так как пакет 3DNA работает только со старым форматом PDB, файлы нужно перевести в старый формат pdb с помощью программы remediator:
remediator --old ‘XXXX.pdb’ > ‘XXXX_old.pdb’Теперь о структурах, записанных в pdb-файлах можно получить довольно много информации с помощью команд find_pair и analyze:
find_pair -t XXXX.pdb stdout | analyzeПроанализируем сначала файлы gatc-a.pdb и gatc-b.pdb
Команда analyze выдает довольно много файлов с разной информацией о оструктуре. Нам нужны файлы gatc-а.out и gatc-а.out
В этих файлах находится информация о торисонных углах нуклеотидов. Сравним эти данные с полученными измерением вручную.
| Форма | α | β | γ | δ | ε | ζ | χ |
| A | -51.7 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| B | -29.9 | 136.3 | 31.2 | 143.3 | -140.8 | -160.5 | -98 |
Если сравните эти данные с полученными вручную, видно, что углы совпадают полностью. Это значит, что я правильно определил торсионные углы.
Применим команды find_pair и analyze к файлу 1QRQ_old.pdb. В полученном файле 1QRQ_old.out найдем информацию о торсионных углах тРНК.
| α | β | γ | δ | ε | ζ | χ |
| -23.79 | 10.68 | 68.39 | 90.33 | -141.33 | -72.76 | -126.72 |
Из значений средних торсионных углов можно сделать вывод, что данная тРНК больше всего похожа на А-форму ДНК.
Теперь применим туже команду к файлу со структурой 1LRR_old_2.pdb
В таблице ниже представлены средние значения торсионных углов, полученные с помощью excel.
| α | β | γ | δ | ε | ζ | χ |
| -50.21 | 83.85 | 37.76 | 140.25 | -92 | -84.48 | -107.51 |
C20 и G21 - нуклеотиды с максимлаьными отклонениями от средних значений.
Теперь в файле 1QTQ_old.out найдем информацию о структуре тРНК, на рисунке 11 цветом выделены пары оснований, образующие стебли тРНК.
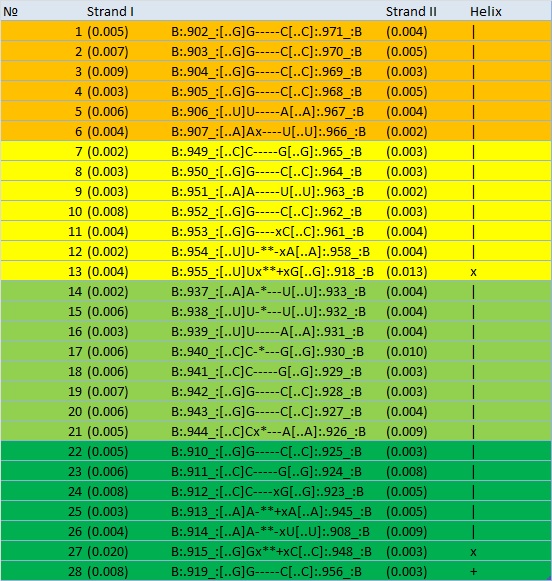
Рис. 11. Пары оснований тРНК из структуры 1QTQ, цветом выделены стебли.
Также в файле outs есть информация о неканонических парах оснований в заданной структуре. В структуре 1QTQ нашлись неканонические пары U-G, U-U, C-A, A-A.
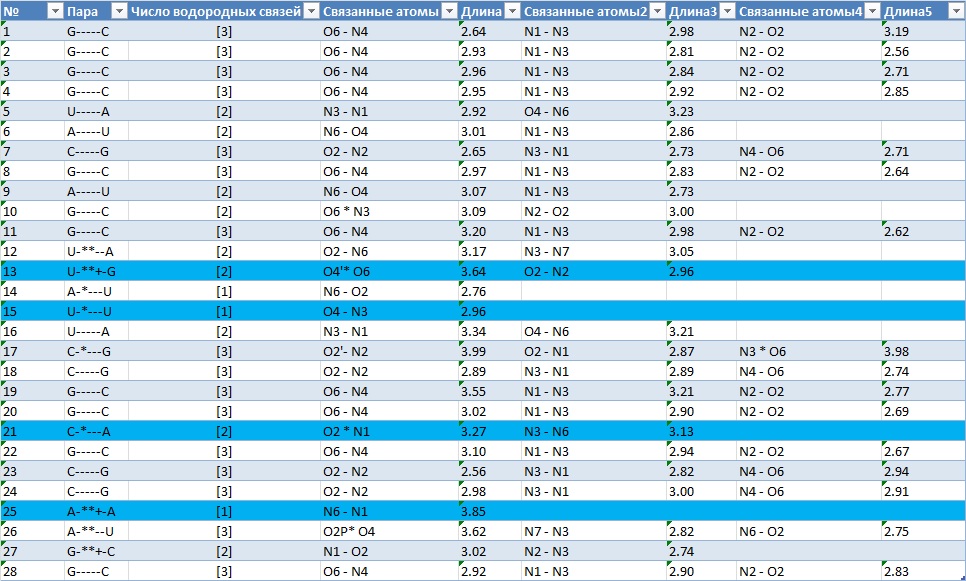
Рис. 12. Неканонические пары оснований выделены небесно голубым.
В структурах РНК встречаются так называемые стекинг взаимодействия, возникающие между расположенными друг над другом основаниями. Сила взаимодействия определяется площадью перекрывания этих оснований. Информацию о стекинг-взаимодействиях в структуре тРНК можно найти в файле out.
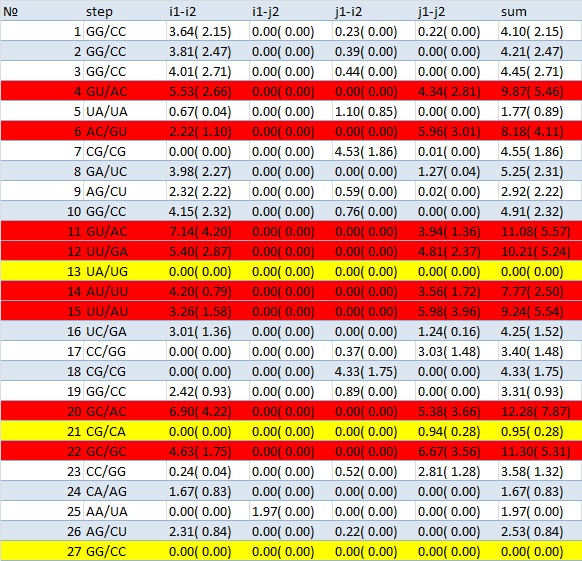
Рис. 13. Красным выделены наиболее перекрывающиеся основания, желтым - наименее перекрывающиеся.
Графическое изображение минимального и максимального перекрываний можно получить с помощью команд:
ex_str -X stacking.pdb stepX.pdb
stack2img -cdolt stepX.pdb stepX.ps
Где Х - это номер пар с наибольшим(№20)/наименьшим(№13) перекрыванием, а следовательно и наибольшим/наименьшим стекинг взаимодействием (см.рис. 13).
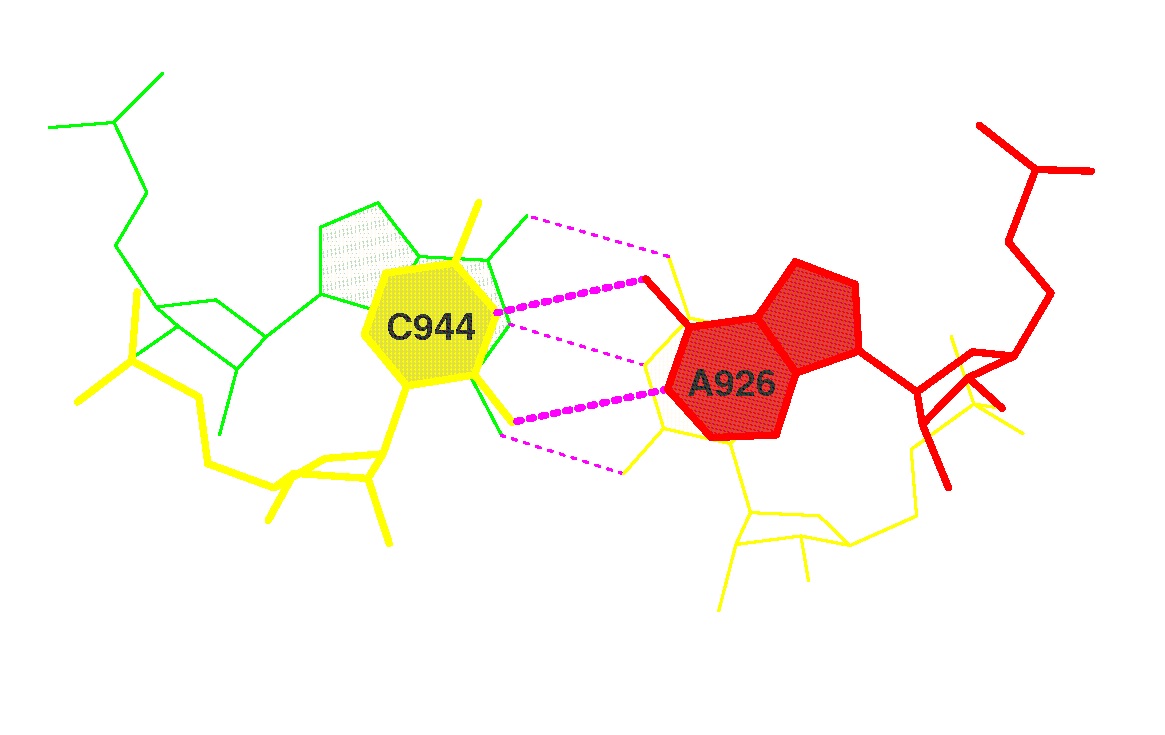
Рис. 14. Максимальное перекрывание в данной тРНК (шаг 20).

Рис. 15. Минимальное (нулевое) перекрывание в данной тРНК (шаг 13).
Последнее обновление: 28.09.2014