
Для укоренения полученного ранее дерева была использована программа retree пакета PHYLIP.

Рис. 1. Работа в программе retree.
Для того, чтобы программа укорененила дерево в среднюю точку надо ввести "М" (Midpoint root the tree).
Записав укорененное дерево в файл через опцию "W", измененив формат этого файла на .Newick, дерево было открыто в программе MEGA.
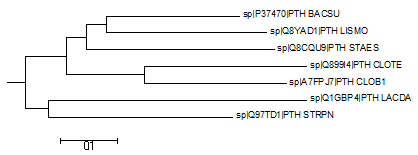
Рис. 2. Визуализация дерева в программе MEGA.
Укоренение произошло между ветвями (LACDA, STRPN) и (BACSU,CLOTE,CLOB1,LISMO,STAES).
Такое укоренение неправильное (не согласуется с данными с NCBI taxonomy), должно было укорениться в ветвь (LACDA, STRPN, BACSU, LISMO, STAES) vs (CLOTE,CLOB1).
К выравниванию пептидил-тРНК гидролаз семи выбранных видов бактерий была добавлена последовательностб того же белка из E. coli, выравнивание было проведено программой MUSCLE c последующей визуализацией в Jalview.
Дерево, полученной методом Maximum parsinomy, невозможно укоренить в среднюю точку, именно поэтому используется внешняя группа E. coli.
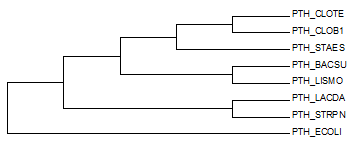
Укоренение производится в ветвь (ECOLI)vs(LACDA,BACSU,CLOTE,CLOB1,LISMO,STAES,STRPN)
 |
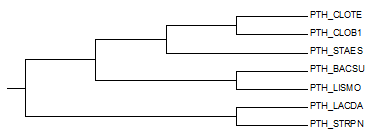 |
| Рис. 3. Неукорененное дерево с внешней группой. | Рис. 4. Укорененое дерево с внешней группой (клада Firmicutes) |
Полученное дерево воспроизводит правильное хуже, чем в предыдущем разделе (рис 2), они имеют три общие ветвь: (BACSU,LISMO)vs(others), (CLOTE,CLOB1)vs(others) и (LACDA,STRPN)vs(others).
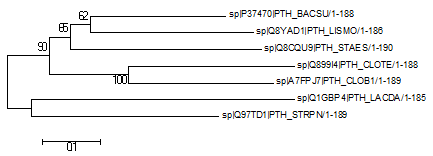
Далее был проведен бутстрэп-анализ взятых белков с чилом реплик 100. Сравним полученные деревья:
 |
 |
| Рисунок 5. Оригинальное дерево | Рисунок 6. Консенсусное дерево |
Заметим, что дерево бутстрэпа имеет все те же ветви, что и дерево при использовании внешней группы.
Последнее обновление: 9.05.2015