Филогенетическое дерево выбранных вдов можно строить по выравниванию нуклеотдных последовательностей (ранее это делал по белковым последовательностям). 16S рибосомная РНК - достаточно консервативная последовательность, которая часто используется для посторения филогенетических деревьев.
Для работы были взяты последовательности РНК 16S субъединицы рибосомы выбранных ранее бактерий с помощью базы последовательностей РНК малых субъединиц рибосом Silva. Fasta-файл с ними можно скачать здесь.
Последовательности были выровняны программой MUSCLE. Ссылка на вырванивание.
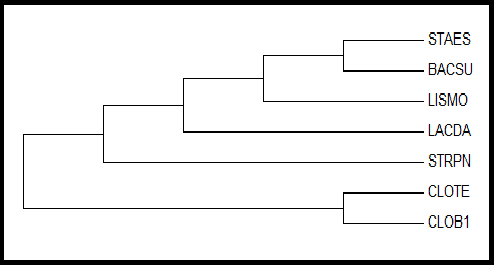
Далее с помощью программы MEGA по алгоритму Neighbour-Joining по выравниванию было посторено филогенетическое дерево.

Рис. 1. Дерево, полученное с помощью алгоритма Neighbour-Joining
Получившееся дерево соответствует не целиком полученному ранее на основе данных NCBI taxonomy, так как организмы BACSU, LISMO и LACDA, STRPN не образуют на нем отдельную ветвь.
С пмощью программы blastp среди выбранных видов были найдены последовательности предполагаемых гомологов бекла CLPX_BACSU (Evalue < 0.001). По их выравниванию с помощью алгоритма UPGMA было построено дерево.
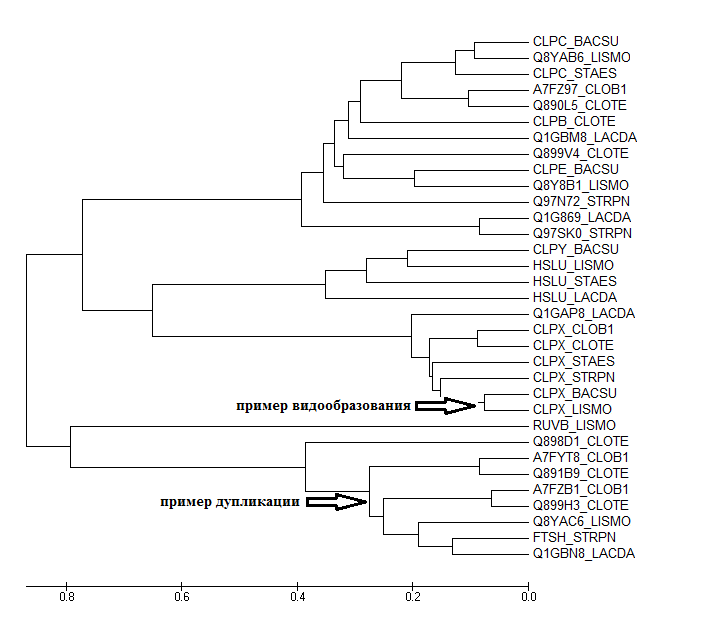
Рис. 2. Дерево гомологов CLPX_BACSU, показаны пример разделения белков в ходе видообразования (появление ортологов) и дупликации белка внутри одного организма (появление паралогов).
Примеры пар паралогов:
Q899H3_CLOTE и Q891B9_CLOTE A7FYT8_CLOB1 и A7FZB1_CLOB1
Примеры пар ортологов:
CLPX_BACSU и CLPX_LISMO CLPX_CLOTE и CLPX_CLOB1 HSLU_LISMO и HSLU_STAES
Последнее обновление: 9.05.2015