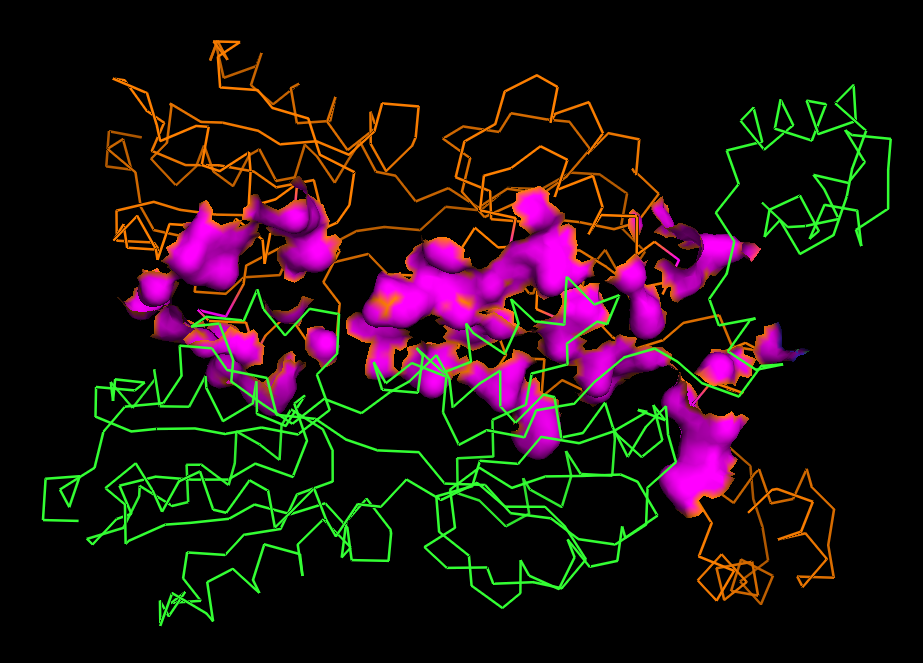
С сайта PDB была получена структура пуринового репрессора из E. coli с PDBid 1JFS. В асимметрической ячейке лежит мономер белка (не функциональная с биологической точки зрения молекула), связанный с одной цепочкой ДНК. Чтобы восстановить димер была использована команда symexp.
Для определения атомов, принадлежащих поверхности контакта был выбран порог расстояния 3.9Å.
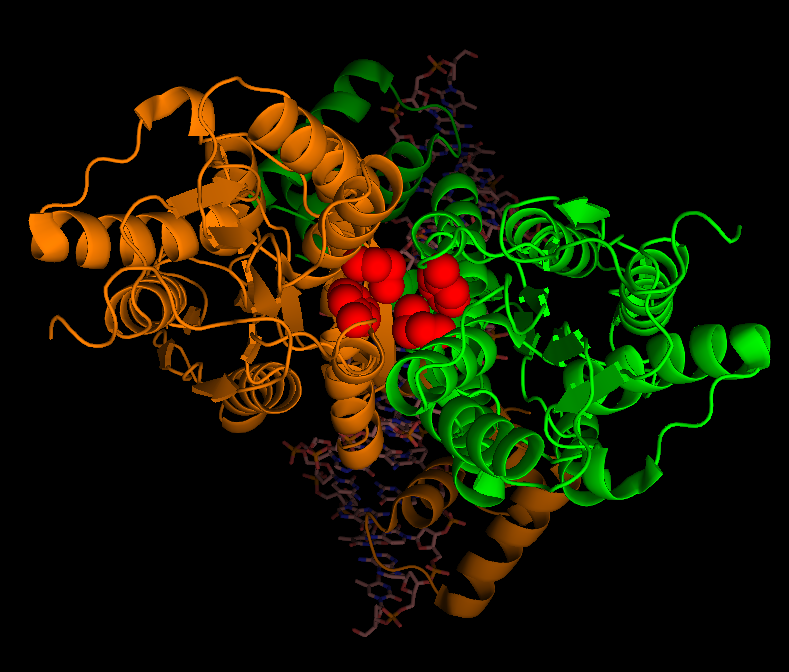
Для выбранного набора атомов можно визуализовать их поверхность (surface). На рисунках 1-3 изображено взаимодействие мономеров (рисунок 1), поверхность белка, контактирующая с ДНК (рисунок 2) и поверхность ДНК, контактирующая с рецептором (рисунок 3). Изображения были получены с помощью Pymol.

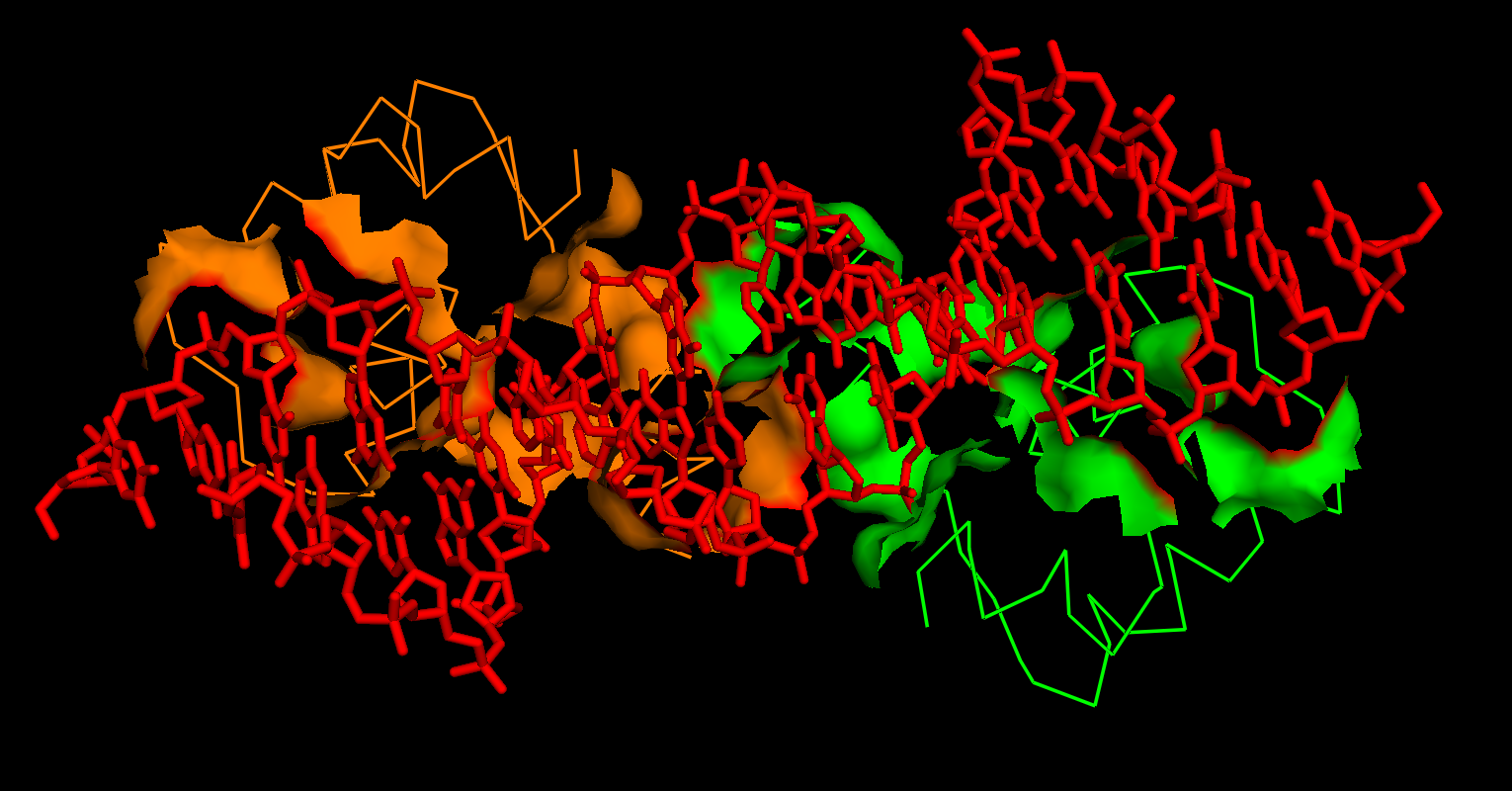
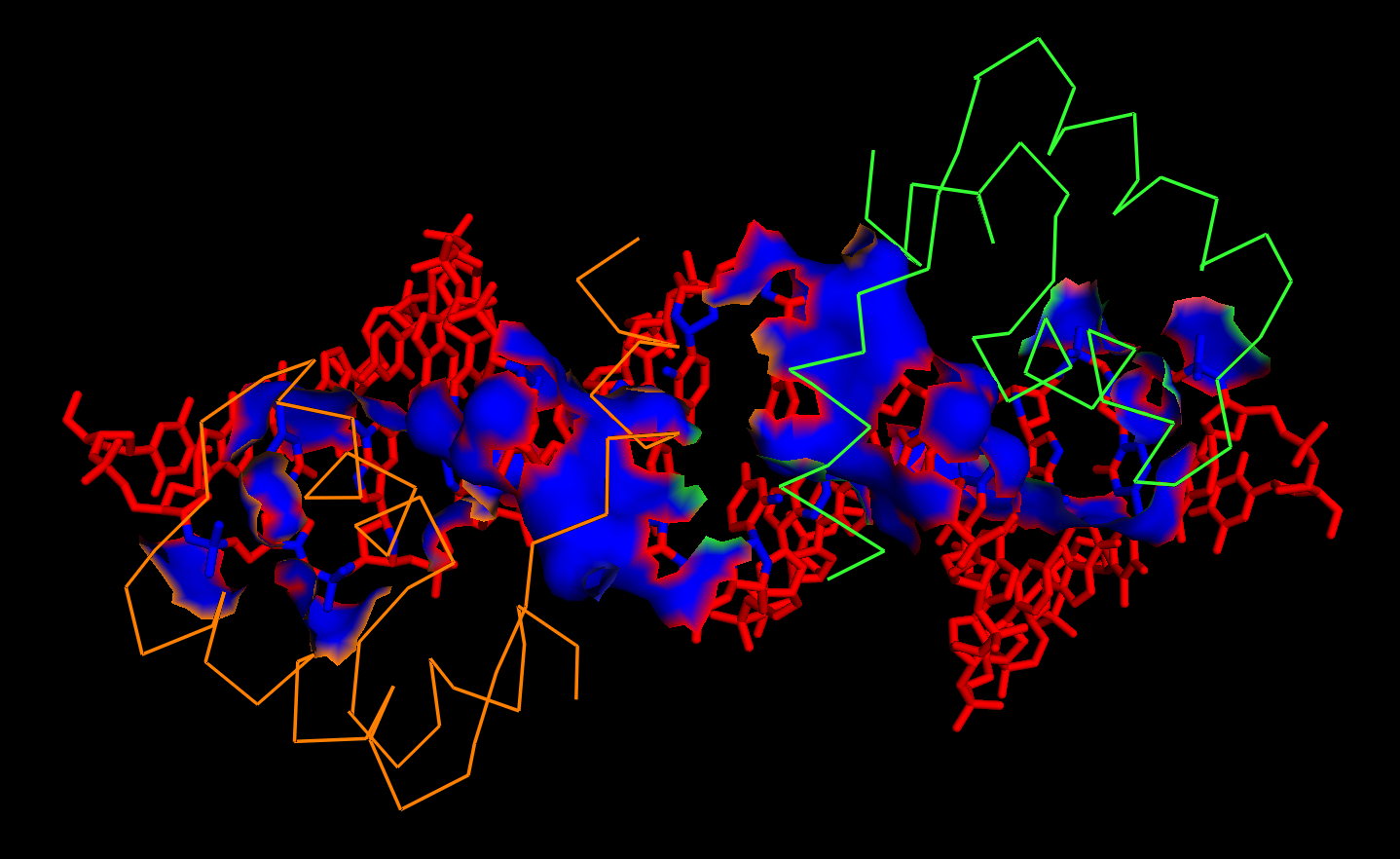
Для поиска гидрофобных кластеров в области контакта двух мономеров использовался сервис Clud. Затем с помощью Clud были найдены гидрофобные кластеры на интрерфейсе мономеров (рис. 5). Минимальный размер кластера - 10 атомов. В области контакта мономеров был определен только 1 гидрофобный кластер (порог расстояния составлял 4.0Å), состоящий из 22 углеродных атомов (рисунок 4).
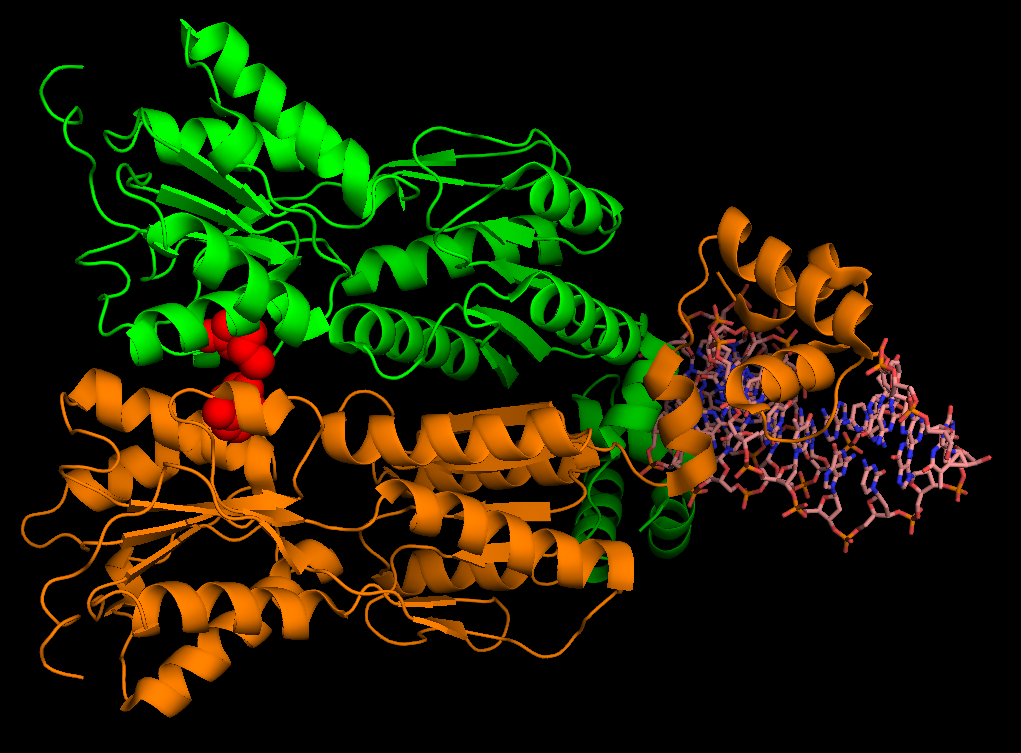 |
 |
Последнее обновление: 10.01.2017