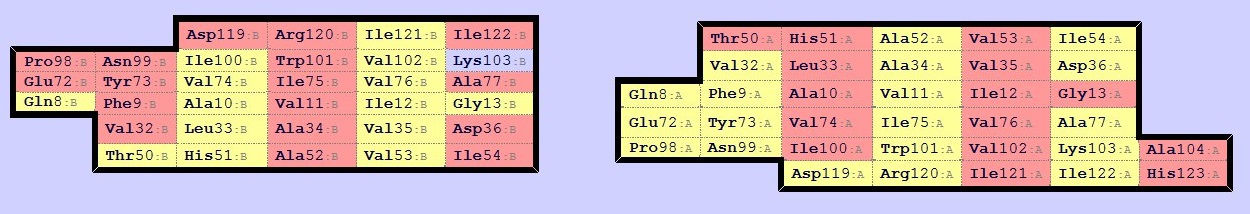
Для работы с программой по предсказанию вторичных структур (SheeP и DSSP) я выбрал белок, с которым я работал с 1 учебного семестра: калий-связывающий белок из системы ktr (идентификатор PDB 2HMV). Асимметрическая единица этого белка содержит 2 цепи (А и В), каждая из которых имеет 6 α-спиралей и 1 β-лист, состоящий из 6 тяжей.
Для предсказания вторичной структуры я использовал программу DSSP. output этой программы можно скачать тут. β-листы были предсказаны правильно, если сравнивать с аннотированными в файле pdb границами этой вторичной структуры. Предсказанные длины α-спиралей больше на несколько аминокислот (в сравнении с PDB-файлом).
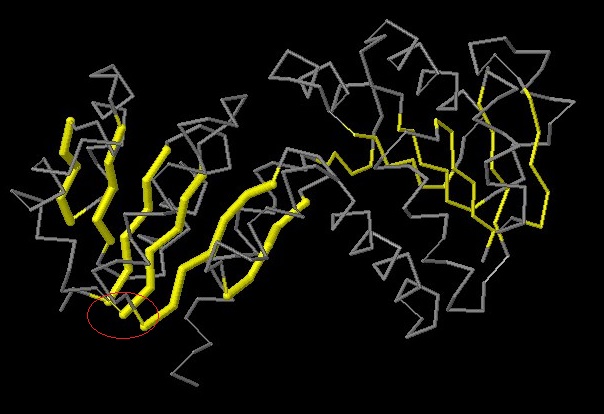
Для получения карты β-листа я использовал программу SheeP. Карта представлена на рисунке 1, а структура β-листа изображена на рисунке 2 (желтый цвет).

Если мы посмотрим на рисунок 1, то увидим, что почти все боковые цепи остатков являются гидрофобными (кроме крайних тяжей), так как β-лист лежит внутри белка.
При помощи программы Stride, установленной на kodomo, был получен список водородных связей между остатками в структуре.
При помощи PyMol были визуализированы те связи из списка, которые проходят между остатками трех тяжей (рисунок 3, слева).
 |
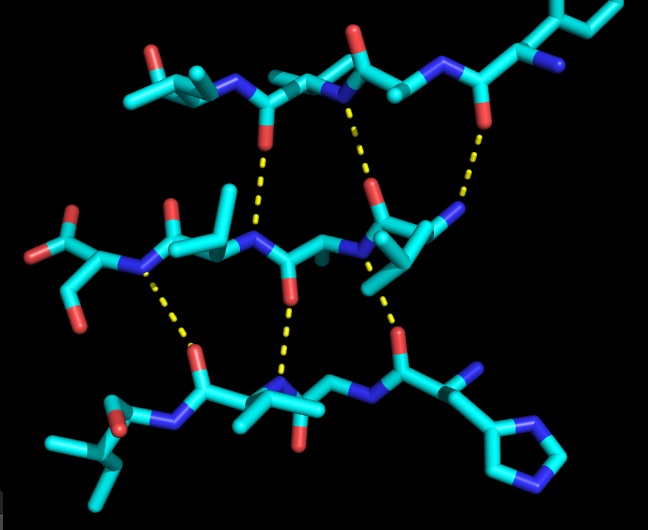 |
Хочется отметить, что предсказания SheeP не соотносятся с аннотацией PDB-файла. Это легко заметить, если сравнить рисунок 3 (левый) с рисунком 4.

Последнее обновление: 23.12.2016