Сравнение выравниваний
Для сравнения программ ClustalO и Clustal, Muscle и Mafft был использован код Лизы Плешко. Выравнивались последовательности белков из 9 практикума (ADEC_PETMO, ADEC_PARDP, ADEC_OLEA2, ADEC_META3, ADEC_ECOLU, ADEC_BACSU, ADEC_ECOLI).Результат показал, что для Clustal и ClustalO процент идентичности выравнивания составляет 64% и 62% соответсвенно, для Mafft и Muscle - 71% и 73%. Нетрудно заметить, что различия в выравниваниях совпадают с участками, где не менее 20 аминокислот у всех последовательностей не выровнялись. Остальные участки совпадают, значит можно утверждать, что выравнивания очень похожие.
Программа Muscle
Muscle - программа для множественного выравнивания, которая строит это самое выравнивание в 3 этапа. На первом этапе строится выравнивание быстрым методом (сложность О(N12*N2 + N22*N1), но я считаю, что быстрые алгоритмы имеют сложность O(logN), значит можно придумать алгоритм быстрее). Перед выравниванием также проходит подсчет расстояния по K-мерам и UPGMA кластеризация. Результат - бинарное дерево. На втором этапе подсчитывается расстояние Кимуры, потом UPGMA кластеризация и более качественное выравнивание, но не для всех последовательностей сразу, а только для близких (близких вершын дерева). На последнем этапе подбирается профиль множественного выравнивания на основе дерева из предыдущего шага. Эта стадия имеет сложность О(N13*N2)
Выравнивание структур
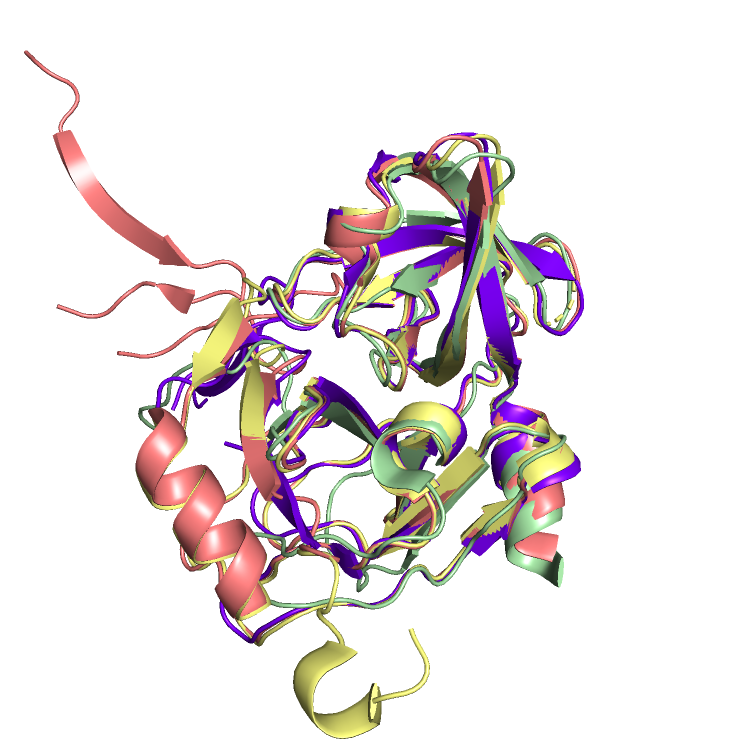
Для выравнивания были взяты белки из семейства Peptidase_S29 из Pfam. Из семейства я взял белки с PBD ID: 2XCF, 6FE6, 2QV1, 6CVX. При выравнивании был использован инструмент align в PyMOL. Структуры белков очень похожи, но белки отличаются по длине. Поэтому в некоторых последовательностях отсутствуют α-спирали или β-структуры. Тем не менее, последовательности очень похожи, высоковероятно родственны.
Выровненные белочки