Секвенирование по Сенгеру
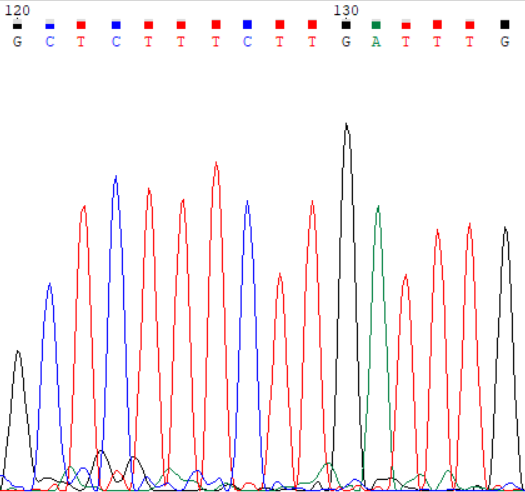
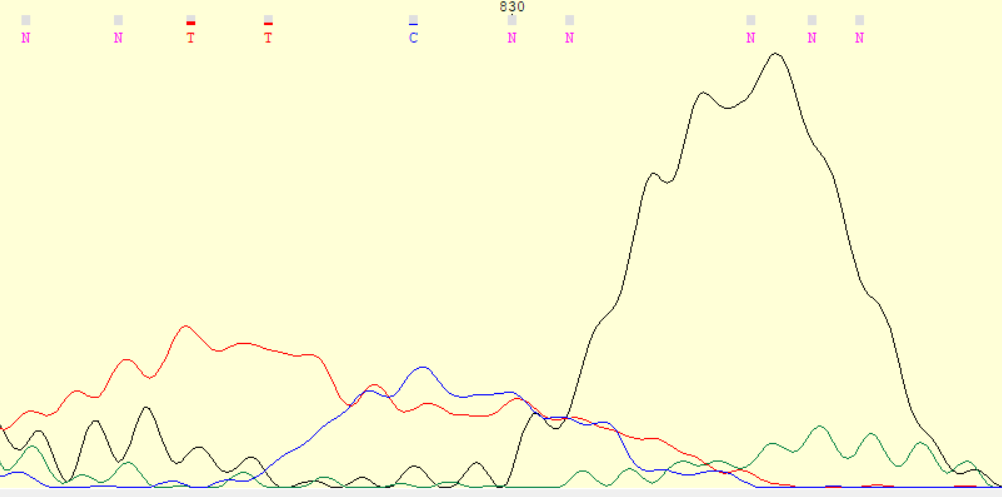
Для анаиза мне был выдан файл 06_F.ab1, длина хроматограммы в котором составляет 834 нуклеотида. Начальный трудночитаемый фрагмент кончается примерно на 25 нуклеотиде, конечный - начинается с 540 нуклеотида. Общее количество шума среднее: пики отличаются по высоте, иногда встречается сигнал двух нуклеотидов сразу. В начальной части шум распределен равномерно, в конце шума значительно больше. На рисунках 1-3 приведены примеры разного количества шума на хроматограмме.
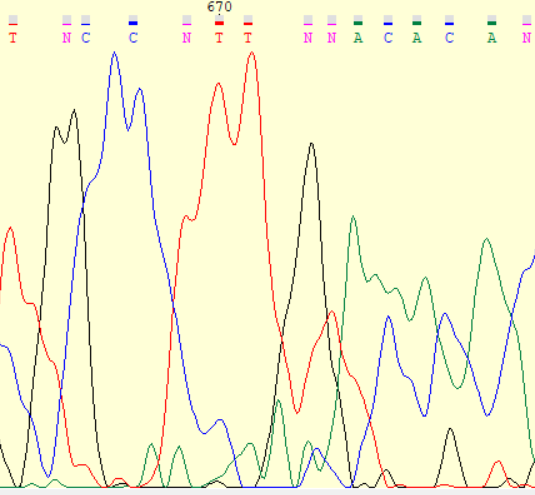
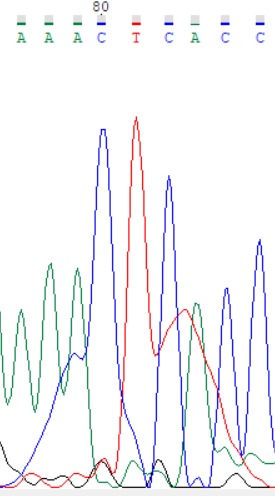
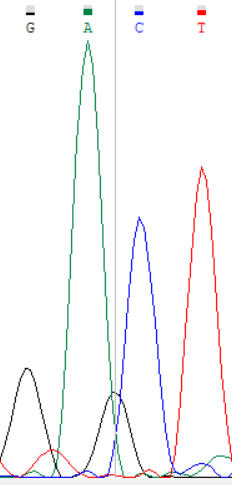
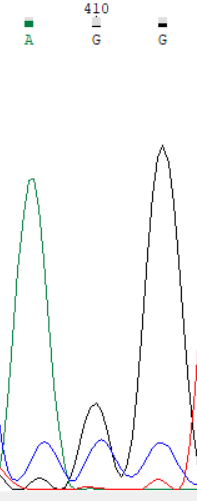
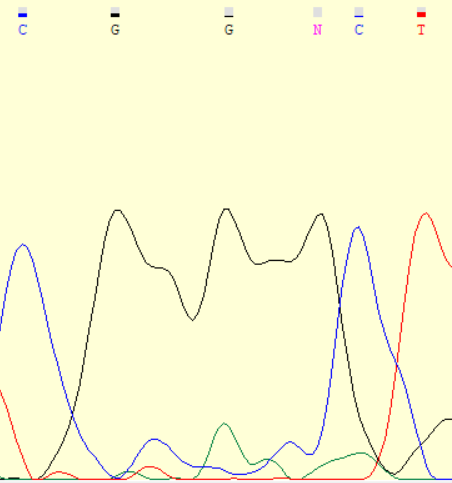
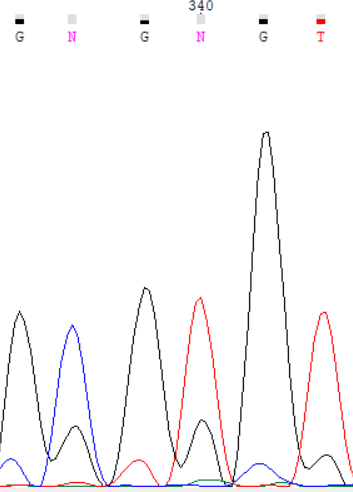
На рисунках 4-8 изображены проблемные участки хроматограммы. На четвертом рисунке изображены нуклеотиды 205-208, между 205 и 206, 206 и 207 нуклеотидами есть малые пики, что, вероятно, связано с наличием ДНК-мусора и на это не нужно обращать внимание. На пятом рисунке показаны последние 10 нуклеотидов, где видно пятно краски, этот участок нужно убрать. На шестом рисунке показан 410 нуклеотид, который имеет 2 похожих пика, что похоже на полиморфизм, но их высота сравнима с уровнем шума. В данном случае подходи либо оставить G, но с низким p-value или поставить N. На седьмом рисунке показаны нуклеотиды 501-506, между нуклеотидами довольно большое расстояние. В данном случае видно, программа не учла 2 пика G, которые следует добавить к последовательности. На последнем рисунке изображены нуклеотиды 337-342, после каждого гуанина здесь наблюдается шумовой пик того же гуанина. На полиморфизм это не похоже, так как высота каждой пары пиков отличается в три раза. На 338 позиции следует поставить C, на 340 позиции - T.