Мотивы в белках
Задание 1-2
Для выполнения этого задания я выбрал домен PF00010 (спираль-поворот-спираль ДНК связывающий домен). Выборка seed соддержит 133 последовательности. При помощи Jalview я нашел мотив E[RKQ].[RYL].{3}[ILMV][NKR].{2}[FLI].{2}L.{4}P - он встречается в выравнивании 70 раз, все находки друг под другом. Далее этот мотив был переведен в формат Prosite - E-[RKQ]-x-[RYL]-x(3)-[ILMV]-[NKR]-x(2)-[FLI]-x(2)-L-x(4)-P. При поиске этого паттерна в PROSITE по бд SwissProt получилось 497 находок. По количеству находок в обоих случаях можно сказать, что мотив не консервативен и довольно редко встречается. Это может быть связано с большой длиной мотива.
В дальнейшем я построил дерево алгоритмом NJ в Jalview и выбрал кладу из 75 последовательностей для анализа ( клада показана на филогенетическом дереве внизу красным цветом). В этой кладе я нашел мотив [RK].{2}[HK].{3}[E][RKQ].[RY]R.{2}, который встречается в 65 последовательностях из 75 и только друг под другом. Далее я попробовал найти этот мотив во всей выборке seed и оказалось, что мотив встречается только внутри этой клады. Это говорит о том, что мотив специфичен для выбранной клады.
задание 3. psi-blast
Для этого задания я методом тыка выбрал АС P17265. Это фактор регуляции трансляции Sinorhizobium meliloti, он вызывает димеризацию 70S рибосом в 100S, которая не способна осуществлять трансляцию. Этот фактор иногда присутствует в фазу экспоненциального роста. Для этого белка я запустил PSI-Blast и он сошелся за 4 итерации, находки вероятно составляют одно семейство, так как E-value у находок находится в пределах 5*10-74-3*10-18.
Табл 1, PSI-BLAST
| N итерации | Число находок ниже порога | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
| 1 | 17 | Q05886.4 | 0.046 | P17265.2 | 10-142 |
| 2 | 27 | - | - | P17265.2 | 2*10-77 |
| 3 | 28 | - | - | P17265.2 | 10-73 |
| 4 | 28 | - | - | Q5HQX7.1 | 5*10-74 |
Задание 4. MemE
Для этого задания я взял белки с доменом PF00010 (из заданий 1-2) статуса reviewed. Всего получислось 758 белков, 110 белков я отложил в выборку для обучения, 648 оставил для теста. Далее я запускал программы MEME и FIM как указано в подсказках. Результаты доступны по ссылкам: MEME, FIMO. Программа MEME нашла 4 мотива - ERRRREKJ, KLDKASIL, AISYIKSL, QQQQQQ, причем качество довольно высокое, самое высокое E-value 1.7*10-058. Программа FIMO в свою очередь нашла 2474 мотива в тестовой выборке (т.е каждый мотив почти в каждой последовательности). Можно сделать вывод, что найденные мотивы действительно имеют место быть.
ЗАДАНИЕ 5
Для этого задания я взял геном бактерии, для которой делал миниобзор на первом курсе - Bacteroides faecis strain BFG-108. Я запустил команду:
$cbcalc -s sites.ltxt -M -o GATC.txt GCF_020091505.1_ASM2009150v1_genomic.fna.gz
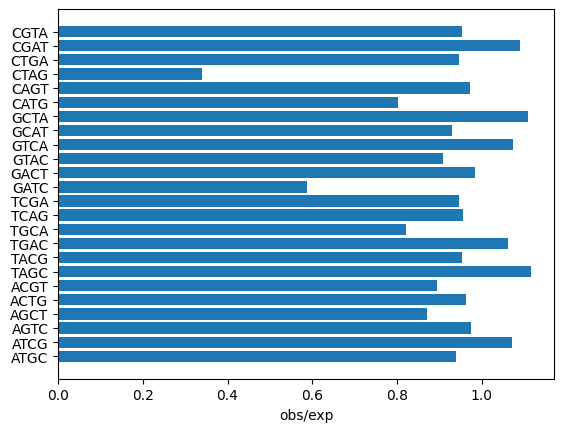
Далее я построил гистограмму контрастов в питоне, рисунок представлен ниже. Как видно, наименее представлены сайты GATC и CTAG, которые являются одним и тем же палиндромом (т.е они оба GATC, только один на прямой цепи, а второй на обратной). Итого, сайт GATC действительно недопредставлен в геноме бактерии (скажем спасибо метилазе dam, которая метилирует аденин в GATC).