HMM профиль
Выбор подсемейства
Для выполнения данного задания я выбрал домен PF08759 (Glycosyltransferase GT-D fold). Этот домен находится в C-конце и N-конце различных гликозилтрансфераз, а также содержит сайт связывания металлов. В качестве подсемейства я выбрал белки, которые содержат 2 домена - PF01501 и PF08759 (Glyco_transf_8 и GT-D соответственно), длина доменной архитектуры 682. В подсемейство попало 157 белков, но для построения HHM профиля я отобрал 44 белка. Для этого я составил выравнивание последовательностей в Jalview при помощи Mafft, и далее С помощью Remove redundancy я удалил последовательности с порогом 94%. Дело в том, что выравнивание получилось слишком хорошим (многие позиции в выравнивании имеют консервативность 90% и выше) и при пороге 90% остается меньше 30 последовательностей. Такая высокая консервативность окажет влияние на результаты в следующих пунктах.
HMM профиль
Далее я построил HMM профиль и нашел при помощи этого профиля сигналы в остальных белках подсемейства:
$hmm2build hmmout pr11.fa
$hmm2calibrate hmmout
$hmm2search --cpu=1 hmmout pr11_full.fa > hmm_results.txt
Длина HMM профиля составила 838, во всех 113 последовательностях из pr11_full.fa доменная структура была надена, причем у всех находок кроме 4 E-value равно 0, самое высокое E-value 3*10-154 (вес 516.8). Это связано с высокой консервативностью белков подсемейства. Для отрицательного контроля я решил взять 757 белков, которые содержат только домен GT-D. Для поиска сигнала я запустил следующую команду:
$hmm2search --cpu=1 hmmout pr11_ctrl.fa > hmm_results_ctrl.txt
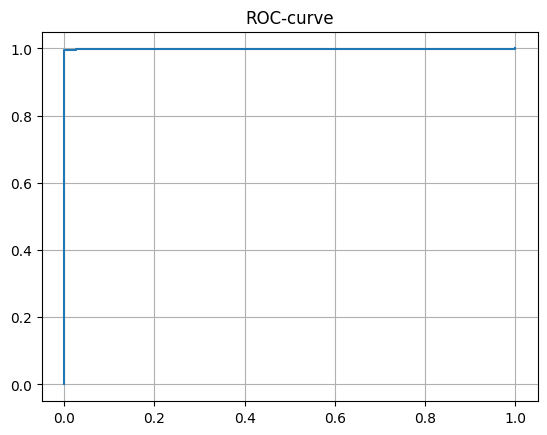
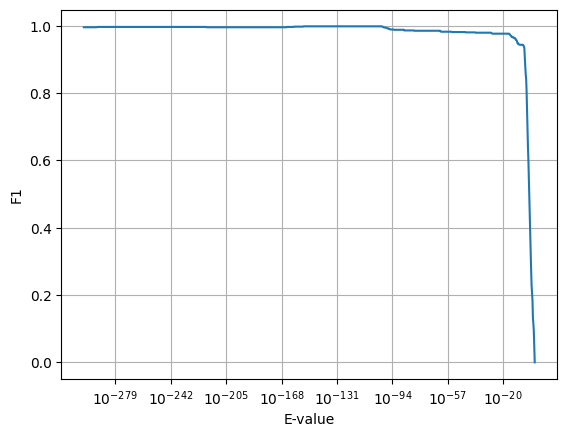
В результате одна находка с E-value 6.8e-219 (вес 734.3), а остальные имеют вес 345.5 и меньше, E-value 7.4e-102 и больше. У каждой следующей находки значение E-value растет на порядок. Уже отсюда можно заметить, что порог E-value 10-120 будет идеаделен, так как в этом случае F1-score будет равен 0.998. Для анализа результатов я использовал библиотеку sklearn, поэтому использовал также функцию classification_report при выбранном пороге, получил precision, recall и accuracy 0.99-1.00. Далее я построил ROC-кривую, которая представлена на рис 1 и график зависимости f1-score от порога по E-value. Как видно из рисунков модель очень хорошо определяет наличие доменной структуры в белках.