Трансмембранные белки
Задание 1
Для выполнения данного задания я выбрал белок Porin 2 (Порин 2, идентификатор Uniprot E3U905_PROST, идентификатор PDB 4D65). Этот белок находится в клеточной мембране Providencia stuartii. Порины это белки, которые пропускают через мембрану молекулы воды. В базе данным OPM координаты трансмембранных участков указаны для 3 одинаковых субъединиц, для всех 3 координаты одни и те же:
1( 11- 23), 2( 33- 44), 3( 53- 63), 4( 72- 82), 5( 86- 92), 6(129-135), 7( 147- 153), 8( 173- 182), 9( 186- 193), 10( 215- 224),11( 227- 234),12( 256- 263),13( 270- 276),14( 294- 302),15( 309- 315),16( 334- 341)
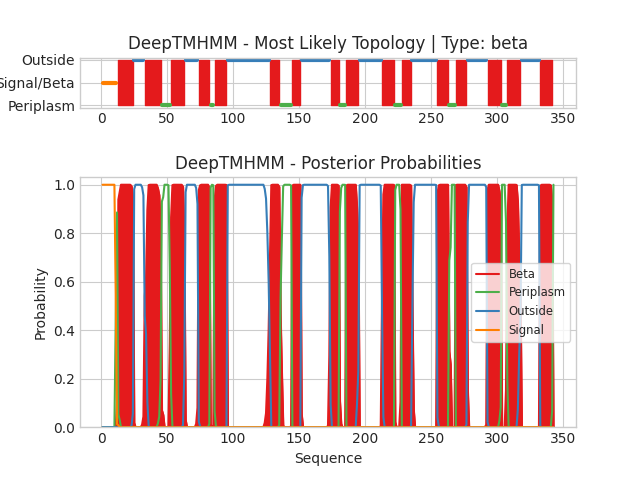
Далее я получил предсказание трансмембранных участков с помощью программы DeepTMHMM (последовательность взял из PDB). Результаты пердставлены на рисунке и в файле. На рисунке показаны вероятности того, что аминокислота в определенной позиции находится в бета-листе/снаружи мембраны/в периплазме. Как можно заметить, предсказания очень похожи. Каждый бета-тяж присутствует в обоих предсказаниях, отличаются только границы этих тяжей. Это не удивительно, ведь определить бета-лист с точностью до одной аминокислоты довольно сложно. Результаты в текстовом формате для сравнения:
1(13-24), 2(33-45), 3(53-63), 4(74-82), 5(86-95), 6(128-135), 7(145-151), 8(174-180), 9(186-195), 10(213-222), 11(228-235), 12(255-263), 13(269-277), 14(293-303), 15(308-318), 16(333-342)
Задание 2
Для этого задания мне был дан белок Y1405_YERPS (Uncharacterized MFS-type transporter YPTB1405, неохарактеризованный транспортер MFS типа бактерии Yersinia pseudotuberculosis). Для этого белка также было полученно предсказание при помощи DeepTMHMM, результаты представлены на рисунке и в файле.

Далее я сделал предсказание при помощи PPM 3.0 с параметрами:
Type of membrane - Gram-negative bacteria inner membrane
Allow curvature - no
Topology (N-ter) - in
Последний аргумент поставить на основании предсказания DeepTMHMM, но я попробовал и in и out (я считаю, что некорректно делать предсказание на основе другого предсказания и сравнивать результат), результат получился одинаковый. Выдачу программы можно скачать по ссылке. В данном случае опять все бета-листы были предсказаны обеими программами, но разница в границах бета-листов уже больше. Это может быть связано с тем, что структура белка предсказана AlphaFold. То есть ошибка при использовании PPM складывается тз ошибки собственно PPM и ошибки AlphaFold. Сравнение результатов, PPM и DeepTMHMM соответственно:
1(8-33), 2(40-61), 3(71-90), 4(94-115), 5(133-152), 6(160-177), 7(204-227), 8(236-256), 9(266-284), 10(288-306), 11(326-345), 12(350-369)
1(9-29), 2(30-44), 3(75-90), 4(95-115), 5(132-152), 6(160-178), 7(204-223), 8(237-255), 9(266-284), 10(288-308), 11(325-345), 12(352-369)