Мини-обзор генома и протеома бактерии Thermus oshimai JL-2
Каримова К.М.
Факультет биоинженерии и биоинформатики, Московский Государственный Университет имени М.В.Ломоносова, Москва
РЕЗЮМЕ
Данный мини-обзор представляет из себя работу с геномом и протеомом термофильной бактерии Thermus oshimai JL-2 с использованием биоинформатического пакета EMBOSS, программирования на языке Python и функционала электронных таблиц. Полученные результаты по возможности объясняются различными биоинформатическими закономерностями.
1. ВВЕДЕНИЕ
Thermus oshimai – неподвижная грамотрицательная неспорообразующая свободноживущая бацилла. Является факультативным анаэробом, денитрификатором, обитает в горячих источниках. Оптимальная температура для жизнедеятельности 70°C (Murugapiran at al., 2013).
Исследование по культивированию гетеротрофных денитрификаторов из высокотемпературных источников (Грейт-Бойлинг-Спрингс и Сэнди-Спринг-Уэст в США) привело к выделению большого количества штаммов бактерий, принадлежащих к Thermus thermophilus и T. oshimai, включая T. oshimai JL-2 (Hedlund at al., 2011). Полная последовательность генома T. oshimai JL-2 была представлена в 2012 году. Анализ генома показал весьма универсальные гетеротрофные способности бактерии, а так же подтвердил ее фенотип неполного денитрификатора (Murugapiran at al., 2013).
Важность этого организма в биогеохимическом цикле азота и его потенциал в качестве источника ферментов для применения в биотехнологии говорят о том, насколько ценным ресурсом является полная последовательность генома T. oshimai JL-2 как для фундаментальных, так и для прикладных исследований (Murugapiran at al., 2013).
В настоящем мини-обзоре рассматриваются и анализируются стандартные данные о геноме и протеоме T. oshimai JL-2 и приводятся статистические данные. Исследуются повторяющиеся последовательности в геноме, появление которых нельзя объяснить случайностью.
2. МАТЕРИАЛЫ И МЕТОДЫ
Данные по геному исследуемой бактерии были взяты с сайта Национального Центра Биотехнологической информации (NCBI) [1]. Для анализа данных использовались электронные таблицы Google Sheets [9], биоинформатический пакет EMBOSS, установленный на kodomo, и программа, написанная на языке Python [6].
3. РЕЗУЛЬТАТЫ
3.1 Описание стандартных данных о геноме
Геном T. oshimai JL-2 включает одну кольцевую хромосому, кольцевую мегаплазмиду pTHEOS01 и кольцевую плазмиду меньшего размера pTHEOS02 (Таблица 1) [2]. GC состав довольно высокий (68.5%) [2], что характерно для термофиллов, так как им необходима устойчивая к денатурации ДНК (между A и T двойная связь, а между G и C - тройная) в таких экстремальных условиях обитания.
| ДНК | Длина (п.н) | GC состав |
|---|---|---|
| Хромосома | 2072393 | 68.5 % |
| Плазмида pTHEOS01 | 271713 | 68.5 % |
| Плазмида pTHEOS02 | 57223 | 68.5 % |
С помощью fasta-файла последовательности генома [3] и программы [6] можно определить нуклеотидный состав ДНК (Таблица 2). Данные показывают, что для исследуемой бактерии выполняется второе правило Чаргаффа – число букв A примерно равно числу букв T (49.96% и 50.04% соответственно от суммы A+T), а число букв G примерно равно числу букв C (50.05% и 49.95% соответственно от суммы G+C) в последовательности одной цепочки геномной ДНК.
| ДНК | A | T | G | C |
|---|---|---|---|---|
| Хромосома | 325635 | 326061 | 710943 | 709754 |
| Плазмида pTHEOS01 | 42305 | 43013 | 92985 | 93410 |
| Плазмида pTHEOS02 | 9317 | 8748 | 20027 | 19131 |
| Всего | 377257 | 377822 | 823955 | 822295 |
3.2 Статистические данные о белках протеома
Были проанализированы количества и процентные содержания таких групп белков: рибосомальные, гипотетические и транспортные – по данным CDS [9, list “CDS”] таблицы особенностей генома [4] (Таблица 3). Среди рибосомальных белков нет генов с одинаковыми названиями [9, list “ribosomal proteins”].
| Белки | Количество | Процент от всех белков |
|---|---|---|
| Рибосомальные белки | 54 | 2.21 % |
| Гипотетические белки | 376 | 15.41 % |
| Транспортные белки | 218 | 8.93 % |
В электронных таблицах была построена гистограмма длин белков (Диаграмма 1) и подсчитаны некоторые статистические параметры для этого распределения (Таблица 4) [9, list “histogram”].
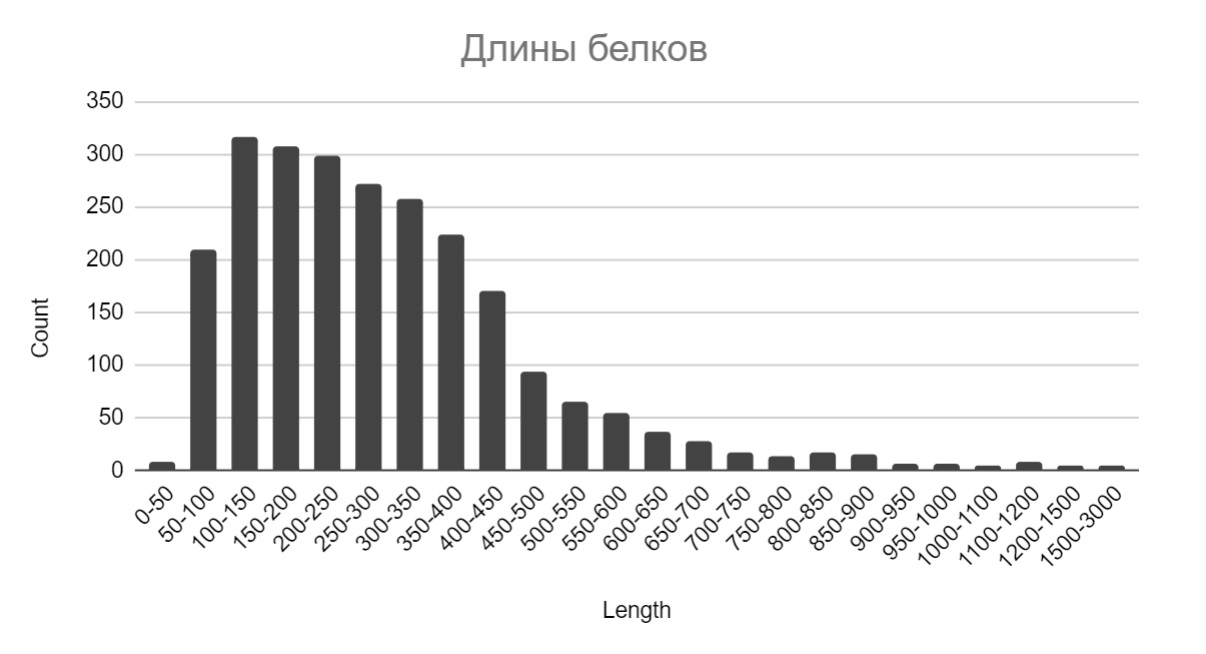
| Средняя длина | 301 |
|---|---|
| Стандартное отклонение | 198.2 |
| Медиана | 261 |
| Минимальное значение | 27 |
| Максимальное значение | 2676 |
С помощью электронных таблиц [9, list “some tables”] было установлено распределение белок-кодирующих генов по цепям ДНК (Таблица 5). По рассчитанной статистической значимости мы можем сделать вывод, что гены по цепям распределяются случайно (уровень значимости >0.01, для всех ДНК, кроме плазмиды pTHEOS02).
| ДНК | «+» цепь | «-» цепь | Статистическая значимость |
|---|---|---|---|
| Хромосома | 1106 | 1013 | 0.046 |
| Плазмида pTHEOS01 | 121 | 132 | 0.530 |
| Плазмида pTHEOS02 | 55 | 13 | <0.001 |
| Всего | 1282 | 1158 | 0.013 |
3.3 Статистические данные о генах РНК
Всего в геноме 60 генов РНК [9, list “RNA”], что составляет 2.32% от всех генов. Генов тРНК – 51, рРНК – 6 (Таблица 6). В геноме представлено по два гена каждого типа рРНК, образующих основу рибосом прокариот (5S, 16S, 23S). Вероятно, они представлены в одинаковой кратности для соотношения их продуктов 1:1:1, ведь в рибосоме присутствуют по одной молекуле каждого типа рРНК.
| РНК | Количество | Процент от всех РНК |
|---|---|---|
| тРНК | 51 | 85 % |
| рРНК | 6 | 10 % |
3.4 Повторяющиеся последовательности в геноме
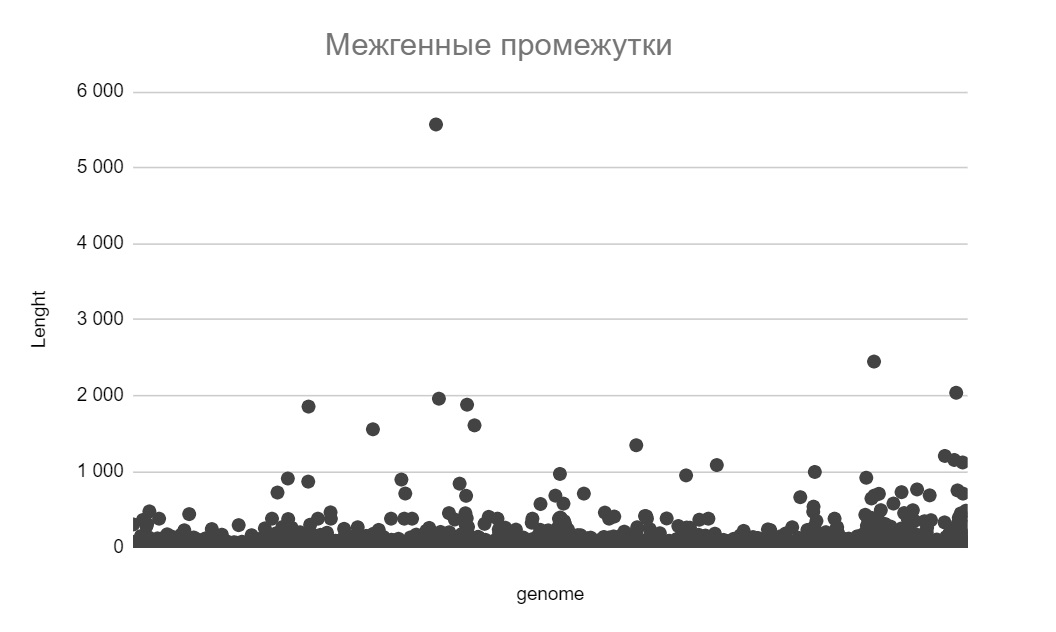
С помощью пакета EMBOSS была найдена последовательность “CGGTCCATCCCCACGGGCGTGGGGACTAC” длиною 29 нуклеотидов, которая встречается в геноме 87 раз (все из которых расположены на “+” цепи). При чем повторы представляют собой длинный одиночный кластер на хромосоме [7]. Проанализировав межгенные промежутки [9, list “intergenic gaps”] (Диаграмма 2), расположение повторов и аннотацию к геному [5], я поняла, что наткнулась на CRISPR повтор. Это самый большой (~4500 п.н.) из пяти CRISPR повторов в геноме, и расположен он в самом большом межгенном промежутке (~5600 п.н.).
Справка. CRISPR (от англ. Clustered Regularly Interspaced Short Palindromic Repeats) – короткие палиндромные повторы, регулярно расположенные группами. Между повторами располагаются уникальные последовательности примерно той же длины – спейсеры. Спейсеры – это “кусочки вирусных геномов”, не что иное, как иммунная память, которая может передаваться по наследству. CRISPR/Cas-системы (cas-гены (от англ. CRISPR-associated – сцепленные с CRISPR)) обеспечивают противовирусный иммунитет у архей и бактерий (ссылка).
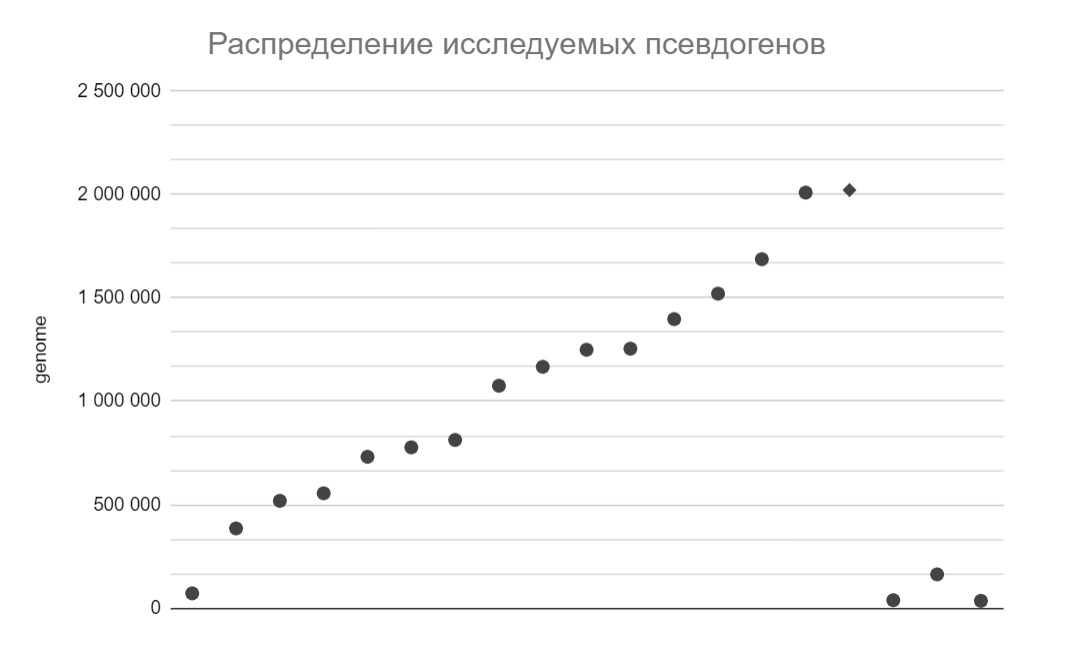
Таким же способом была найдена достаточно длинная повторяющаяся последовательность “GGGGAAGGTGGTGGTGGG GGACGCGGGGTACCTGTACCCGGAG” длиною 43 нуклеотида, повторяющаяся в геноме 19 раз (10 раз на “+” цепи и 9 - на ”-” цепи) [8]. Сравнив расположение повторов с расположением генов, я выявила интересную закономерность: эта последовательность расположена на одном и том же расстоянии от старт кодона 19 генов, из которых только один функциональный (и эта последовательность входит в его состав), а все остальные – псевдогены (с преждевременным стоп-кодоном, в которые эта последовательность не входит) [9, list “pseudo genes”]. При чем эти гены распределены по геному равномерно (Диаграмма 3). Вероятно, все эти гены являются результатом дупликации и дивергенции. Отсутствие между ними других общих длинных последовательностей можно объяснить большим количеством мутаций, так как псевдогены не функциональны и по ним не идет отбор.
Справка. Псевдогены (англ. pseudogenes) – нефункциональные аналоги структурных генов, утратившие способность кодировать белок и не экспрессирующиеся в клетке (ссылка).
СОПРОВОДИТЕЛЬНЫЕ МАТЕРИАЛЫ
- Данные NCBI по геному бактерии T. oshimai JL-2
- Описание генома с NCBI
- Fasta-файл последовательности генома с NCBI
- Таблица особенностей генома с NCBI
- Аннотация к геному с NCBI
- Программа, написанная на Python, для определения нуклеотидного состава ДНК
- Встречи в геноме повтора из 29 нуклеотидов
- Встречи в геноме повтора из 43 нуклеотидов
- Таблицы Google Sheets
СПИСОК ЛИТЕРАТУРЫ
- Murugapiran SK, Huntemann M, Wei C-L, Han J, Detter JC, Han C, … Hedlund BP (2013). Thermus oshimai JL-2 and T. thermophilus JL-18 genome analysis illuminates pathways for carbon, nitrogen, and sulfur cycling. Standards in Genomic Sciences, 7(3), 449–468. doi:10.4056/sigs.3667269
- Hedlund BP, McDonald AI, Lam J, Dodsworth JA, Brown JR, Hungate BA (2011). Potential role of Thermus thermophilus and T. oshimai in high rates of nitrous oxide (N2O) production in ∼80 °C hot springs in the US Great Basin. Geobiology, 9(6), 471–480. doi:10.1111/j.1472-4669.2011.00295.x
- Статья на Биомолекуле "CRISPR-системы: иммунизация прокариот"
- Статья в Википедии "Псевдогены"