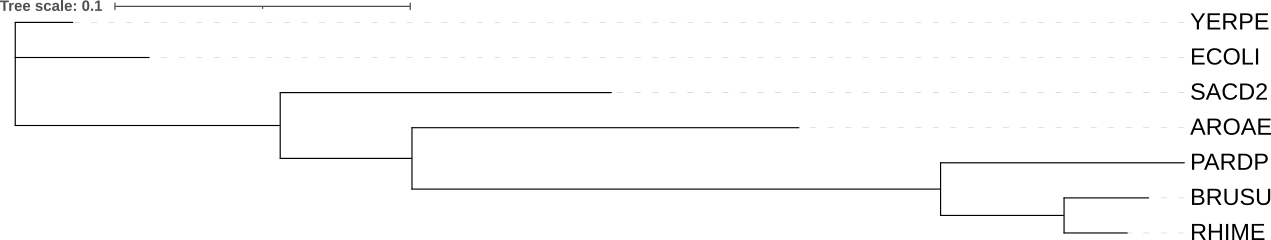
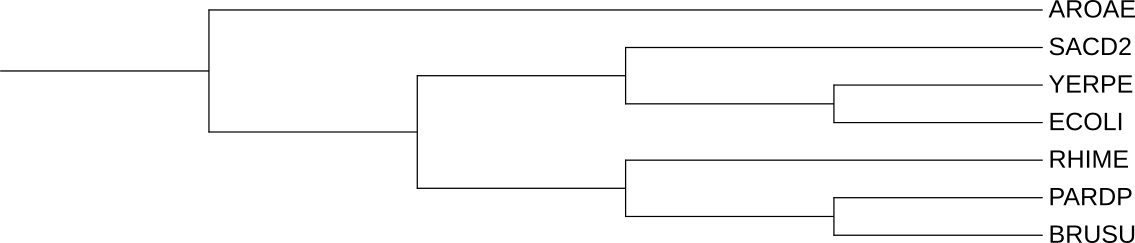
Филогенетическое дерево
Отобранные бактерии
| Название | Мнемоника |
|---|---|
| Aromatoleum aromaticum | AROAE |
| Brucella suis | BRUSU |
| Escherichia coli | ECOLI |
| Paracoccus denitrificans | PARDP |
| Rhizobium meliloti | RHIME |
| Saccharophagus degradans | SACD2 |
| Yersinia pestis | YERPE |
Скобочная формула дерева
((PARDP, (BRUSU,RHIME)), (AROAE, (SACD2, (ECOLI,YERPE))));
Изображение дерева
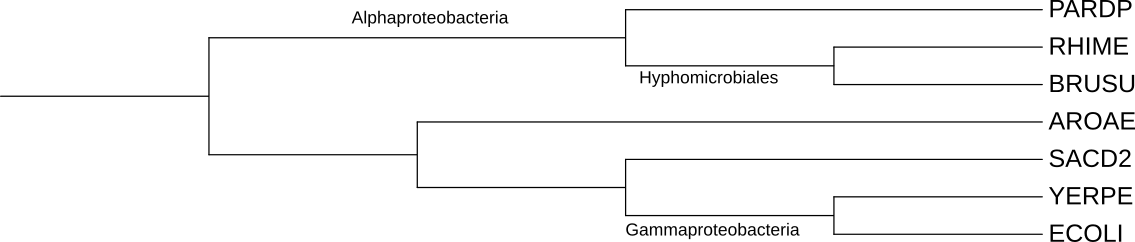
Нетривиальные ветви дерева
{BRUSU, RHIME} vs {PARDP, AROAE, SACD2, ECOLI, YERPE}
{BRUSU, RHIME, PARDP} vs {AROAE, SACD2, ECOLI, YERPE}
{BRUSU, RHIME, PARDP, AROAE} vs {SACD2, ECOLI, YERPE}
{BRUSU, RHIME, PARDP, AROAE, SACD2} vs {ECOLI, YERPE}
Реконструкция филогении
С помощью Taxonomy NCBI были подписаны некоторые нетривиальные ветви на дереве отобранных бактерий (рис. 1).
Для реконструкции филогенетического дерева была взята функция белка RL21 (один из белков большой субъединицы рибосомы). Из Uniprot были получены белковые последовательности для каждой из бактерий.
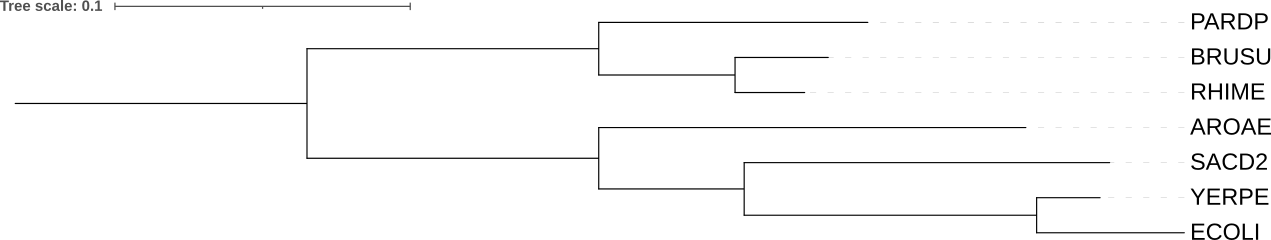
На сайте NGPhylogeny.fr была проведена реконструкция деревьем тремя алгоритмами: FastME (минимальная эволюция), TNT (максимальная экономия), PhyML (максимальное правдоподобие) (рис 2-6).
Получившиеся деревья в формате Newick: FastME, TNT, PhyML.

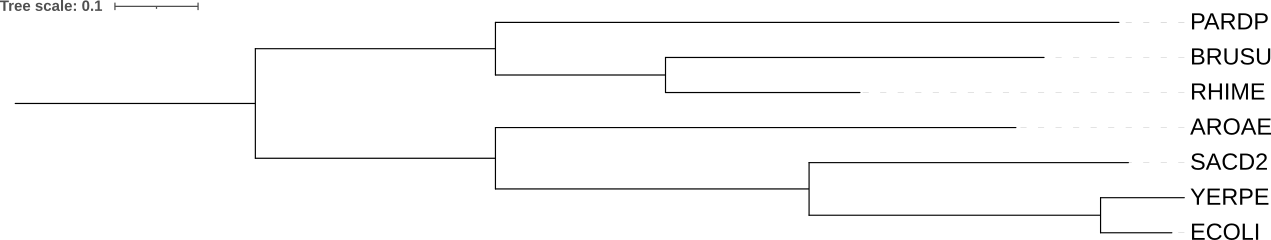
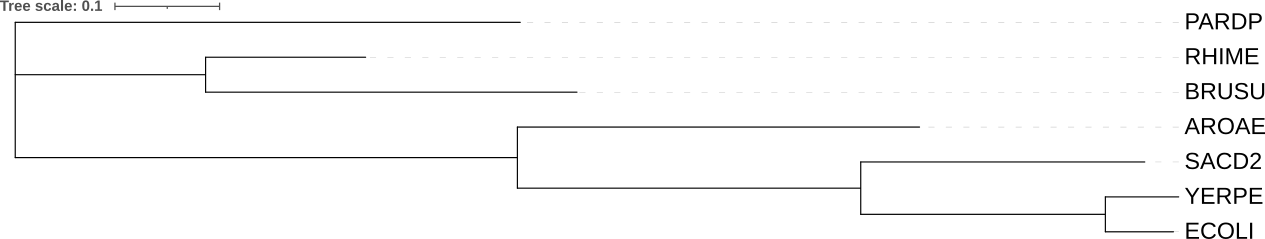
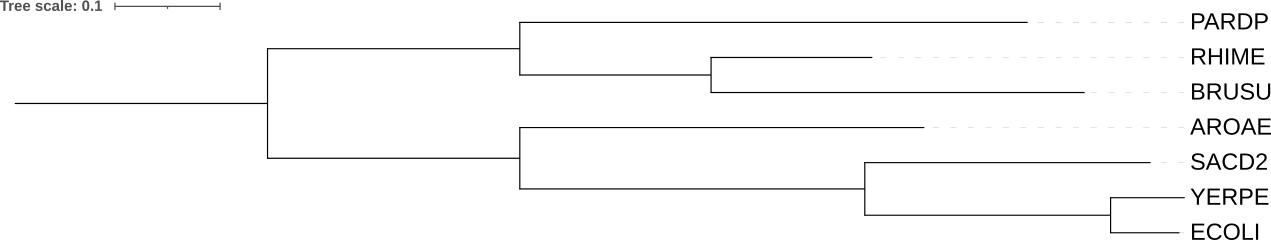
Укоренение с использованием внешней группы
Белковые последовательности RL21 для выбранных бактерий и внешней группы


Бутстреп
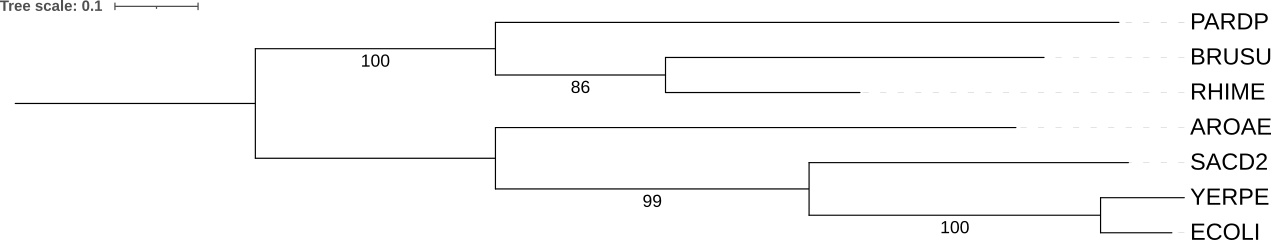
Построение дерева по нуклеотидным последовательностям
Нуклеотидные последовательности 16S рРНК для выбранных бактерий