Ортологи и паралоги
Отобранные бактерии
| Название | Мнемоника |
|---|---|
| Aromatoleum aromaticum | AROAE |
| Brucella suis | BRUSU |
| Escherichia coli | ECOLI |
| Paracoccus denitrificans | PARDP |
| Rhizobium meliloti | RHIME |
| Saccharophagus degradans | SACD2 |
| Yersinia pestis | YERPE |
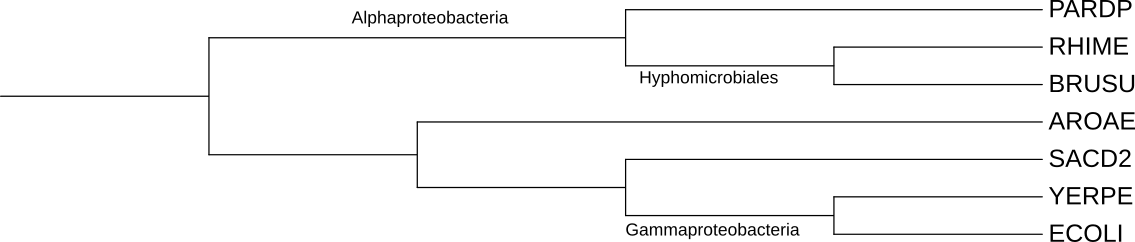
Скобочная формула дерева
((PARDP, (BRUSU,RHIME)), (AROAE, (SACD2, (ECOLI,YERPE))));
Изображение дерева
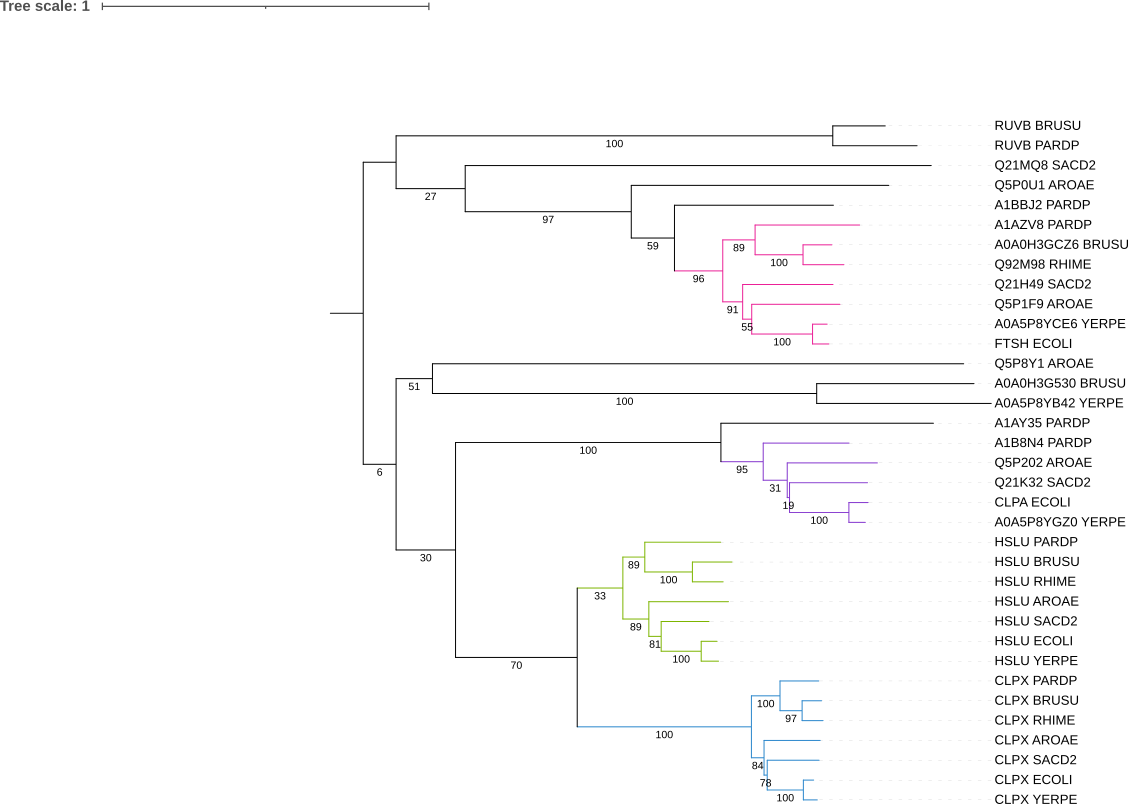
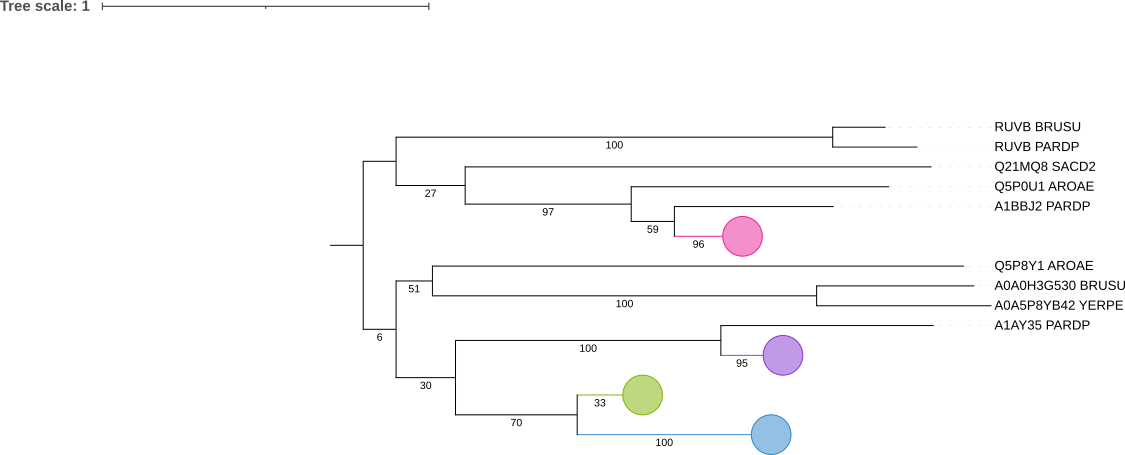
Составление списка гомологичных белков, включающих паралоги
С помощью blastp в протеомах выбранных бактерий были найдены гомологи белка CLPX_ECOLI (АТФ-связывающая субъединица протеазы Clp). Список находок, последовательности находок.
Реконструкция и визуализация
Дерево было реконструировано программой FastME с параметрами: 'Gamma distributed rates across sites' — No, 'Starting tree' — BIONJ, 'No refinement', 100 бутстреп реплик. Newick формула дерева.
Три пары ортологов: HSLU_AROAE и HSLU_SACD2, CLPX_RHIME и CLPX_ECOLI, CLPX_RHIME и CLPX_AROAE.
Три пары паралогов: CLPX_ECOLI и FTSH_ECOLI, Q21H49_SACD2 и HSLU_SACD2, Q92M98_RHIME и CLPX_RHIME.