Трансмембранные белки
Сравнение предсказаний трансмембранных участков в бета-листовом белке
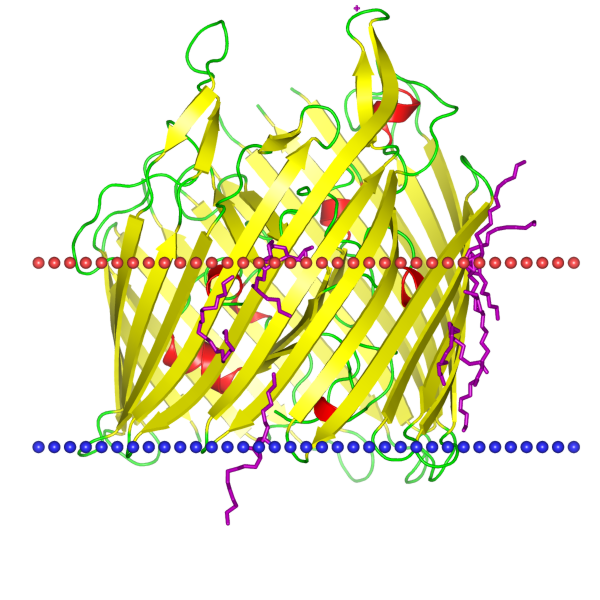
Для этого задания был выбран белок BtuB, расположенный на внешней мембране E.coli. Этот белок является транспортером витамина B12 через наружную мембрану в периплазматическое пространство.
OPM: 1nge
PDB: 1NGE
UniProt: BTUB_ECOLI
Координаты трансмембранных участков белка, приведенные в OPM:
1(136-145), 2(149-158), 3(164-173), 4(201-209), 5(214-222), 6(249-257), 7(261-269), 8(297-306), 9(309-316), 10(339-348), 11(351-359), 12(371-378), 13(383-390), 14(418-426), 15(429-436),
16(464-472), 17(475-482), 18(503-510), 19(514-521), 20(547-554), 21(559-566), 22(586-593)
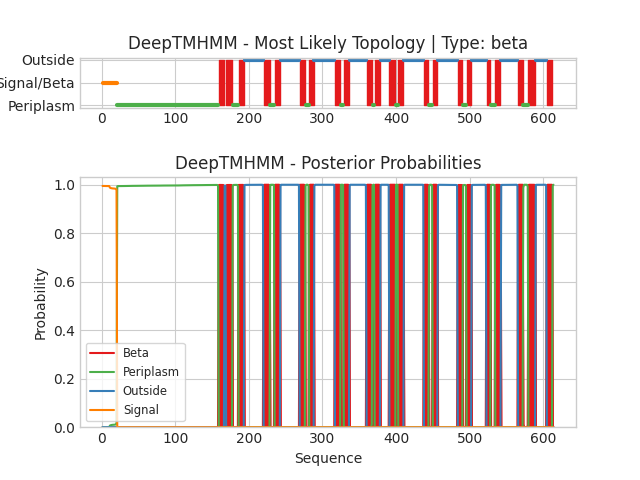
Запустим DeepTMHMM для последовательности BtuB (последовательность взята из Uniprot).
Текстовая выдача DeepTMHMM
Координаты трансмембранных участков белка, предсказанные DeepTMHMM:
1(159-166), 2(169-177), 3(186-193), 4(220-228), 5(235-242), 6(269-276), 7(282-288), 8(317-324), 9(329-336), 10(360-367), 11(371-378), 12(391-399), 13(403-410), 14(438-444), 15(450-456),
16(484-490), 17(496-502), 18(523-528), 19(535-542), 20(566-572), 21(580-589), 22(605-612)
Предсказание DeepTMHMM оказалось достаточно плохим, только один трансмембранный участок полностью совпадает (371-378) и еще четыре участка перекрываются более чем на три аминокислоты.
Сравнение предсказаний трансмембранных участков в альфа-спиральном белке
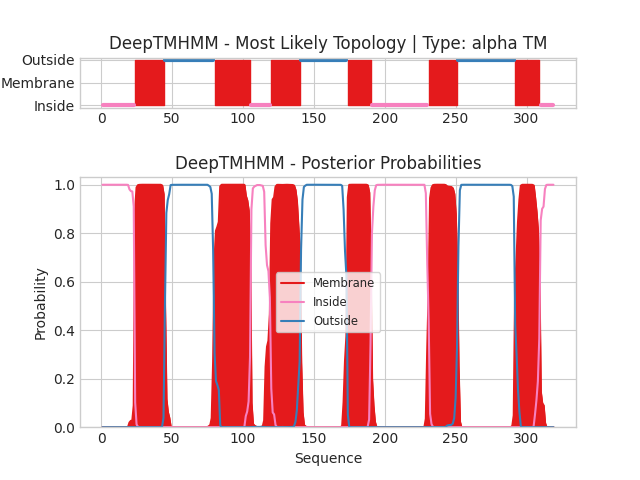
В этом задании рассмотрим белок PstC, расположенный на мембране Mycobacterium leprae. PstC — часть белково-зависимой транспортной системы фосфата. PstC, вероятно, ответственен за транслокацию субстрата через мембрану.
UniProt: PSTC_MYCLE
Запустим DeepTMHMM для последовательности PstC (последовательность взята из Uniprot).
Текстовая выдача DeepTMHMM
Координаты трансмембранных участков белка, предсказанные DeepTMHMM:
1(24-44), 2(80-105), 3(120-140), 4(174-190), 5(231-251), 6(292-309)
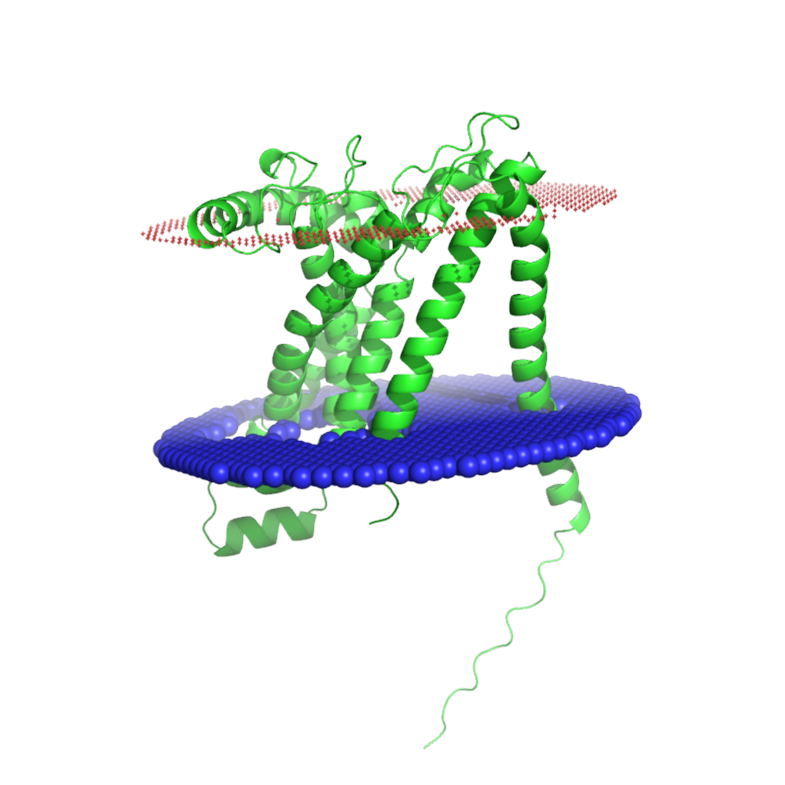
Теперь предскажем топологию белка с помощью PPM и подадим ему на вход структуру предсказанную AlphaFold, представленную на странице белка в UniProt.
Параметры: мембрана грамположительных бактерий, топология N-конца — внутри клетки. В литературе не была указана топология N-конца, поэтому было решено установить этот параметр "внутри клетки" по выдаче DeepTMHMM. Если же выбрать, что N-конец "вне клетки", то PPM все равно выдает такую же топологию (где N-конец внутри клетки), видимо, данный алгоритм в первую очередь учитывает заряд аминокислот, а не заданный нами параметр.
Топология, предсказанная PPM в формате pdb
Координаты трансмембранных участков белка, предсказанные PPM:
1(27-49), 2(77-105), 3(113-136), 4(174-194), 5(227-250), 6(291-313)
Предсказания DeepTMHMM и PPM вполне похожи, все трансмембранные участки примерно одинаковые и большей своей частью перекрываются.