Базы данных Reactome и STRING
Выданный для исследования обогащения терминами список белков подадим в базу данных Reactome:
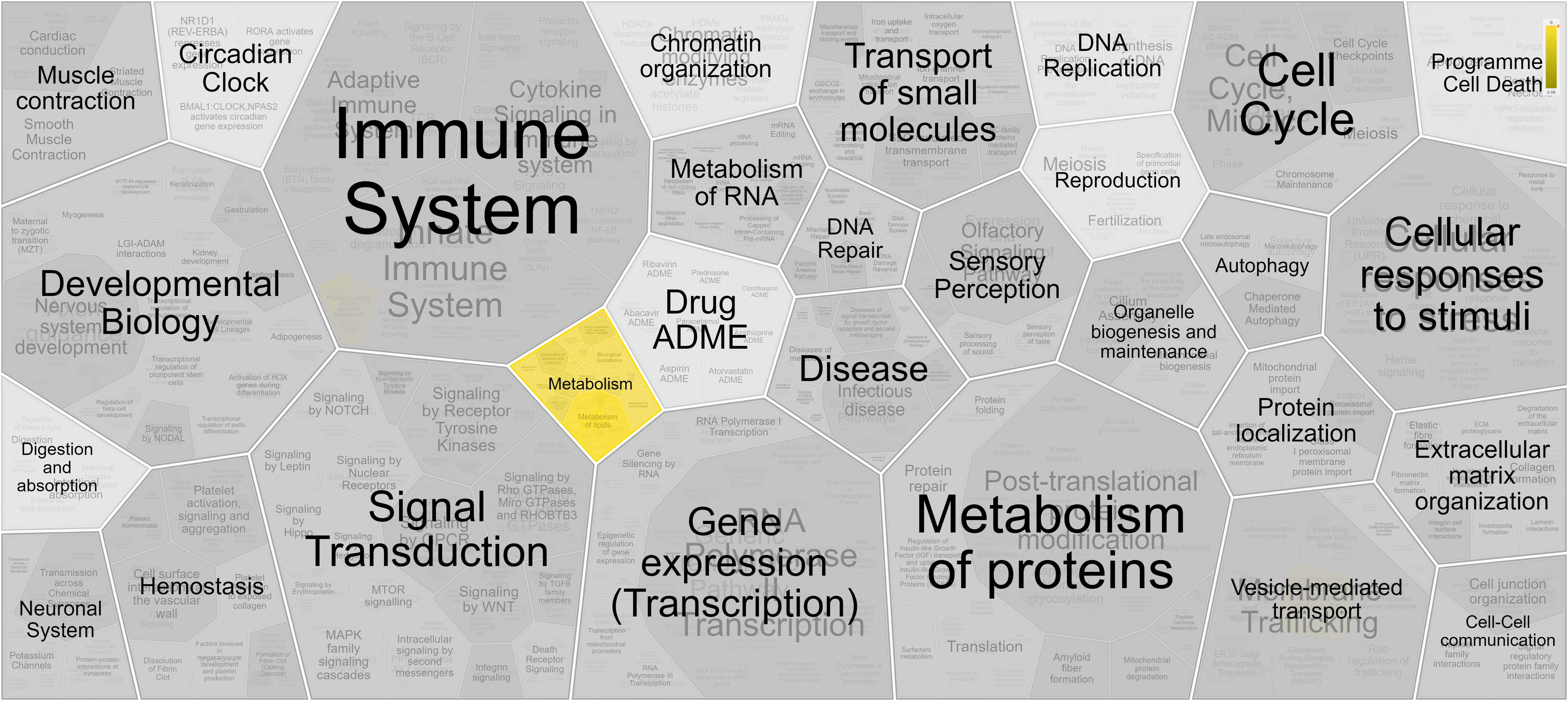
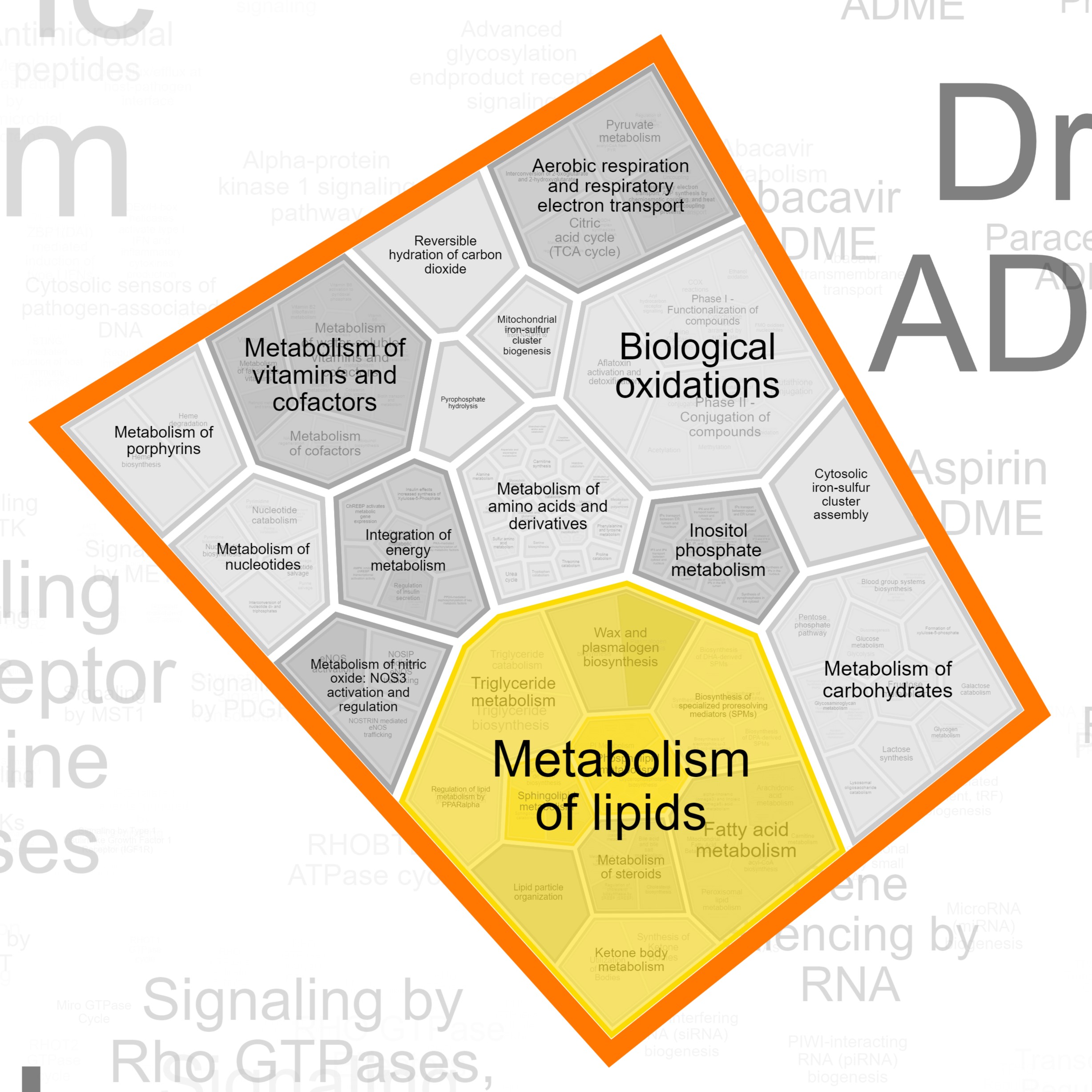
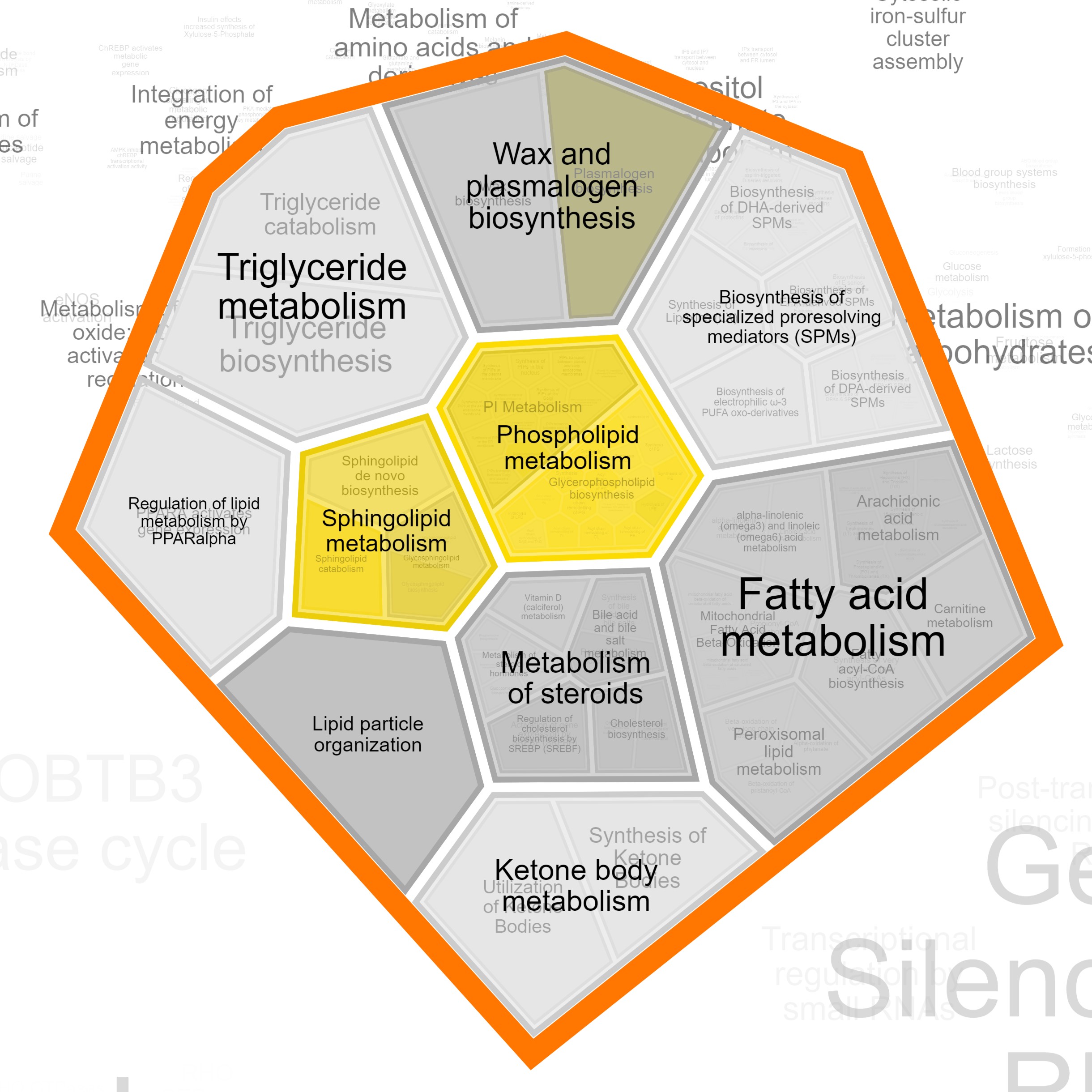

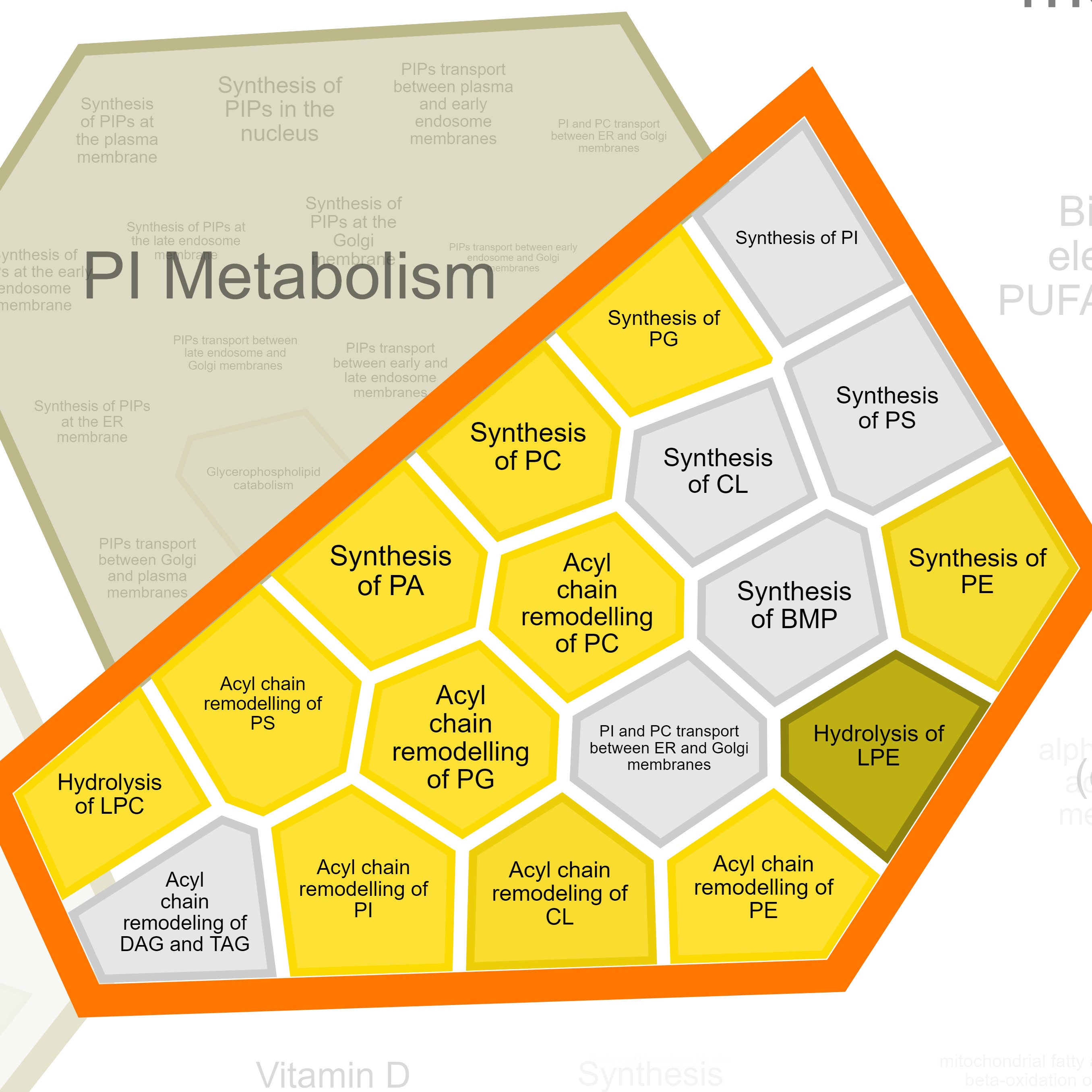
Всего в выданном списке было 49 белков, если обогащение терминами в выдаче Reactome записать схематически, включая в схему только крупные группы, то получается:
Метаболизм 41
⬑Метаболизм липидов 40
⬑Метаболизм сфинголипидов 5
⬑Метаболизм фосфолипидов 34
⬑Метаболизм PI 3
⬑Биосинтез глицерофосфолипидов 31
Теперь обратимся в базу данных STRING и проведем поиск по нашему списку белков (по таксону Homo sapiens). Получился граф с большим количеством ребер, PPI enrichment p-value меньше 1.0e-16. Это говорит о том, что эти белки имеют больше взаимодействий между собой, чем можно было бы ожидать для случайного набора белков такого же размера. То есть у нас точно есть обогащение!
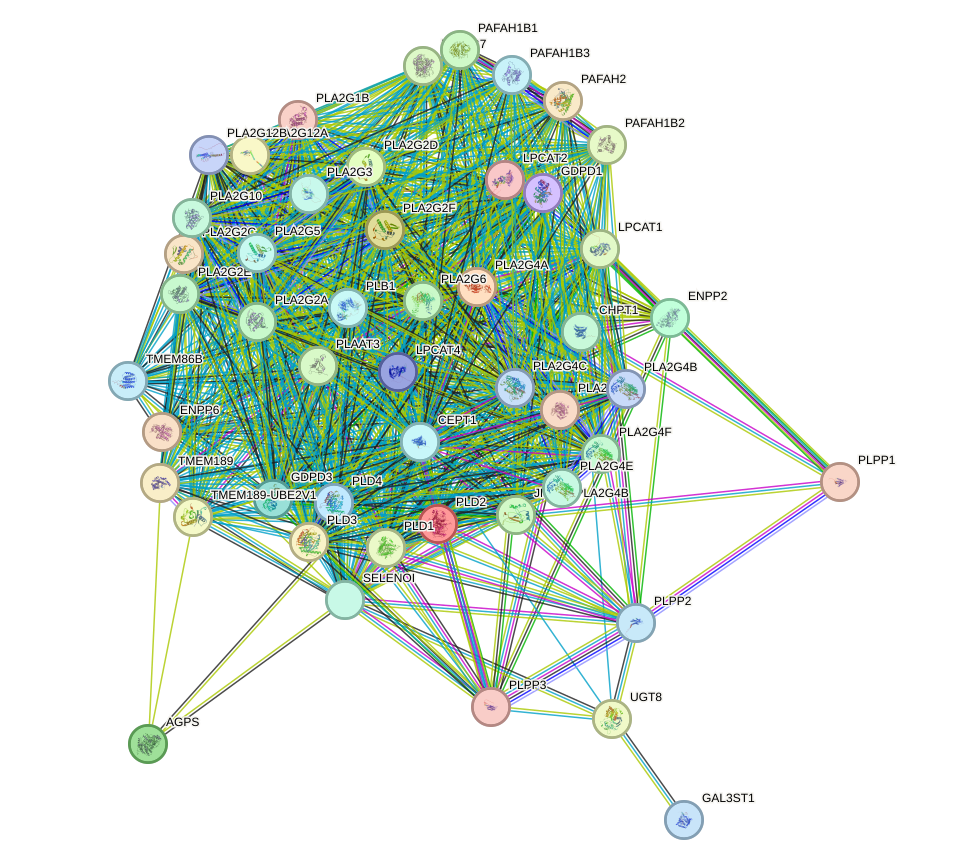
Так же есть возможность покрасить узлы нашего графа в зависимости от участия белков в каком-либо биологическом процессе согласно определенной базе данных. Посмотрим расположение в графе белков, участвующих в метаболизме глицерофосфолипидов по данным Gene Ontology. Эти белки, действительно, располагаются очень близко друг к другу (хотя у нас все тут близко друг у другу). Частота ложных обнаружений (False Discovery Rate) этой группы равно 2.47e-44, что подверждает значимость обогащения этим термином.
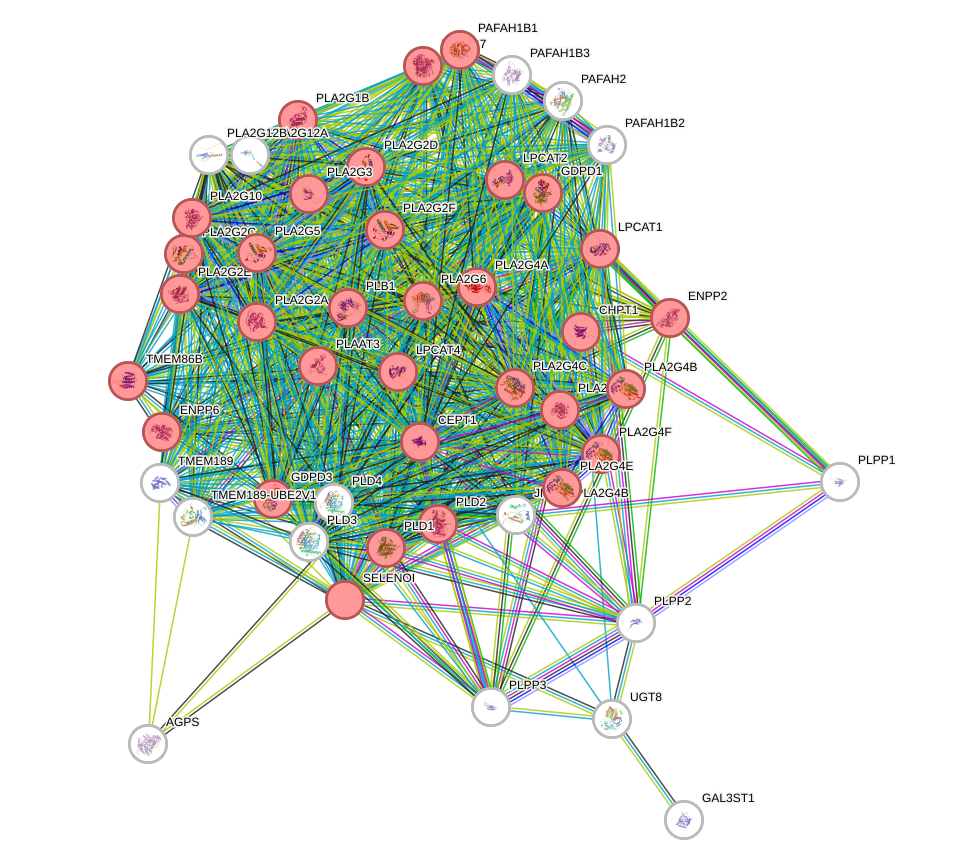
Вывод: наш список белков обогащен белками, участвующими в метаболизме липидов, в особенности метаболизме глицерофосфолипидов.