Поиск de novo сигналов в ДНК
Подготовка данных
В этом практикуме снова возьмем Thermus thermophilus HB8.
Ссылка на аннотированную геномную сборку
С помощью сервиса Operon-mapper на основе fasta-файла последовательности генома и таблицы особенностей в формате .gff были определены координаты оперонов.
Затем по списку оперонов были получены промоторные области (участки 100 пн перед оперонами), из них вручную были выбраны 24 промоторных областей генов домашнего хозяйства. Тестовая выборка — все промоторные области, негативная выборка — участки от +900 до +1000 от старт-кодона первого гена в опероне.
Cкрипт, с помощью которого были получены выборки
Запуск MEME
Далее не сервисе MEME-suit был запущен MEME для отобранных последовательностей генов домашнего хозяйства со следующими параметрами:
Maximum Number of Motifs 3
Site Strand Handling Sites must be on the given strand
Minimum Motif Width 5
Maximum Motif Width 50


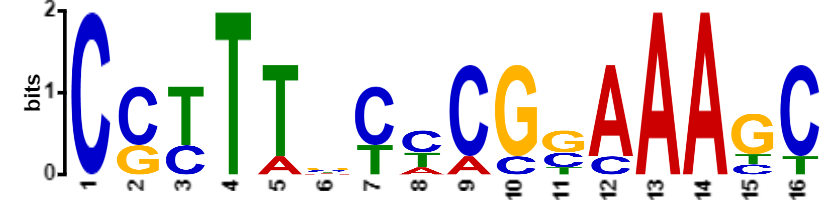
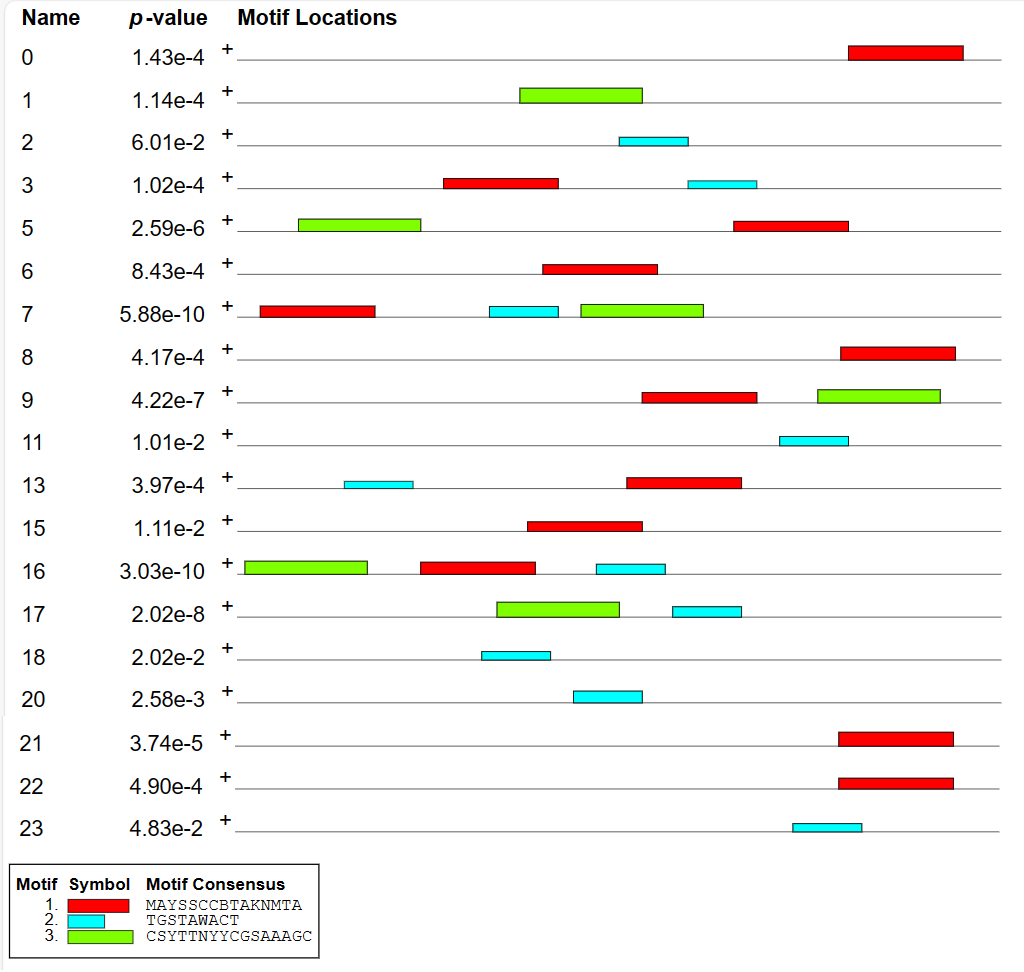
Было найдено три мотива, для дальнейшей работы был взят первый мотив, так как у него наименьший E-value. (Хочется верить что это мотив -10, так как он расположен в этом месте у 4-х промоторов)
Поиск сигнала в материале для тестирования с помощью FIMO
Далее была запущена программа FIMO для первого мотива для тестовой и негативной выборки:
fimo --norc -motif MAYSSCCBTAKNMTA -thresh 0.001 ./meme.txt ./promoters_test100.txt
fimo --norc -motif MAYSSCCBTAKNMTA -thresh 0.001 ./meme.txt ./negative.txt
Мотив был найден в 353 промоторах и в 130 последовательностях внутри генов (негативгая выборка), при размере выборок 759. Вполне неплохой результат, выборки различаются в количестве сигнала(+) почти в три раза.
| Тестовая выборка | Негативная выборка | |
|---|---|---|
| Сигнал(+) | 353 | 130 |
| Сигнал(-) | 406 | 629 |