Чтения были скачены, с помощью данной команды:
wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/006/SRR4240356/SRR4240356.fastq.gz
Далее собираем адаптеры в один файл:
cat /mnt/scratch/NGS/adapters/* >> adapters.fasta
Теперь удаляем возможные остатки адаптеров:
java -jar /usr/share/java/trimmomatic.jar SE -phred33 -threads 10 SRR4240356.fastq.gz trimmed.fastq.gz ILLUMINACLIP:adapters.fasta:2:7:7 2>trim.log
2.04% (153091) последовательностей чтений оказалось остатками адаптеров.
Далее удаляем c правых концов чтений нуклеотиды с качеством ниже 20 и длиной больше 32 нуклеотиддов.
java -jar /usr/share/java/trimmomatic.jar SE -phred33 -threads 10 trimmed.fastq.gz trimmed2.fastq.gz TRAILING:20 MINLEN:32 2>trim2.log
Было удалено 4.15% (305092) чтений. Самый исходный файл весил 174.26 мегабайт, а полученный 162.02 мегабайт.
Чтобы получить k-меры длины 31 выполним следующую команду:
velveth assemble 31 -fastq.gz -short trimmed2.fastq.gz 2> velveth.log
В созданной директории 'assemble' мы получили три файла: Log, Roadmaps, Sequences.
Запустим программу velvetg:
velvetg assemble &> velvetg.log
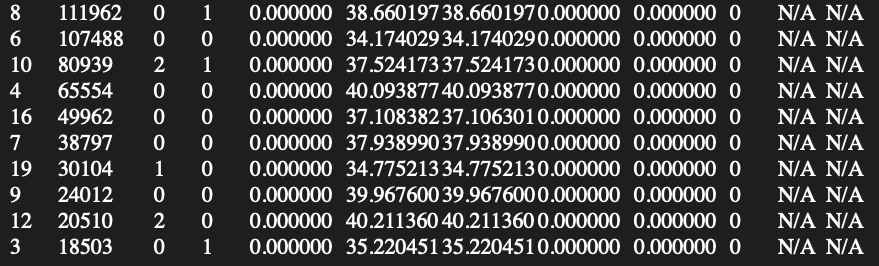
Из выхода, можем узнать, что N50 = 65554
Далее узнаем длину самых длинных контигов (команду запускаем непосредственно в директории 'assemble'):
sort -n -k 2 -r stats.txt | head

Контиги с аномально большим или аномально малым покрытием (команды запускаем непосредственно в директории 'assemble'):
sort -n -k 6 -r stats.txt | head
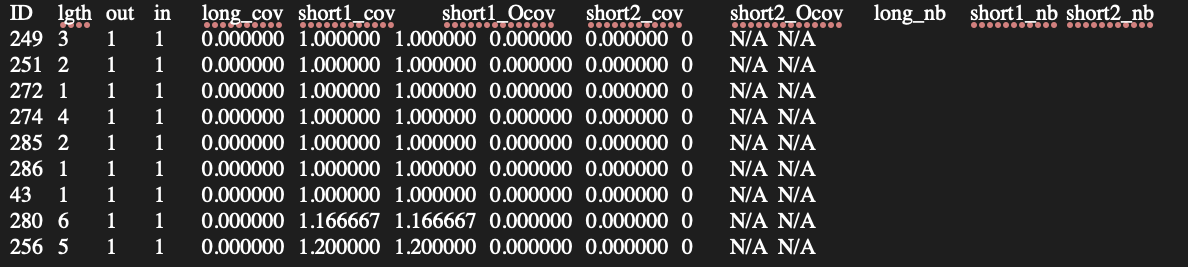
sort -n -k 6 stats.txt | head Аномально большое покрытие встречается у контиг с ID64 (покрытие - 266951.0). Несколько контигов с покрытием 1.0 представлены ниже. (К примеру ID249, ID251, ID272)
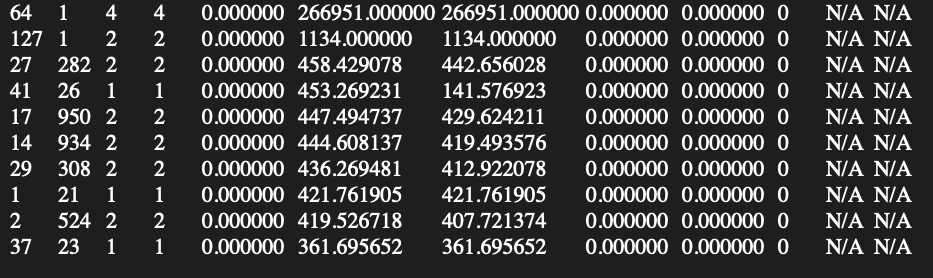
Выравним самые длинные контиги - ID: 6, 8, 10 с геномом Buchnera aphidicola (GenBank/EMBL AC — CP009253) с помощью megablast. Анализ каждого выравнивания можно посмотреть ниже.
Контиг 8.Координаты на хромосоме 451729 - 555905. Покрытие - 75%. Per. Identity - 81.46%. Суммарный вес выравнивания - 50932. Имеется 5 разрывов, самый крупный примерно 10000 нуклеодитов. Карту локального сходства можно посмотреть на Рисунке 1. Выдача Blast
Контиг 6.Координаты на хромосоме 220869 - 323043. Покрытие - 74%. Per. Identity - 78.76%. Суммарный вес выравнивания - 43281. Имеется 9 разрывов, самый крупный примерно 7000 нуклеодитов. Карту локального сходства можно посмотреть на Рисунке 2. Выдача Blast
Контиг 10.Координаты на хромосоме 528794 - 555905. Покрытие - 65%. Per. Identity - 74.88%. Суммарный вес выравнивания - 30801. Имеется 5 разрывов, самый крупный примерно 7000 нуклеодитов. Карту локального сходства можно посмотреть на Рисунке 3. Выдача Blast