Последовательность Шайна — Дальгарно. Это сайт связывания рибосом на молекуле мРНК. Встречается у прокариот. Находится на расстоянии ≈ 10 нуклеотидов от стартового кодона AUG. Консенсус - AGGAGG. Если в последовательности Шайна — Дальгарно появятся мутации, то они снизят эффективность трансляции. Этот сигнал высокоэффективен, тк обеспечивает связывание рибосом в молекуле мРНК. Так же является консервативной.
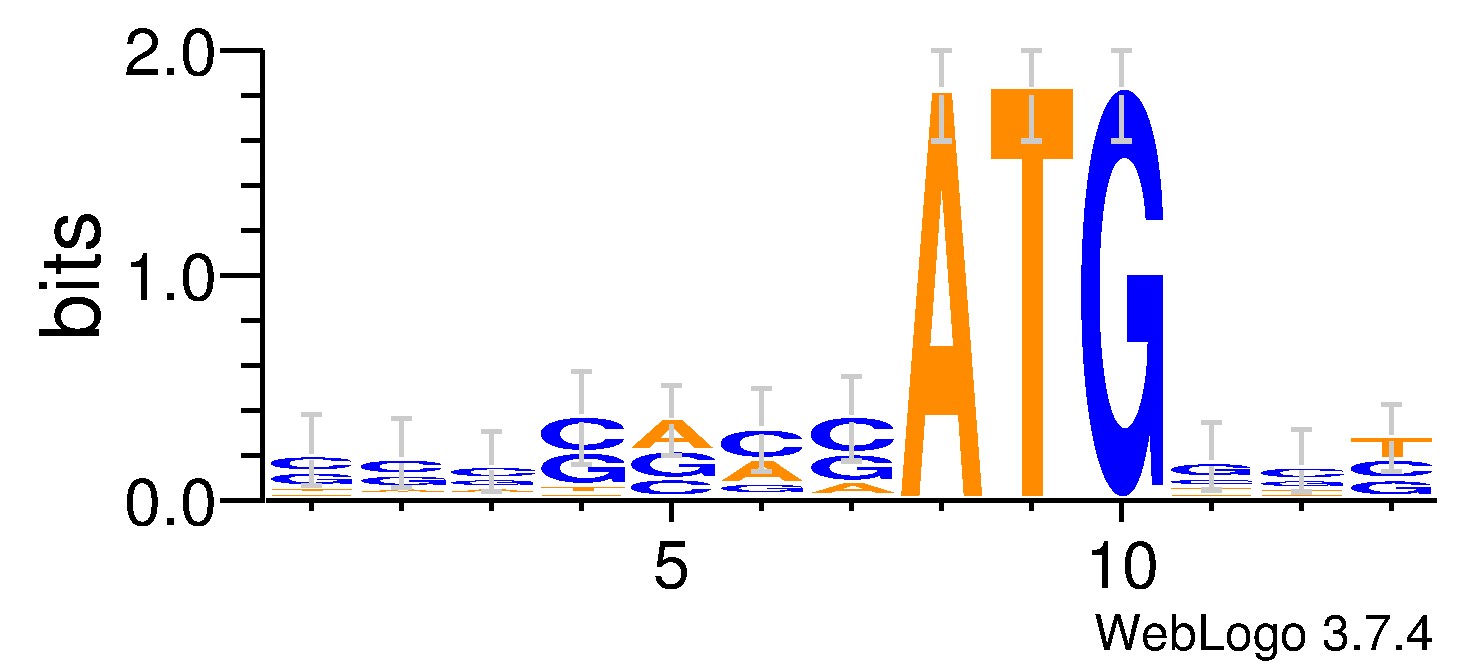
В этом задании нужно было исследовать последовательность Козак и сделать выводы о ее специфичности. Чтобы это сделать я воспользовалась скриптом Максима Смирнова. Этот скрипт выбирает 100 случайных генов человека. В них вырезается фрагмент 7 bp до + стартовый ATG + 3 bp после. Выдачи скрипта: n=40 n = 60. Далее по выравниваниям (без гэпов) из нашей выборки была посмтроена PWM.
| letter | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 |
| A | -2.291 | -0.369 | -0.649 | 0.107 | -0.824 | -0.499 | -0.824 | 1.221 | -4.083 | -4.083 | -0.369 | -0.499 | -1.31 |
| T | 0.372 | -0.499 | -1.31 | -0.369 | -1.31 | -0.824 | -1.685 | -4.083 | 1.221 | -4.083 | -1.038 | -1.038 | -0.824 |
| G | 0.307 | 0.307 | 0.736 | 0.11 | 0.846 | 0.544 | 0.11 | -3.719 | -3.719 | 1.585 | 0.991 | 0.392 | 0.945 |
| C | 0.307 | 0.544 | 0.544 | 0.213 | 0.471 | 0.544 | 1.077 | -3.719 | -3.719 | -3.719 | -0.285 | 0.736 | 0.307 |
Положительный контроль - последовательности из тестового файла,отрицательный контроль - 60 последовательностей, которые не содержат стартовый ATG (+)-контроль.
✔ positive control mean score: 5.242721611434907
✔ negative control mean score: 1.0094930169853094
✔ p-value: 3.645543287263915e-13
✔ ic:9.784051189548972
P-value имеет очень маленькое значение. negative control mean score меньше чем positive control mean score в 5 раз. Из этого можно сделапть вывод, что у нас разные последовательности, и что Козак специфичен для стартового кодона. Эти данные так же посчитаны с помощью скрипта.
Была построена матрица информационного содержания:
| letter | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 |
| A | -0.099 | -0.108 | -0.144 | 0.053 | -0.154 | -0.128 | -0.154 | 1.777 | 0 | 0 | -0.108 | -0.128 | -0.151 |
| T | 0.234 | -0.128 | -0.151 | -0.108 | -0.151 | -0.154 | -0.133 | 0 | 1.777 | 0 | -0.156 | -0.156 | -0.154 |
| G | 0.126 | 0.126 | 0.46 | 0.038 | 0.589 | 0.281 | 0.038 | 0 | 0 | 2.305 | 0.797 | 0.175 | 0.726 |
| C | 0.126 | 0.281 | 0.281 | 0.08 | 0.227 | 0.281 | 0.945 | 0 | 0 | 0 | -0.063 | 0.46 | 0.126 |
Далее с помощью программы WebLOGO3 было визуализировано ИС последовательности. Из порлученного результата, можно сделать вывод, что 7 нуклеотидов до ATG и 3 нуклеотида после имеет значимый информационный вес.
