В базе данных Pfam я выбрала домен Arc_C (PF18162).
Белки:
✏ 1 - seed
✏ 157 - full
✏ 409 - UniProt
Average length of the domain - 82.9
Average identity of full alignment - 86 %
Average coverage of the sequence by the domain - 20.89 %
Model length - 83
Для этого домена была выбрана двухдоменная архитектура, состоящая из Arc_MA и Arc_C. Белков с данной архитектурой - 155.

Далее были созданы следующие файлы:
✏ файл с последовательностями всех белков данной архитектуры
✏ файл со списком АС белков данной архитектуры
✏ файл со списком АС белков для анализа
Далее с помощью Jalview было сделано выравнивание с помощью алгоритма muscle. В этом варавнивании было удалено 74 последовательности из 155 основываясь на Remove redundancy = 99%. Так же были удалены концы последовательностей, которые не входили в состав доменов. Итоговое выравнивание
Далее с помощью перечисленных ниже команд сделали HMM-профиль.
✏ hmm2build profile aligment.fa
✏ hmm2calibrate profile
✏ hmmsearch --cpu=1 profile PF18162_full_length_sequences.fasta > res_pr9.txt
С помощью выше перечисленных команд были созданы следующие файлы:
✏ HMM-профиль с калибровкой
✏ результаты HMMsearch
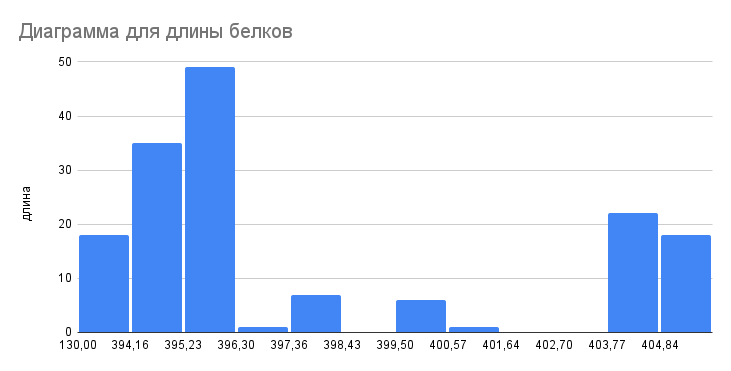
Основываясь на полученных данных была сделена таблица в Excel. Из полученной ниже гистограммы распределения длин белков видно, что для выбранного домена характера длина в интервале 395 - 396.