Мембранные белки
На главную страницу четвертого семестра
Задача — предсказать топологию мембранного белка и сравнить предсказание
с ориентированной в мембране 3D-структурой белка-прототипа.
Идентификаторы:
белок-прототип AC UniProt P02722
PDB ID 1OKC
белок для исследования Q8AYM3_CHICK: AC UniProt Q8AYM3
Построение парного выравнивания исследуемого белка и заданного прототипа
Т.к. последовательности и нумерация остатков в БД PDB и БД UniProt могут
различаться, поэтому необходимо было получить и сравнить обе последовательности
белка-прототипа.
В БД UniProt была получена последовательность белка-прототипа (запрос:
[uniprot-AccNumber:P02722*]).
На главной страничке сайта PDB в поле
запроса был введен ID белка-прототипа. На появившейся страниче, с помощью опции
Download Files > FASTA Sequence, была сохранена последовательность белка-прототипа.
С помощью программы needle было сделано выравнивание данных последовательностей:
needle prot1.fasta prot2.fasta aln.needle -auto
ВЫРАВНИВАНИЕ:
########################################
# Program: needle
# Rundate: Tue Apr 10 2007 13:07:04
# Commandline: needle
# [-asequence] prot1.fasta
# [-bsequence] prot2.fasta
# [-outfile] aln.needle
# -auto
# Align_format: srspair
# Report_file: aln.needle
########################################
#=======================================
#
# Aligned_sequences: 2
# 1: ADT1_BOVIN
# 2: SEQUENCE
# Matrix: EBLOSUM62
# Gap_penalty: 10.0
# Extend_penalty: 0.5
#
# Length: 298
# Identity: 297/298 (99.7%)
# Similarity: 297/298 (99.7%)
# Gaps: 1/298 ( 0.3%)
# Score: 1542.0
#
#
#=======================================
ADT1_BOVIN 1 MSDQALSFLKDFLAGGVAAAISKTAVAPIERVKLLLQVQHASKQISAEKQ 50
|||||||||||||||||||||||||||||||||||||||||||||||||
SEQUENCE 1 -SDQALSFLKDFLAGGVAAAISKTAVAPIERVKLLLQVQHASKQISAEKQ 49
ADT1_BOVIN 51 YKGIIDCVVRIPKEQGFLSFWRGNLANVIRYFPTQALNFAFKDKYKQIFL 100
||||||||||||||||||||||||||||||||||||||||||||||||||
SEQUENCE 50 YKGIIDCVVRIPKEQGFLSFWRGNLANVIRYFPTQALNFAFKDKYKQIFL 99
ADT1_BOVIN 101 GGVDRHKQFWRYFAGNLASGGAAGATSLCFVYPLDFARTRLAADVGKGAA 150
||||||||||||||||||||||||||||||||||||||||||||||||||
SEQUENCE 100 GGVDRHKQFWRYFAGNLASGGAAGATSLCFVYPLDFARTRLAADVGKGAA 149
ADT1_BOVIN 151 QREFTGLGNCITKIFKSDGLRGLYQGFNVSVQGIIIYRAAYFGVYDTAKG 200
||||||||||||||||||||||||||||||||||||||||||||||||||
SEQUENCE 150 QREFTGLGNCITKIFKSDGLRGLYQGFNVSVQGIIIYRAAYFGVYDTAKG 199
ADT1_BOVIN 201 MLPDPKNVHIIVSWMIAQTVTAVAGLVSYPFDTVRRRMMMQSGRKGADIM 250
||||||||||||||||||||||||||||||||||||||||||||||||||
SEQUENCE 200 MLPDPKNVHIIVSWMIAQTVTAVAGLVSYPFDTVRRRMMMQSGRKGADIM 249
ADT1_BOVIN 251 YTGTVDCWRKIAKDEGPKAFFKGAWSNVLRGMGGAFVLVLYDEIKKFV 298
||||||||||||||||||||||||||||||||||||||||||||||||
SEQUENCE 250 YTGTVDCWRKIAKDEGPKAFFKGAWSNVLRGMGGAFVLVLYDEIKKFV 297
#---------------------------------------
#---------------------------------------
|
|
Вывод:
Последовательноть белка-прототипа из БД UniProt и последовательность того же белка, но
из БД PDB практически идентичны (Identity 99.7%), отличаются лишь первым элементом. Нумерация
отличается на единицу и идет со второго атома.
Далее идентификатору UniProt была получена последовательность заданного белка (Q8AYM3_CHICK).
Запрос:[uniprot-AccNumber:Q8AYM3*]. С помощью программы needle было
построено выравнивание последовательностей заданного белка (Q8AYM3_CHICK) и
белка-прототипа (последовательность из БД PDB).
needle serch.fasta prot2.fasta aln1.needle -aformat3 msf
ВЫРАВНИВАНИЕ:
########################################
# Program: needle
# Rundate: Tue Apr 10 2007 13:21:47
# Commandline: needle
# [-asequence] serch.fasta
# [-bsequence] prot2.fasta
# [-outfile] aln2.needle
# -auto
# Align_format: srspair
# Report_file: aln2.needle
########################################
#=======================================
#
# Aligned_sequences: 2
# 1: Q8AYM3_CHICK|
# 2: SEQUENCE
# Matrix: EBLOSUM62
# Gap_penalty: 10.0
# Extend_penalty: 0.5
#
# Length: 298
# Identity: 267/298 (89.6%)
# Similarity: 285/298 (95.6%)
# Gaps: 1/298 ( 0.3%)
# Score: 1425.0
#
#
#=======================================
Q8AYM3_CHICK| 1 MADQAISFLKDFLARGVAAAISKTAVAPIERVKLLLQVQHASKQIAADKQ 50
:|||:||||||||.||||||||||||||||||||||||||||||:|:||
SEQUENCE 1 -SDQALSFLKDFLAGGVAAAISKTAVAPIERVKLLLQVQHASKQISAEKQ 49
Q8AYM3_CHICK| 51 YKGIIDCVVRIPKEQGVLSFWRGNLANVIRYFPTQALNFAFKDKYKQVFL 100
||||||||||||||||.||||||||||||||||||||||||||||||:||
SEQUENCE 50 YKGIIDCVVRIPKEQGFLSFWRGNLANVIRYFPTQALNFAFKDKYKQIFL 99
Q8AYM3_CHICK| 101 GGVDKHTQFWRYFAGNLASGGAAGATSLCFVYPLDFARTRLAADVGKAGA 150
||||:|.||||||||||||||||||||||||||||||||||||||||..|
SEQUENCE 100 GGVDRHKQFWRYFAGNLASGGAAGATSLCFVYPLDFARTRLAADVGKGAA 149
Q8AYM3_CHICK| 151 DREFSGLGDCLVKITKSDGLRGLYQGFNVSVQGIIIYRAAYFGIYDTAKG 200
.|||:|||:|:.||.||||||||||||||||||||||||||||:||||||
SEQUENCE 150 QREFTGLGNCITKIFKSDGLRGLYQGFNVSVQGIIIYRAAYFGVYDTAKG 199
Q8AYM3_CHICK| 201 MLPDPRNTHIVISWMIAQTVTAVAGVVSYPFDTVRRRMMMQSGRKGADIM 250
|||||:|.||::|||||||||||||:||||||||||||||||||||||||
SEQUENCE 200 MLPDPKNVHIIVSWMIAQTVTAVAGLVSYPFDTVRRRMMMQSGRKGADIM 249
Q8AYM3_CHICK| 251 YSGTIDCWRKIARDEGGKAFFKGAWSNVLRGMGGAFVLVLYDEFKKVI 298
|:||:|||||||:|||.||||||||||||||||||||||||||.||.:
SEQUENCE 250 YTGTVDCWRKIAKDEGPKAFFKGAWSNVLRGMGGAFVLVLYDEIKKFV 297
#---------------------------------------
#---------------------------------------
|
|
Далее это выравнивание было открыто программой GeneDoc и сохранено в файле
marking.msf
Идентичность - 89.6%.
Разметка мембранных сегментов на выравнивании
По идентификатору PDB белка-прототипа нашел описание ориентации белка в мембране в БД
OPM (Orientations of Proteins in Membranes database).
Получилось, что в сторону межмембранного пространства 2-10, 92-111, 198-209;
в сторону матрикса расположены сегменты 34-72, 139-171, 236-268, 293-298;
внутри мембраны расположены сегменты 11-33, 73-91, 112-138, 172-197, 210-235, 269-292.
В файле marking.msf ниже последовательности прототипа добавил последовательность
с названием "OPM". Позиции мембранных сегментов отмечены буквой "Н", позиции цитоплазматических петель знаком "+",
остальные - знаком "-".
Получил новый файл.
Предсказание топологии заданного белка с помощью наиболее популярной программы (TMHMM)
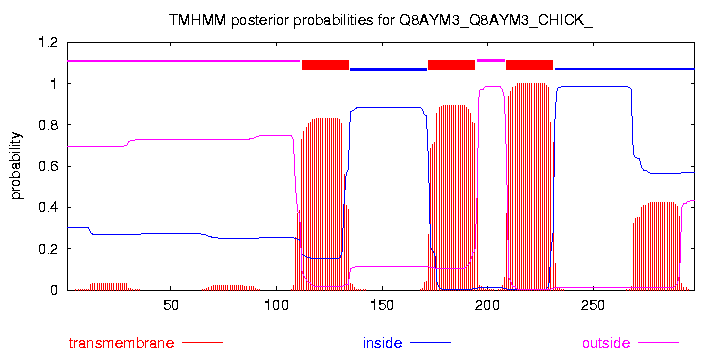
Результат предсказания топологии заданного белка Q8AYM3:
в сторону межмембранного пространства распологаются сегменты 1-111, 195-208;
в сторону матрикса 135-171, 232-298;
внутри мембраны 112-134, 172-194, 209-231.
# Q8AYM3_Q8AYM3_CHICK_ Length: 298
# Q8AYM3_Q8AYM3_CHICK_ Number of predicted TMHs: 3
# Q8AYM3_Q8AYM3_CHICK_ Exp number of AAs in TMHs: 70.89884
# Q8AYM3_Q8AYM3_CHICK_ Exp number, first 60 AAs: 0.61985
# Q8AYM3_Q8AYM3_CHICK_ Total prob of N-in: 0.30300
Q8AYM3_Q8AYM3_CHICK_ TMHMM2.0 outside 1 111
Q8AYM3_Q8AYM3_CHICK_ TMHMM2.0 TMhelix 112 134
Q8AYM3_Q8AYM3_CHICK_ TMHMM2.0 inside 135 171
Q8AYM3_Q8AYM3_CHICK_ TMHMM2.0 TMhelix 172 194
Q8AYM3_Q8AYM3_CHICK_ TMHMM2.0 outside 195 208
Q8AYM3_Q8AYM3_CHICK_ TMHMM2.0 TMhelix 209 231
Q8AYM3_Q8AYM3_CHICK_ TMHMM2.0 inside 232 298
Выравнивание с искусственной последовательностью, отражающей результаты данного предсказания.
Эту последовательность назвали "TMHMM", а так же в текстовом формате "TMHMM".
Оценка качества предсказания
В этом задании необходимо сравнить полученное предсказание с данными OPM.
Для этого нужно было подсчитать 4 числа TP,TN,FP,FN, описание которых дано в
таблице
Результаты предсказания топологии мембранного белка Q8AYM3_CHICK
| |
Число а.к. остатков |
| Всего а.к. остатков |
298 |
| Остатки, предсказанные как локализованные в мембране (всего) |
69 |
| Правильно предсказали (true positives, TP) |
65 |
| Предсказали не то, что нужно (а.о. предсказаны как мембранные, а по данным ОРМ таковыми не являются, false positives, FP) |
4 |
| Правильно не предсказали ( не предсказаны, и по данным ОРМ не находятся в мембране, true negatives, TN) |
149 |
| Не предсказали то, что нужно (остатки по данным ОРМ находятся в мембране, false negatives, FN) |
80 |
| Чувствительность (sensivity) = TP / (TP+FN) |
0.448 |
| Специфичность (specificity) =TN / (TN+FP) |
0.974 |
| Точность (precision) = TP / (TP+FP) |
0.942 |
| Сверхпредсказание = FP/ (FP+TP) |
0.058 |
| Недопредсказание = FN / (TN+FN) |
0.349 |
Вывод:
Были получены следующие данные:
Чувствительность: отношение правильно предсказанных к реальному значению( TP / (TP+FN))
- способность программы отыскивать нужное - в данном случае невысокая(0.448)
Специфичность: по отношению TN/(TN+FP) можно судить о способности программы не брать лишние остатки, в данном
случае равное 0.974
Недопредсказание = FN / (TN+FN),где TN+FN=остатки, предсказанные как немембранные, - доля неправильно предсказанных
немембранных участков (0.349).
На основе данных таблицы можно сделать вывод, что сервер TMHMM сделал не очень качественное
предсказание для белка Q8AYM3_CHICK, так как значение чувствительности достаточно низкое, а недопредсказания достаточно
высокое.Общее число ошибок (80+4=84 - неправильно предсказанных позиций) составляет почти 28,19% от общего
числа остатков исследованной последовательности (298 а.о.), что достаточно много.
©Долудин Юрий
