Для поиска гомологов белка TENA_BACSU был задействован алгоритм BLASTP по базе данных RefSeq, результатом работы которого оказались белки с наиболее совпадающими аминокислотными последовательностями. Гомологами считались белки с E-value < 1e-5. Дополнительные параметры поиска представлены в таблице 1.
| Таблица 1 Параметры поиска BLAST | ||||||||||||||||||||
| Поиск | Алгоритм BLAST | Название базы данных | Ограничения по таксонам | Порог e-value | Максимальное количество хитов | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| По прокариотам | BlastP | Reference proteins | exclude Firmicutes и exclude Eukaryota |
0.00001 | 1000 | |||||||||||||||
| По эукариотам | BlastP | Reference proteins | только Eukaryota | 0.00001 | 250 | |||||||||||||||
Поиск по прокариотам
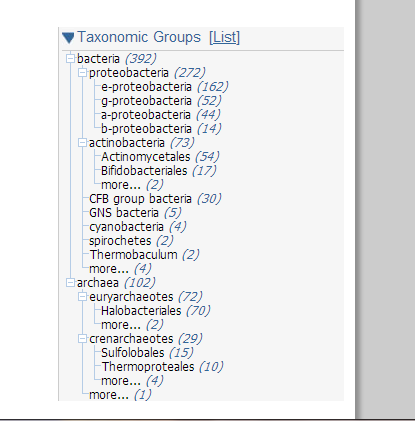
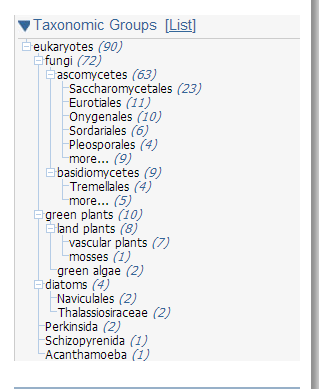
Для поиска по прокариотам из всех живых организмов были исключены Eukaryota и Firmicutes ( исключение Firmicutes связано с тем, что Bacillus subtilis относится к этому филуму и поэтому, вероятнее всего, что для полученных хитов идентичность будет настолько высокой с TENA_BACSU, что выравнивание будет слабо информативным ). Также было получено 1000 хитов алгоритма BLAST, из которых по первым 500 хитам была получена сводка GenPept базы данных GenBank (рис.1) После этого вручную случайным образом было выбрано 20 белков прокариот из разных филумов. Находки были добавлены в выборку, отображенную в таблице 2.

Для этого поиска BLAST проводился только по доминиону Eukaryota, с ограничением в 250 хитов. Обнаружилось 90 находок для которых была получена сводка GenPept базы данных GenBank (рис.2) После этого также вручную случайным образом было выбрано 20 белков эукариот из разных филумов. Отчет по выборке также представлен в таблице 2.
| Таблица 2 Встречаемость белков в различных таксонах | ||||||||||||||||||||
| Домен | Филум/Царство | Название организма | Количество белков | |||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Archaea | Euryarchaeotes | Natronobacterium gregoryi SP2 | 2 | |||||||||||||||||
| Natrialba aegyptia DSM 13077 | ||||||||||||||||||||
| Crenarchaeota | Sulfolobus islandicus LAL14/1 | 2 | ||||||||||||||||||
| Pyrobaculum neutrophilum V24Sta | ||||||||||||||||||||
| Bacteria | Actinomycetales | Microlunatus phosphovorus NM-1 | 2 | |||||||||||||||||
| Mycobacterium fortuitum subsp. fortuitum DSM 46621 | ||||||||||||||||||||
| Bifidobacteriales | Scardovia inopinata F0304 | 2 | ||||||||||||||||||
| Bifidobacterium animalis subsp. lactis CNCM I-2494 | ||||||||||||||||||||
| CFB group bacteria | Zunongwangia profunda SM-A87 | 2 | ||||||||||||||||||
| Bacteroides coprosuis DSM 18011 | ||||||||||||||||||||
| Cyanobacteria | Stanieria cyanosphaera PCC 7437 | 1 | ||||||||||||||||||
| GNS bacteria | Chloroflexus aurantiacus J-10-fl | 1 | ||||||||||||||||||
| Firmicutes | Bacillus subtilis strain 168 | 1 | ||||||||||||||||||
| Spirochaetes | Leptospira meyeri serovar Hardjo str. Went 5 | 1 | ||||||||||||||||||
| Planctomycetes | Isosphaera pallida ATCC 43644 | 1 | ||||||||||||||||||
| Nitrospirae | Candidatus Nitrospira defluvii | 1 | ||||||||||||||||||
| Proteobacteria | Helicobacter pylori OK310 | 5 | ||||||||||||||||||
| Acinetobacter baumannii SDF | ||||||||||||||||||||
| Octadecabacter arcticus 238 | ||||||||||||||||||||
| Achromobacter xylosoxidans A8 | ||||||||||||||||||||
| Comamonas testosteroni S44 | ||||||||||||||||||||
| Eukaryotes | Acanthamoeba | Acanthamoeba castellanii str. Neff | 1 | |||||||||||||||||
| Fungi | Magnaporthe oryzae 70-15 | 12 | ||||||||||||||||||
| Torulaspora delbrueckii | ||||||||||||||||||||
| Vanderwaltozyma polyspora DSM 70294 | ||||||||||||||||||||
| Gibberella zeae PH-1 | ||||||||||||||||||||
| Penicillium chrysogenum Wisconsin 54-1255 | ||||||||||||||||||||
| Magnaporthe oryzae 70-15 | ||||||||||||||||||||
| Tuber melanosporum Mel28 | ||||||||||||||||||||
| Ustilago maydis 521 | ||||||||||||||||||||
| Verticillium albo-atrum VaMs.102 | ||||||||||||||||||||
| Schizosaccharomyces pombe 972h- | ||||||||||||||||||||
| Coprinopsis cinerea okayama7#130 | ||||||||||||||||||||
| Puccinia graminis f. sp. tritici CRL 75-36-700-3 | ||||||||||||||||||||
| Green plants | Oryza sativa Japonica Group | 3 | ||||||||||||||||||
| Solanum lycopersicum | ||||||||||||||||||||
| Physcomitrella patens subsp. patens | ||||||||||||||||||||
| Diatoms | Thalassiosira pseudonana CCMP1335 | 2 | ||||||||||||||||||
| Phaeodactylum tricornutum CCAP 1055/1 | ||||||||||||||||||||
| Heterolobosea | Naegleria gruberi strain NEG-M | 1 | ||||||||||||||||||
| Alveolata | Perkinsus marinus ATCC 50983 | 1 | ||||||||||||||||||
После составления выборки, были получены fasta-файлы последовательностей гомологов белка TENA_BACSU (ссылка на fasta-файл со всеми полученными последовательностями).
Файл с последовательностями использовался для получения множественного выравнивания при помощи программы Muscle.
Выравнивание оказалось большим (рис. 3), но достаточно информативным.
 Рисунок 3. Выравнивание гомологов белка TENA_BACSU (полноразмерное изображение можно найти по этой ссылке). Серым цветом выделен идентификатор исходного белка. Последовательность белка TENA_BACSU находится последней в списке.
Использована стандартная окраска аминокислотных остатков ClustalX. Также к выравниванию было добавлено несколько строк аннотаций:
Рисунок 3. Выравнивание гомологов белка TENA_BACSU (полноразмерное изображение можно найти по этой ссылке). Серым цветом выделен идентификатор исходного белка. Последовательность белка TENA_BACSU находится последней в списке.
Использована стандартная окраска аминокислотных остатков ClustalX. Также к выравниванию было добавлено несколько строк аннотаций:
Следует отметить, что для гомологов белка из надцарства эукариот свойственны достаточно большие последовательности в сравнении с последовательностью самого белка TENA_BACSU и его гомологами из надцарства прокариот. (количество а.а. в эукариотных гомологах приблизительно 500 и чуть выше, тогда как в самом белке TENA_BACSU их всего 236)
В целом, аминокислотная последовательность гомологов белка оказалась почти консервативна.
Наихудшим образом выровнены концевые участки белков ( особенно в случае 5 последних эукариотных белков). Это связано с тем, что они не учавствуют в формировании вторичной структуры белка и являются петлями, которые могут уменьшаться, увеличиваться или в них могут могут возникать ни на что не влияющие мутации.
Можно было догадаться, что участки консервативности напрямую связаны с элементами вторичной структуры белка: они почти полностью покрывают каждый из таких участков. Но не все участки вторичной структуры являются строго консервативными.
На элементы вторичной структуры белка почти не приходится колонок-гэпов в отличие от других участков белка. Практически все выбранные мною блоки соотвествуют участкам вторичной структуры и аминокислотам, связывающих лиганды. Блоки выбирались согласно критерию, приведённому в подсказаках: "всегда под 'блоками' подразумевают как-то выровненные по всей толщине (не обязательно сильно консервативные) участки выравнивания, противопоставляя им вообще не выровненные петли".
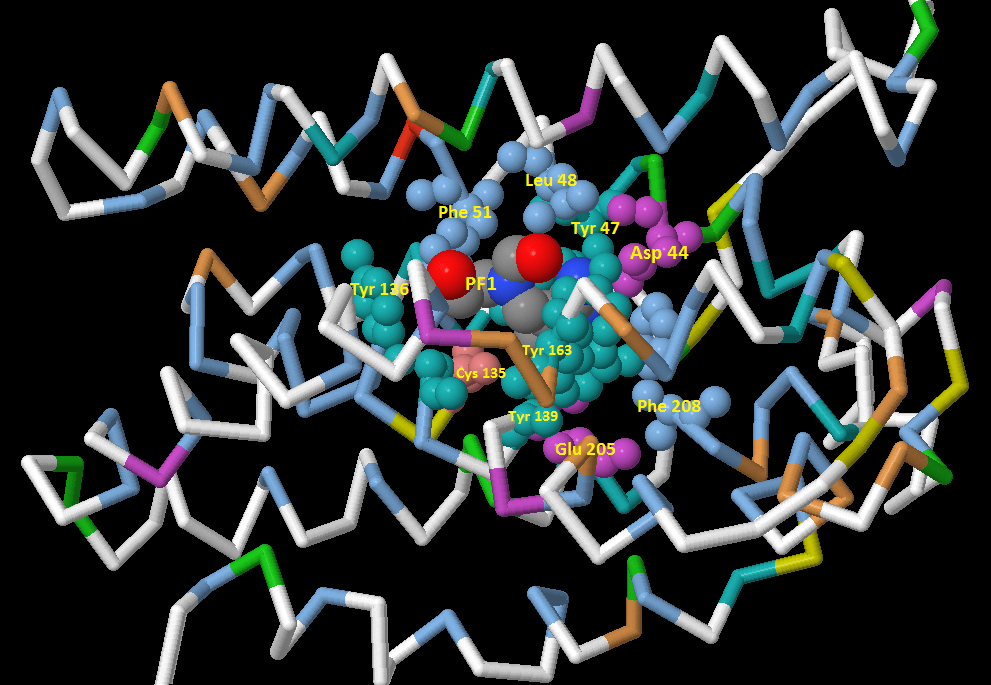
Как выяснилось, в структуре изучаемого белка имеются как предельно консервативные аминокислоты, связывающие лиганд, так и совсем не консервативные. В моем случае, была попытка проанализировать свойства аминокислот, связывающих лиганд - PF1 (риc. 4).
 |