Далее создадим скрипт, вызов которого даёт в программе RasMol последовательно следующие изображения:
- вся структура,
- только ДНК (в проволочной модели);
- проволочная модель ДНК с выделенными шариками множества атомов set1,
- проволочная модель ДНК с выделенными шариками множества атомов set2,
- проволочная модель ДНК с выделенными шариками множества атомов set3.
Таблица 1. Изображения, полученные с помощью скрипта
| Вся структура |  |
|---|---|
| Только ДНК |  |
| Проволочная модель ДНК с выделенными шариками множества атомов кислорода 2'-дезоксирибозы |  |
| Проволочная модель ДНК с выделенными шариками множества атомов кислорода в остатке фосфорной кислоты |  |
| Проволочная модель ДНК с выделенными шариками множества атомов азота в азотистых основаниях | 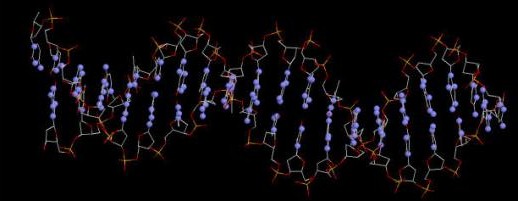 |
Описание ДНК-белковых контактов в заданной структуре
Для исследования ДНК-белковых контактов использована структура 1R4O.pdb.
Договоримся считать полярными атомы кислорода и азота, а неполярными - атомы углерода, фосфора и серы.
Тогда полярным контактом будет называться случай, в котором расстояние между полярным атомом белка и полярным атомом ДНК меньше 3.5Â. Аналогично, неполярным контактом будем считать пару неполярных атомов на расстоянии меньше 4.5Â.
Написан скрипт sk3.spt, последовательно выводящий на экран изображения структуры и атомов, необходимых для определения ДНК-белкового контакта соответствующего типа. Результаты определения числа контактов приведены в таблице 2 ниже:
Таблица 2. Контакты разного типа в комплексе 1R4O.pdb
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 1 | 20 | 21 |
| остатками фосфорной кислоты | 13 | 16 | 29 |
| остатками азотистых оснований со стороны большой бороздки | 4 | 9 | 13 |
| остатками азотистых оснований со стороны малой бороздки | 0 | 1 | 1 |
Как видно из таблицы, неполярных контактов белка с молекулой ДНК больше, чем полярных, предположительно это связано с тем, что определение полярного контакта более строгое, чем неполярного - 3.5Å против 4.5Å, да и неполярных атомов в ДНК и белке больше, чем полярных. Нетрудно заметить, что наибольшее число контактов: между белком и остатками фосфорной кислоты, возможно это объясняется тем, что атомы остатков фосфорной кислоты наиболее доступны для взаимодействия благодаря своему расположению. Также наблюдается отсутствие полярных контактов атомов белка с остатками азотистых оснований со стороны малой бороздки, скорее всего это связано с тем, что атомы азотистых оснований, направленных в сторону малой бороздки пространственно недоступны.
Получение популярной схемы ДНК-белковых контактов
Для получения популярной схемы ДНК-белковых контактов использовалась программа nucplot. Эта программа работает только со старым форматом .pdb, для работы с ней был использован файл 1R4O_old.pdb, полученный с помощью программы remediator:
nucplot 1R4O_old.pdb
Одним из полученных файлов является файл nucplot.ps, содержание которого представлено на изображении ниже:

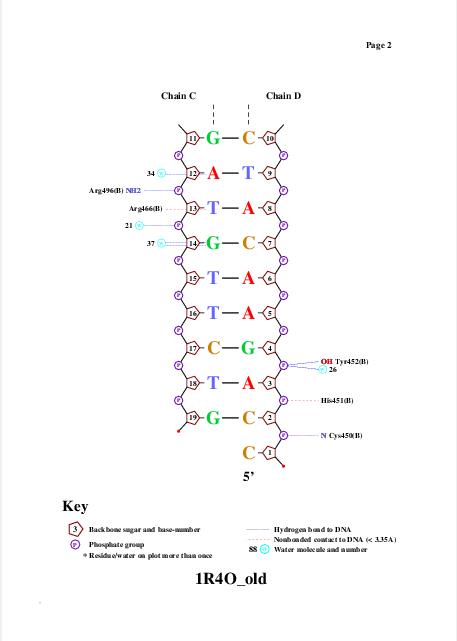
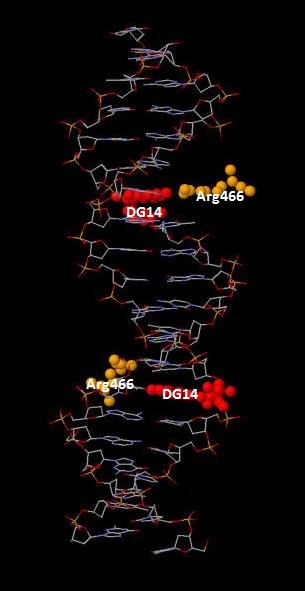
На данной схеме видно, что наибольшее число контактов с ДНК образует аминокислотный остаток Lys461(A) (3 контакта с DG4). Контакты этого аминокислотного остатка с ДНК показаны на рисунках 2 и 3, полученных с помощью программы RasMol:
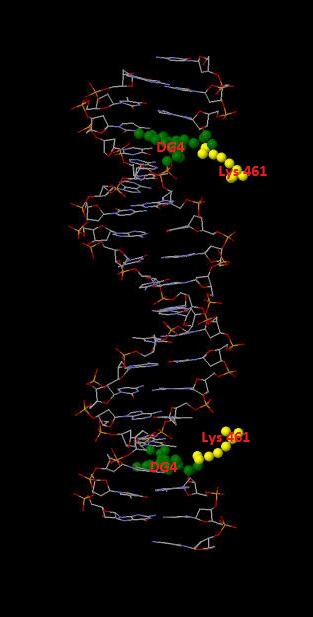
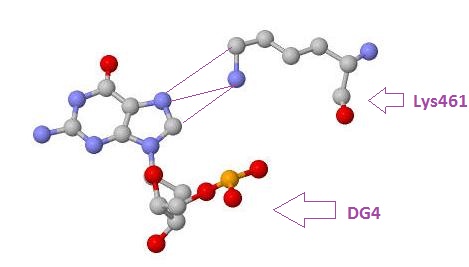
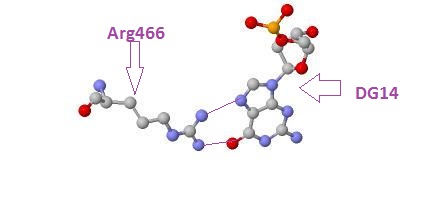
Для участия в распознавании последовательности ДНК аминокислотный остаток, очевидно, должен связываться с азотистым основанием. Вероятно, что Arg466 наиболее важнен для распознавания последовательности ДНК, так как он имеет в некотрых местах по 2 водородные связи, к тому же данная аминокислота соединяется именно с азотистым основанием, а не сахаром или остатком фосфорной кислоты. На рисунках 4, 5 показаны контакты Arg466 с ДНК.
Предсказание вторичной структуры заданной тРНК
Программа einverted из пакета EMBOSS позволяет найти инвертированные участки в нуклеотидных последовательностях. Найдём возможные комплементарные участки в последовательности исследуемой тРНК (последовательность тРНК можно найти в файле 1FFY.fasta) с помощью следующей команды:
einverted 1FFY.fasta
При параметрах по умолчанию инвертированных участков найдено не было. Поэтому я подобрала более удачные параметры :
- Gap penalty: 5
- Minimum score threshold: 10
- Match score: 3
- Mismatch score: 0
- Для антикодонового стебля
- Gap penalty: 2
- Minimum score threshold: 10
- Match score: 2
- Mismatch score: -1
- Для акцепторного стебля
Предсказание вторичной структуры тРНК по алгоритму Зукера
Алгоритм Зукера реализует программа mfold. Единственный параметр, интересующий нас на данном этапе это - P,
который указывает, на сколько процентов выдаваемое предсказание структуры может отличаться по своей вычисленной энергии от оптимального
(соответственно, чем больше значение этого параметра, тем больше вариантов предсказания будет выдано).
Можно использовать программу mfold онлайн на сервере Mobyle @Pasteur.
Применение программы mfold для последовательности 1FFY.fasta с минимальным параметром P = 7% приводит к получению предсказания, близкого к реальной структуре. На рисунке 6, мы можем наблюдать предсказание вторичной структуры РНК по алгоритму Зукера.
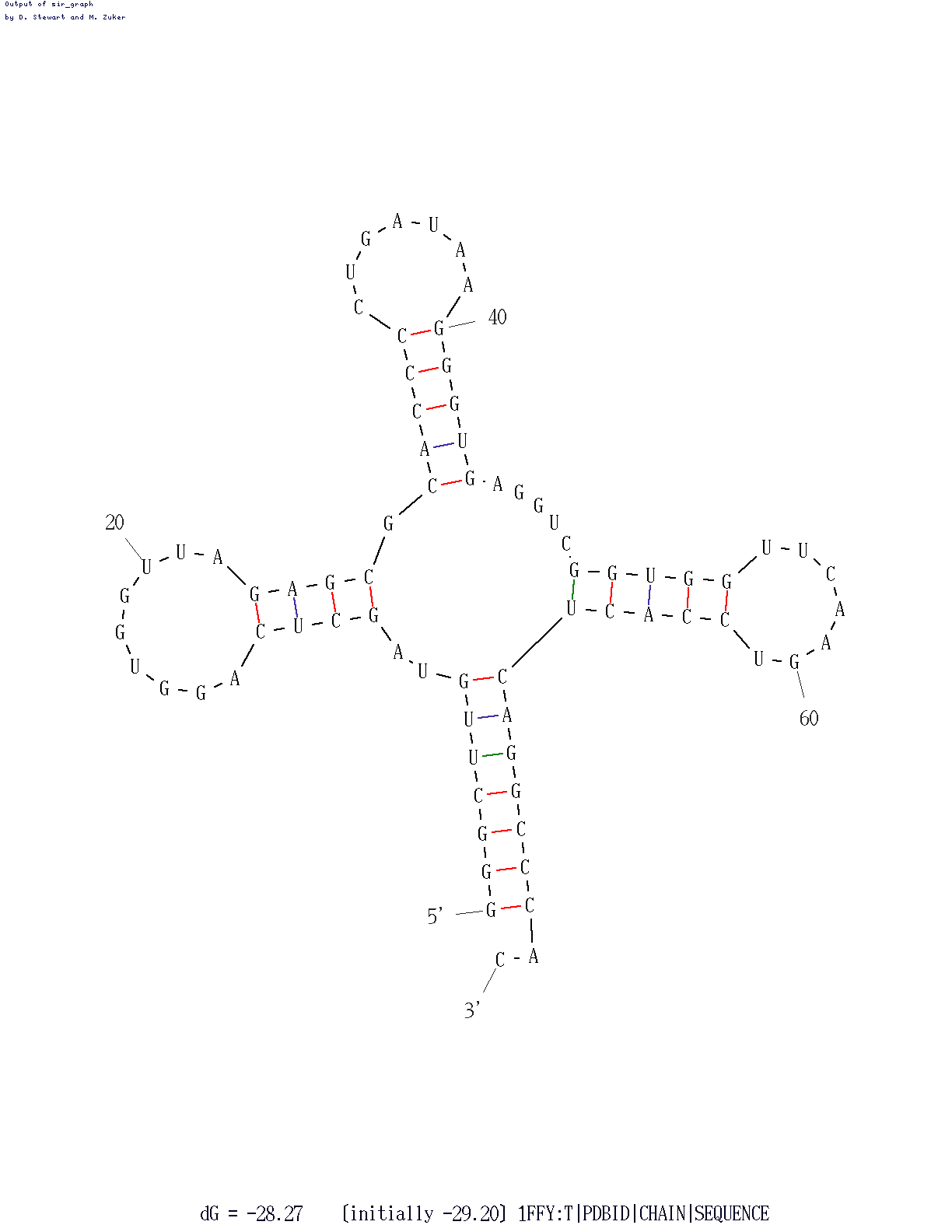
Таблица 3. Реальная и предсказанная вторичная структура тРНК из файла 1FFY.pdb
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5'-1-7-3' 5'-66-72-3' Всего 7 пар | Нет предсказаний | 5'-1-7-3' 5'-67-73-3' Всего 7 пар |
| D-стебель | 5'-10-13-3' 5'-22-25-3' Всего 4 пары | Предсказано 6 пар | 5'-10-13-3' 5'-23-26-3' Всего 4 пары |
| T-стебель | 5'-49-53-3' 5'-61-65-3' Всего 5 пар | Нет предсказаний | 5'-50-54-3' 5'-62-66-3' Всего 5 пар |
| Антикодоновый стебель | 5'-38-44-3' 5'-26-32-3' Всего 7 пар | Предсказано 5 пар | 5'-40-44-3' 5'-28-32-3' Всего 5 пар |
| Общее число канонических пар нуклеотидов | 19 | 11 | 19 |
Наиболее точными способами определения вторичной структуры тРНК, на мой взгляд, являются программа find_pair и алгоритм Зукера. Второй способ является предпочтительней, так как создает схему строения тРНК, что гораздо наглядней. При помощи einverted можно предсказать только часть вторичной структуры тРНК, причём результаты сильно меняются в зависимости от введения разных параметров.