Программа getorf используется для работы с записями последовательностей различных банков данных.
Была получена последовательность D89965 из банка EMBL, эта последовательность матричной РНК, полученная в эксперименте из желудка крысы Rattus norvegicus:
entret embl:D89965 -outfile D89965.out
С помощью программы getorf был получен набор трансляций всех открытых рамок нуклеотидной последовательности из данной записи, определенных при использовании стандартного кода и одновременно удовлетворяющих следующим условиям:
getorf -find 1 -maxsize 90 -outfile D89965.orf D89965.out
После проделанной работы было обнаружено 5 последовательностей. Одна из них (D89965_3) соответствует исходной CDS (прямой рамке) из записи. Было замечено, что координаты начала совпадают - 163, координаты конца отличаются на 3 (из-за стоп-кодона) .
Также была получена запись данной последовательности из банка Swiss-Prot: HSLV_ECOLI.fasta (белок из кишечной палочки), на которую ссылается EMBL. Эта последовательность соотвествует другой рамке - D89965_5 (обратной рамке).
Как можно объяснить наблюдаемую ситуацию?
Также известно, что EMBL - база архивная, в которой за достоверность информации отвечает только человек, который её туда поместил. В то же время Swiss-Prot база реферируемая, и поэтому данным из неё можно доверять в значительно большей степени. Таким образом, мы видим ошибку интерпретации результатов сиквенирования (по неверной ORF).
Запущенные команды и их параметры:
seqret sw:adh*_* adh.fasta
infoseq adh.fasta -only -usa > adh.infoseq
grep -f pattern.txt adh.infoseq > adh_ka.infoseq
grep -f pattern.txt adh.infoseq > adh_te.infoseq
seqret @adh_te.infoseq mysequences.fasta
Файл с последовательностями моих алкогольдегидрогеназ : mysequences.fasta
В данном разделе представлена некоторая информация о гене человека, кодирующего белок NCF1_HUMAN (AC в Swiss-Prot: P14598), полученная с помощью сервиса EnsEMBL, используемого для визуализации известной информации о геномах человека и животных.
Далее была попытка найти последовательность гена этого белка, используя сервис "BLAST/BLAT". Но вместо выдачи получили странную страницу:

Тогда с главной страницы EnsEMBL с помощью поиска искался ген по имене белка. Оказалось, что названия гена и белка совпадают. Тогда со страницы гена была открыта страница Region in detail. Некоторая информация раздела выдачи Region in detail перечислена ниже.
Ген закодирован на 7 хромосоме, координаты гена: 74,238,919-74,254,272. На рисунке 1 красной рамочкой обведен участок хромосомы, в котором расположен ген.
Есть разные варианты проверки генов: автоматическая - компьютерная, ручная - человеком. Наиболее надежными транскриптами считаются проверенные обоими путями.
Mожно, выделить касной рамочкой любой участок этой хромосомы. Информация о нём отобразится ниже.

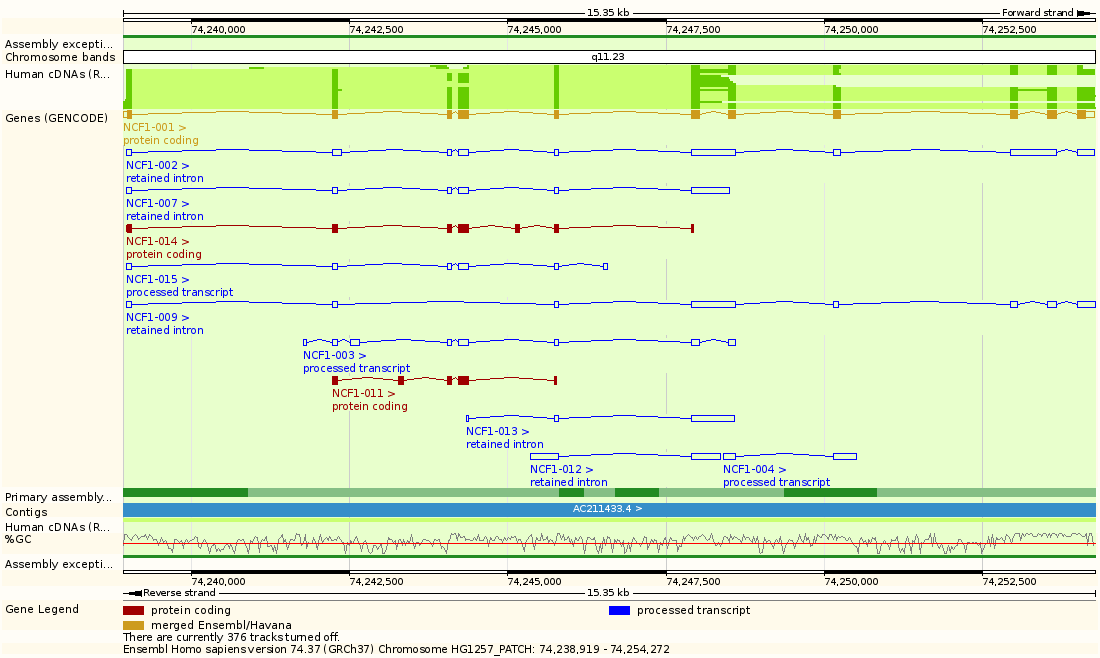
На рисунке 2 представлено более детальное окружение гена. В строке "Contigs" указаны рядом лежащие гены с обозначенными АС. В строке Genes также показаны гены, кодирующие только РНК (выделены фиолетовым). Рисунок 2 это картинка из выдачи по умолчанию. Можно изменять различные параметры рисунка, например, масштаб, искать только экзоны гена, открытые рамки считывания, делать выравнивания с соответствующими участками хромосом различных животных и многое другое. Можно также добавлять свои данные.

На рисунке 3 представлена схема расположения интронов и экзонов в гене по различным данным: CCDS set -consensus CDS, проект по выявлению основных кодирующих учасков генома человека и мыши, аннотированных с большой точностью, RefSeq/ENA, GENCODE.
Кроме того со странички гена возможен переход к скачиванию различного рода информации об этом гене (меню Export Data). Возможно скачивание в различных форматах, есть меню, в котором определяется, что будет входить в итоговый файл, можно захватить, например, определенное число нукеотидов вокруг гена по своему желанию. Помимо выдачи Region in detail существует еще множество разделов в EnsEMBL. Например, в разделе "Chromosome summary" можно увидеть некоторую информацию о генах в разных участках хромосомы в целом.