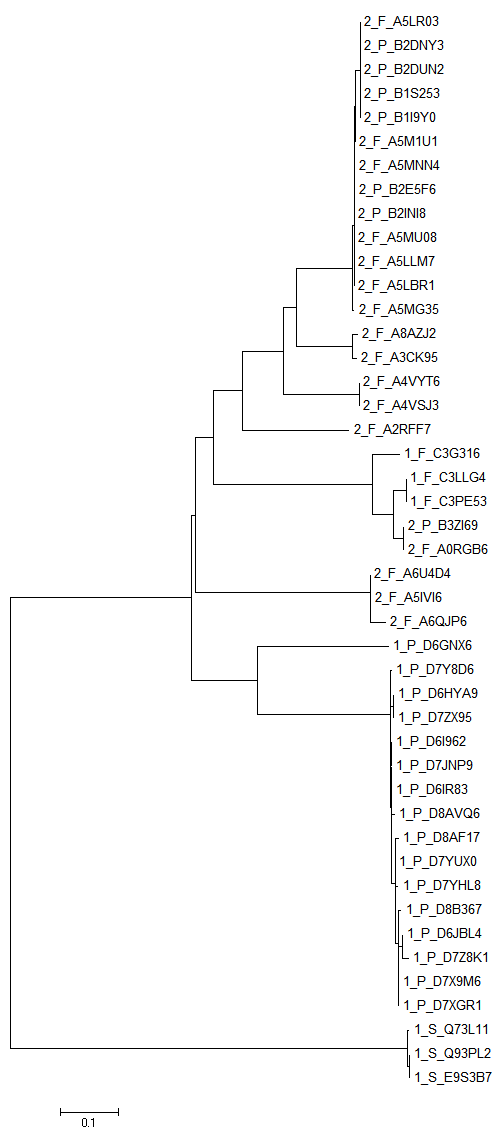
Закодировала доменные архитектуры (1 для YodA, 2 для YodA+TroA), закодировала сравниваемые таксоны (P - Proteobacteria, F - Firmicutes и S - Spirochaetes). К именам всех последовательностей спереди добавила коды архитектуры и таксона. Построила филогенетическое дерево с помощью программы MEGA. Использовала методы Neighbor-Joining (Рис. 1 и 2) (не использует гипотезу о молекулярных часах и строит неукоренённое дерево) и UPGMA (Рис. 3 и 4) (предполагает молекулярные часы, строит укоренённое дерево) с параметрами по умолчанию.




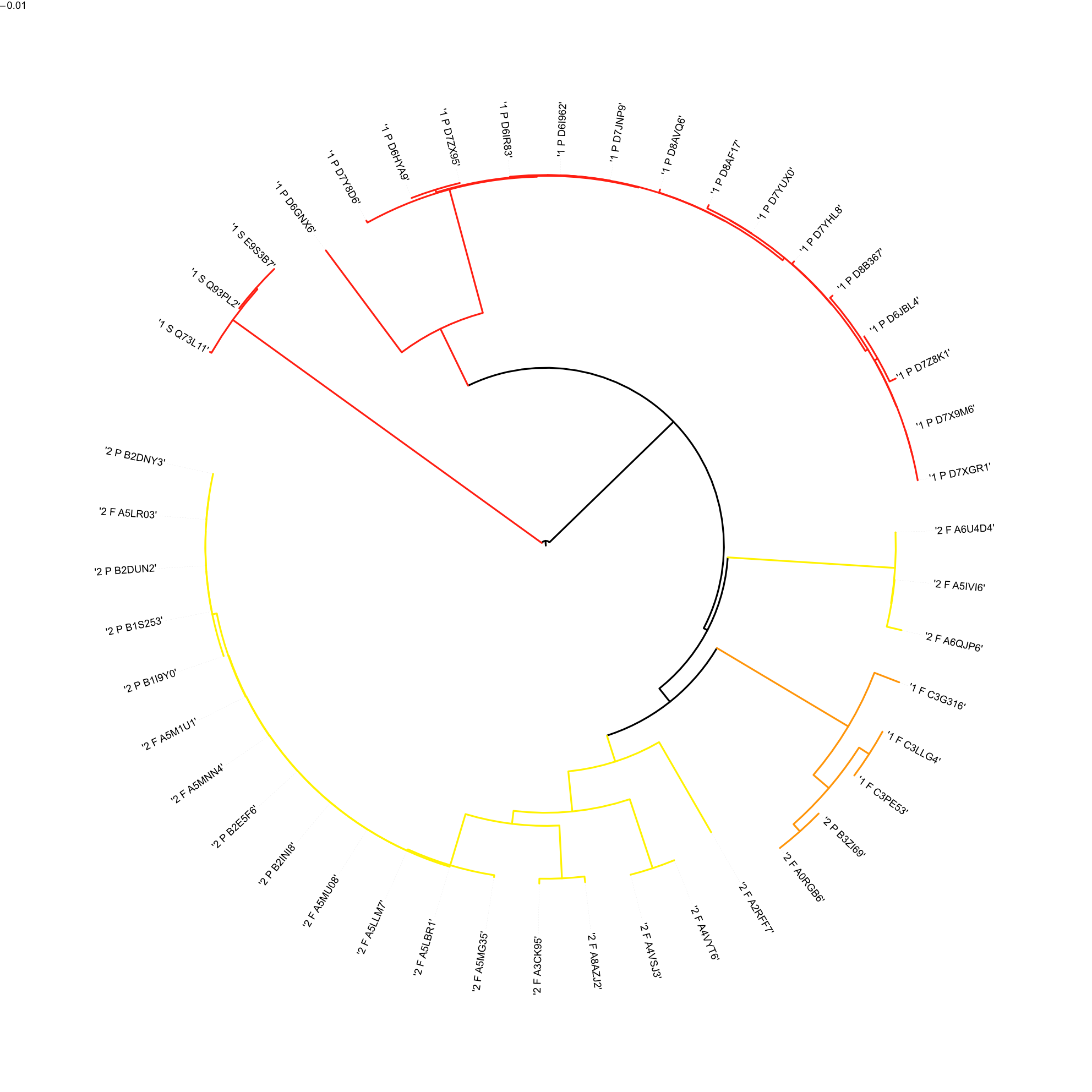
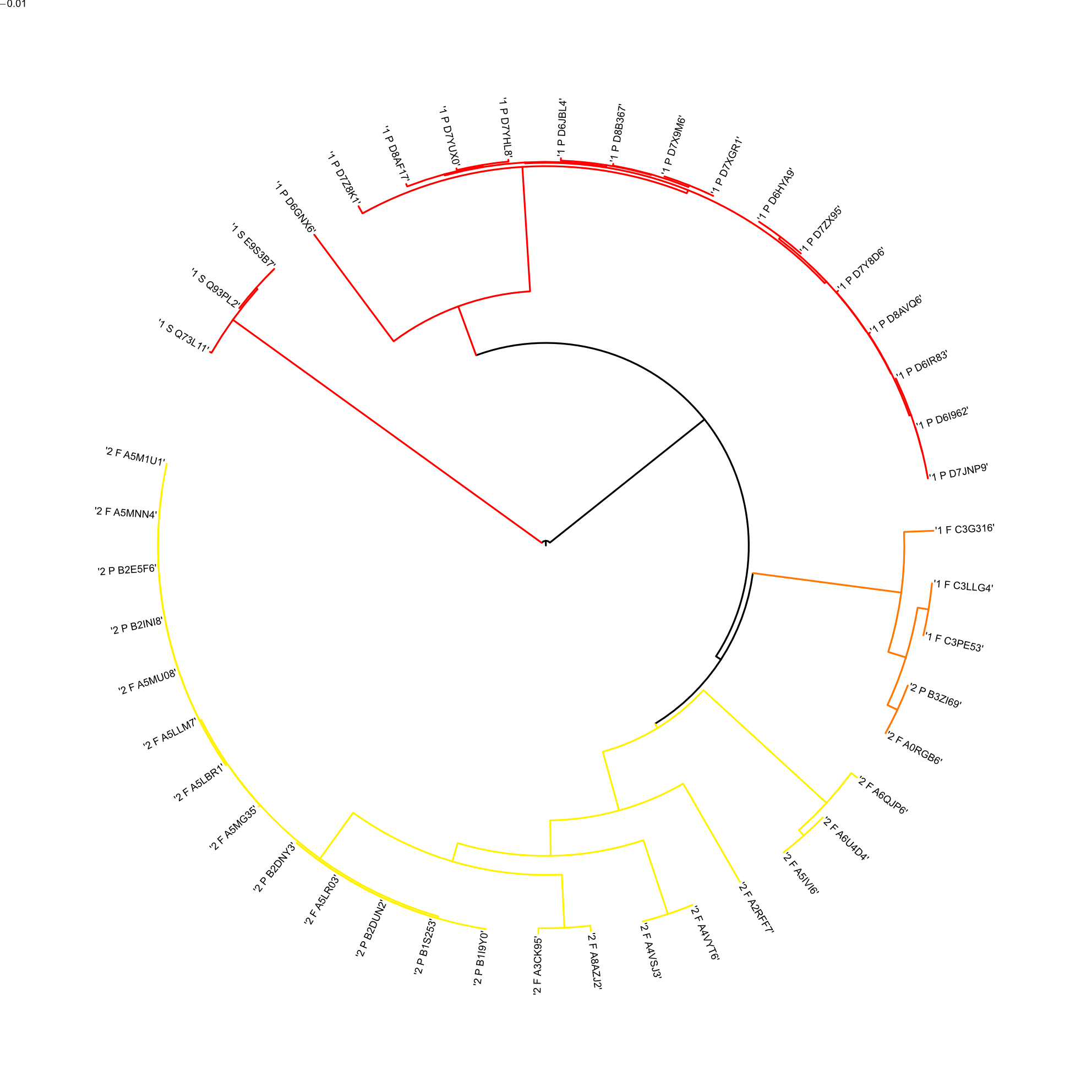
Создала рисунки деревьев с использованием методов выделения ветвей для наглядной демонстрации результата с помощью программы ITOL (рис. 5 и 6).
Скобочные формулы деревьев, построенных методом Neighbor-Joining (1.nwk) и UPGMA (2.nwk) в формате Newick с длиной ветвей.
На получившихся деревьях видно, что однодоменные белки у Spirochaetes не претерпели изменений в течение эволюции. Разделение на однодоменные и двудоменные белки произошло после отделения белков у Spirochaetes примерно в то же время, что и разделение Firmicutes и Proteobacteria. Далее двудоменные белки подвергались дальнейшей эволюции.