Для трех трансмембранных бета-баррелей и трех трансмембранных альфа-спиральных белков (на мой выбор) определила параметры, перечисленные в Таблице 1, и заполнила такую таблицу на своем сайте.
| PDB код | Тип (спираль, баррель) | Какая мембрана (внутренняя или внешняя, организм, органелла) | Толщина гидрофобной части мембраны в ангстремах | Медиана числа остатков в одном трансмембранном участке |
| 3h90 | спираль | Внутренняя, Escherichia coli | 29,6 | 18 |
| 2m6x | спираль | Мембрана ЭПР, Hepatitis C virus | 26,8 | 15 |
| 4aw6 | спираль | Мембрана ЭПР, Homo sapiens | 30,8 | 25 |
| 2qdz | баррель | Внешняя мембрана, Bordetella pertussis | 24,4 | 10 |
| 1k24 | баррель | Внешняя мембрана, Neisseria meningitidis | 25,4 | 11 |
| 2lme | баррель | Внешняя мембрана, Yersinia enterocolitica | 23,4 | 10 |
Из таблицы видно, что в среднем длина трансмембранных участков со спиральным типом больше (примерно 20 аминокислот), а с типом баррель - меньше (около 10 аминокислот). Аномально короткая медиана в 2m6x объясняется тем, что белок, продуцируемый вирусом гепатита C, находится в человеческой мембране ЭПР.
Отобрала репрезентативную выборку гомологов выданного белка 2WSW (15 штук). Данный белок взят из Proteus mirabilis, эта бактерия относится к филуму Proteobacteria. Поэтому на сайте NCBI я исключила поиск по данному филуму, по эукариотам, выставила порог e-value равный 1 и количество последовательностей, выдаваемых при поиске 5000. В качестве базы для поиска выбрала RefSeq. Полученные находки содержали всего несколько хитов с e-value, близким к единице, поэтому выборку гомологов можно считать достоверной. Далее, пользуясь таксономическим деревом, выбрала 15 последовательностей для выравнивания.
Использовала базу данных OPM для того, чтобы найти в ней выданный белок 2WSW.
| PDB ID | Организм | Тип мембраны | TC-код | Угол наклона спиралей (β-тяжей) к нормали | Количество трансмембранных спиралей (β-тяжей в бочонке) |
| 2WSW | Proteus mirabilis | Внутренняя грам-отрицательная | 2.A.15.2.2 | 14 | 12 |
Для данного белка ТС-код означает:
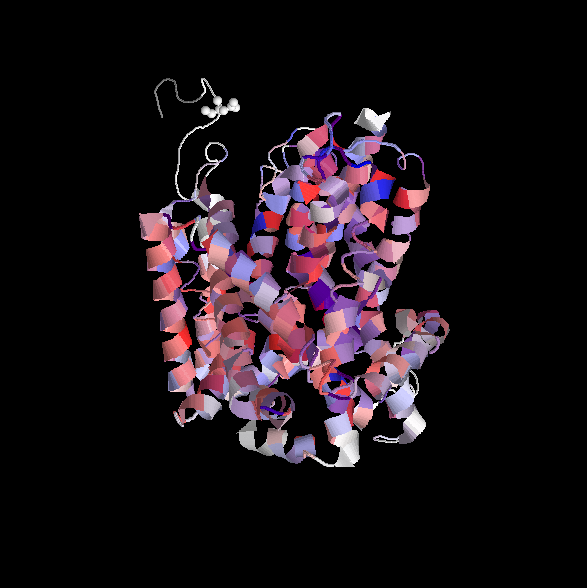
Построила множественное выравнивание отобранных гомологов с помощью программы Muscle. Загрузила множественное выравнивание в программу JalView. Добавила к выравниванию дополнительную аннотацию положения трансмембранных спиралей. Для этого добавила новую пустую строку аннотации и назвала ее "TM_REAL". Переместила исходный белок, для которого есть структура, в верхнюю строку выравнивания. Прикрепила к нему структуру 2WSW, цепь А. Используя появившуюся связь между последовательностью и структурой, пометила участки выравнивания, отвечающие трансмембранным спиралям в белке со структурой в строке-аннотации буквой "М". Добавила к выравниванию предсказание трансмембранных спиралей, выдаваемых программой TMHMM, для гомолога WP_006768969.1| Corynebacterium efficiens. Взяла последовательность гомолога и получила для него результат предсказания TMHMM. Создала новую строку аннотации и назвала ее "TM_PREDICTED", после чего нанесла вручную участки, предсказанные TMHMM, в эту строку. Выбрала цветовую схему, позволяющую визуально различать гидрофобные и гидрофильные остатки. Затем установила галочку на "By Conservation" (теперь интенсивность цвета зависит от того, насколько позиция консервативна). Покрутила белок так, чтобы его часть, ориентированная в n-сторону мембраны оказалась сверху, а ориентированная в p-сторону - снизу. Сохранила полученное изображение структуры белка (рисунок 1) и изображение выравнивания (рисунок 2).

В моем белке участки, относящиеся к трансмембранным спиралям, довольно консервативны. Участки между спиралями менее консервативны. В трансмембранных спиралях есть заряженные остатки и просто полярные остатки, их наличие не противоречит формированию альфа-спирали. Результаты программы TMHMM и реальной структурной информации совпадают по количеству выявленных трансмембранных участков и перекрываются на 50-85%. Я считаю, что это может быть связано с тем, что в моем белке много гидрофобных аминокислот, что затрудняет определение трансмембранных участков.