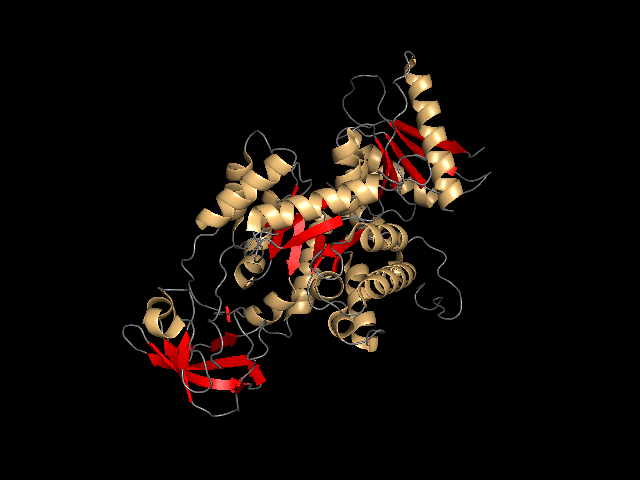
Рисунок 1.1. Разметка вторичной структуры 4FXJ в .pdb-файле. Золотым цветом обозначены α-спирали, красным - β-тяжи.
Для выполнения задания была взята структура 4FXJ. 4FXJ - структура M2 пируват киназы в комплексе с фенилаланином. На рисунке 1.1 представлена цепь A белкового комплекса. Разметка данной структуры была взята, исходя из аннотации в .pdb-файле.

Для предсказания вторичной структуры использовалась программа DSSP. Границы спиралей и тяжей в выдаче программы DSSP оказалась близки к таковым, указанным в pdb-файле модели структуры 4FXJ. Интересно, что Stride) выдал пустой результат. Результаты работы программы DSSP и сравнение их с разметкой в .pdb-файле приведены в таблице 1.
| № | Аннотация PDB | Разметка DSSP |
| α-спираль | ||
| 1 | 57-68 | 58-67 |
| 2 | 80-96 | 81-95 |
| 3 | 145-149 | отсутствует |
| 3 | 163-168 | 164-167 |
| 4 | 222-235 | 223-234 |
| β-тяж | ||
| 1 | 46-50 | 46-50 |
| 2 | 71-75 | 71-75 |
| 3 | 109-113 | 109-113 |
Как видно из таблицы 1, программа DSSP в сравнении с разметкой в .pdb-файле предсказывает α-спирали менее точно, чем β-тяжи.
С помощью SheeP была построена карта β-листа в цепи A структуры 4FXJ (были использованы параметры по умолчанию) (рис. 1.2). По карте видно, что выбранный β-лист состоит из восьми тяжей. Изображение его в структуре приведено на рисунке 1.3. Один столбец карты соответствует одному «хребту» (crest) β-листа. Один из таких «хребтов», соответствующий аминокислотам в одном столбце карты, показан на рисунке 1.4. По распределению гидрофильных и гидрофобных остатков на карте оказалось трудно сразу определить, какие остатки листа обращены внутрь гидрофобного ядра, а какие - наружу. Посмотрев на расположение активного центра, можно сделать вывод, что "желтой" стороной β-лист обращен к гидрофобному ядру.
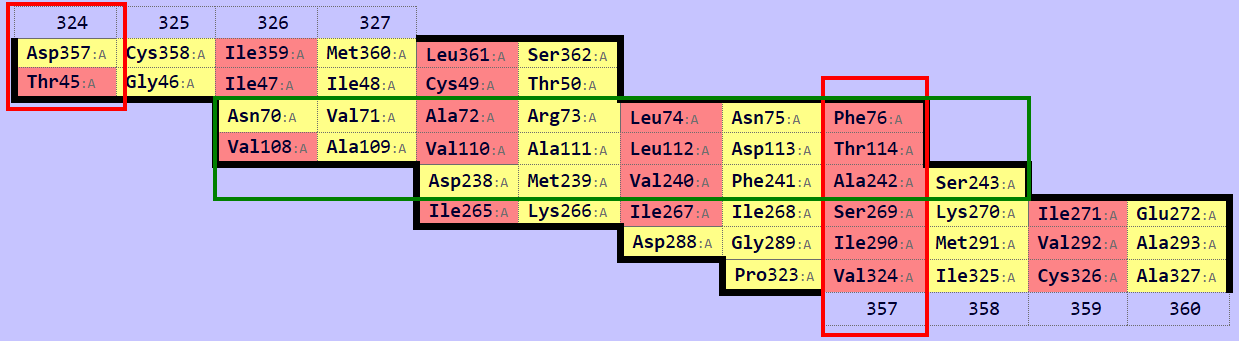
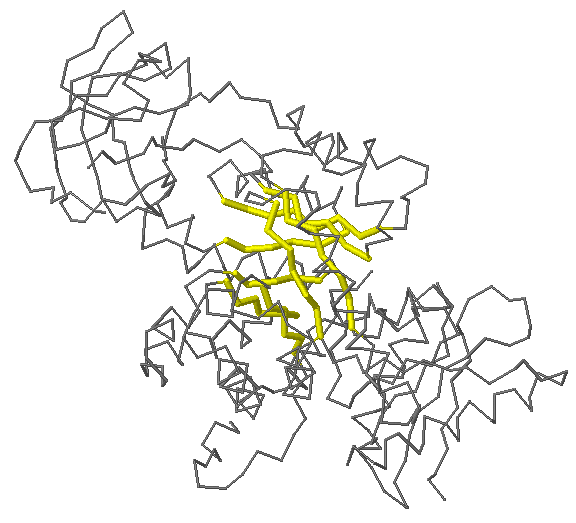
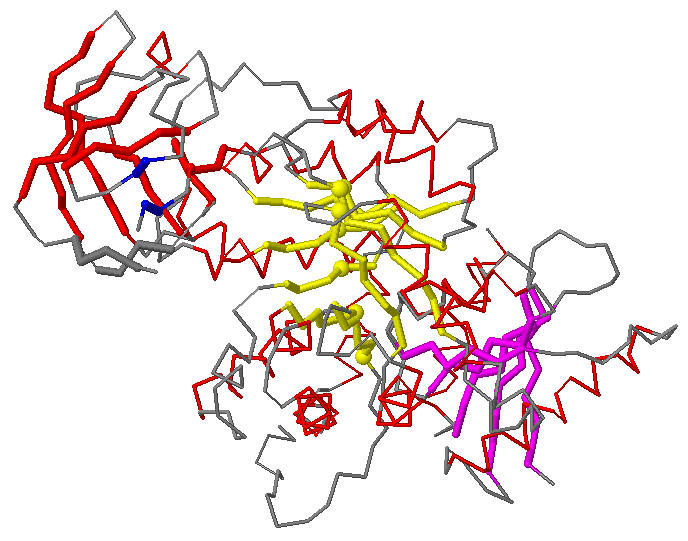
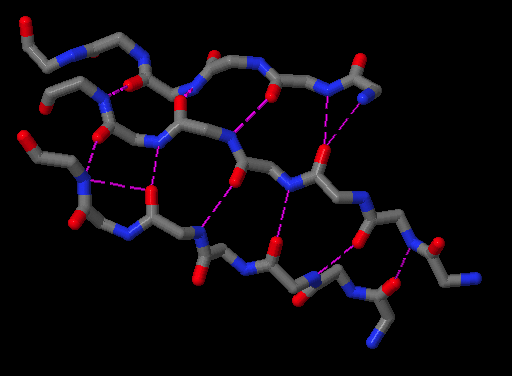
Для участка β-листа (3 тяжа, 4-5 гребней) (рис. 1.2) на основании данных программы stride была получена схема водородных связей (рис. 1.5).
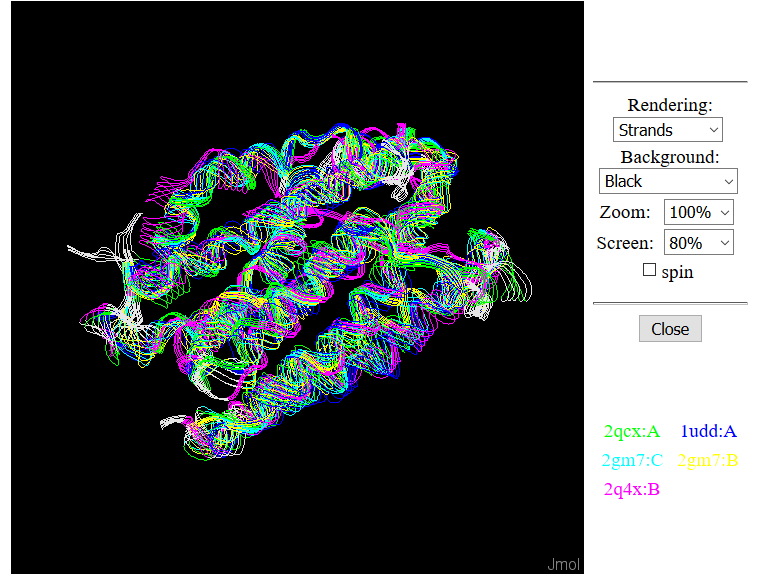
С помощью сервиса PDBeFold были выбраны 4 структурных гомолога 2QCX: 1UDD, 2GM7, 2Q4X. Для гомологов 2QCX и самого белка были загружены выравнивание последовательностей по совмещению структур (попарное и множественное) и само совмещение структур представлено на рисунке 2.1:
Выравнивание по структуре (большие буквы в файле с выравниванием) можно сравнить с выравниванием последовательностей, выполненным, например, в программе JalView (с помощью Tcoffee со стандартными параметрами):
При сравнении множественного выравнивания последовательностей и выравнивания последовательностей по структуре удаётся выявить несколько различий. Так, в выравнивании по последовательности Glu150 выравнивается с Gly150, в выравнивании по структуре мы такого не наблюдаем. Можно отметить, что все найденные несоответствия наблюдаются именно в неструктурированных участках (или иногда захватывают граничные остатки элементов вторичной структуры) и отсутствуют в спиралях или β-тяжах.
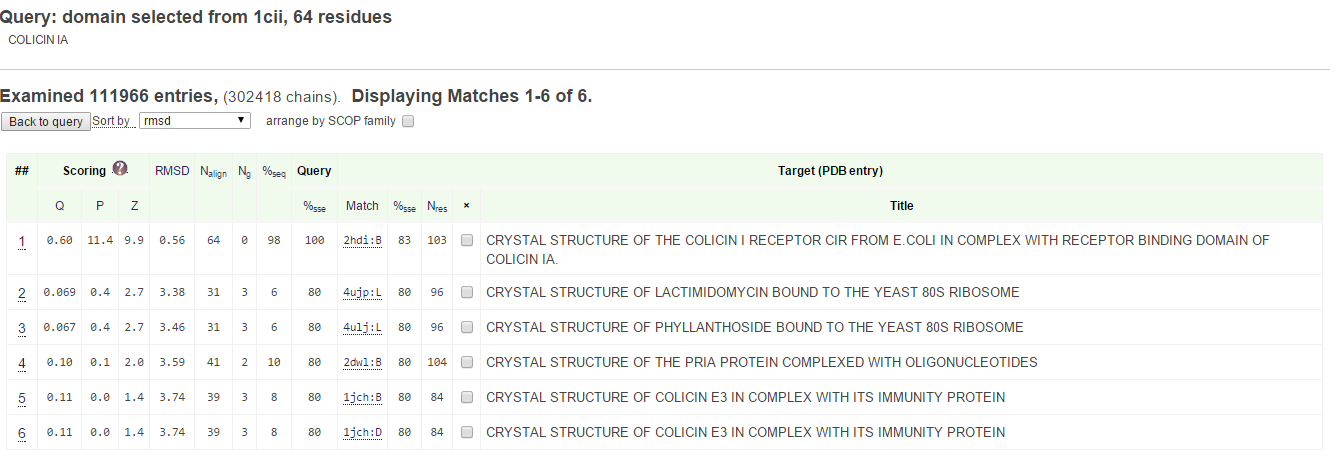
При попытках визуализировать подобные несоответствия возникли проблемы с сохранением информации из интернет-версии Jmol. К сожалению, исправить проблему не удалось.В сервисе PDBeFold был осуществлен поиск стрктурных гомологов для домена 1cii A: 284-384. Однако сам этот домен найден не был. На рисунке 2.5 показана выдача с сайта. Результаты отсортированы по RMSD.
Для выполнения данного заданий были выбраны структуры константного домена человеческого Т-клеточного рецептора из цепи α (1kgc, region d:118-206) и из цепи β (1kgc, region e:119-247) (рисунок 2.6). .
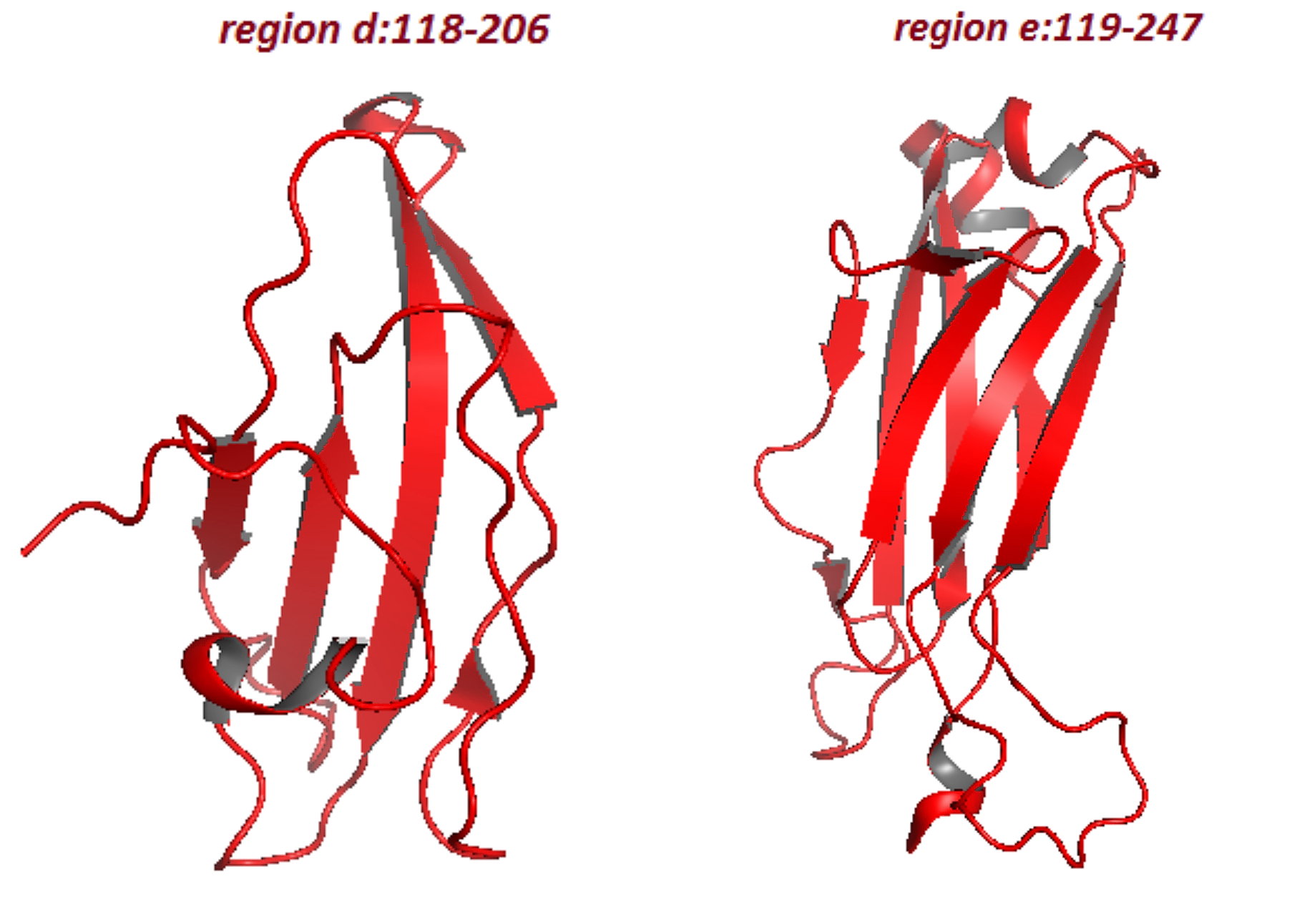
Рисунок 2.7. Карта бета-листа для домена из цепи α (map0).
Карты этих листов находятся в одной ориентации.
Было построено выравнивание этих листов. Консервативные цистеины должны оказаться выровненными. Их выравнивание задает выравнивание всего центрального тяжа. Для совмещения были взяты по 6 остатков из каждого тяжа, так, чтобы консервативные остатки были в одинаковых позициях. Для совмещения использовалась команда:
select alph, alpha and name CA and resi (137-142+177-180) select bet, beta and name CA and resi (146-151+192-195) pair_fit alph, bet
Ниже приведено изображение участков совмещенных структур.

Рисунок 2.10. Совмещение доменов T-клеточного рецептора. Большинство тяжей совпадает.
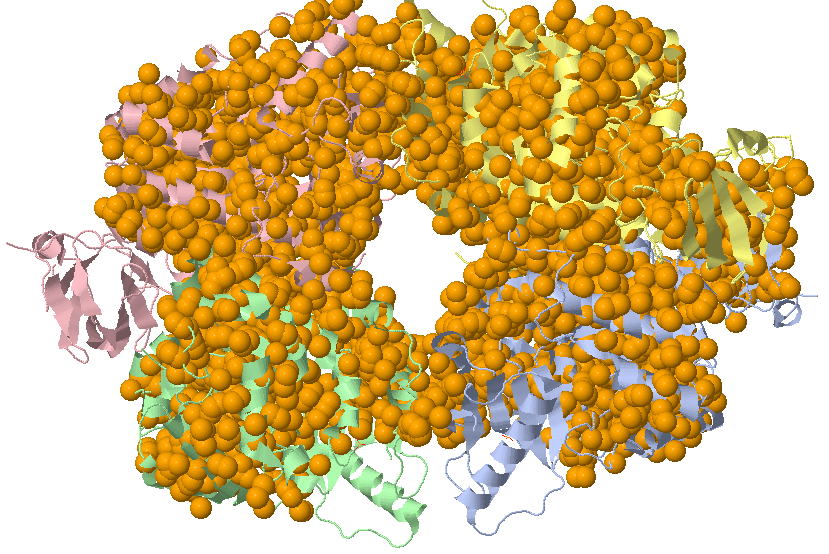
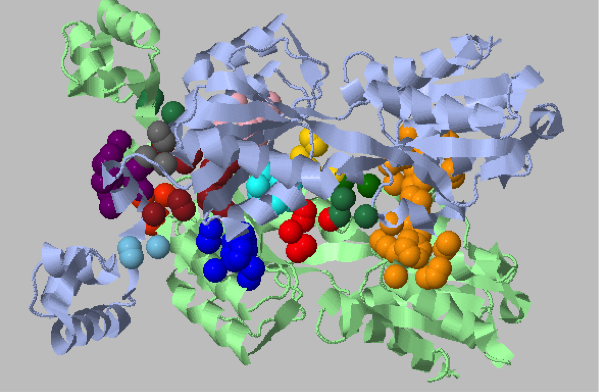
С помощью сервиса Clud в структуре 4FXJ был произведён поиск гидрофобных кластеров. Со значениями 5.4 и 3 для порога расстояния и размера кластера, соответственно, было найдено 2 кластера, охватывающих сразу всю структуру и две ее цепи (рис. 3.1 и 3.2, соответственно). Также было обнаружено 7 гидрофобных кластеров в каждой из цепей, в силу их симметричности была рассмотрена только цепь А (рис. 3.3). Пожалуй, можно сказать, что найденные кластеры отвечают группам (с одним и более) элементов вторичной структуры белка, определяемым визуально.
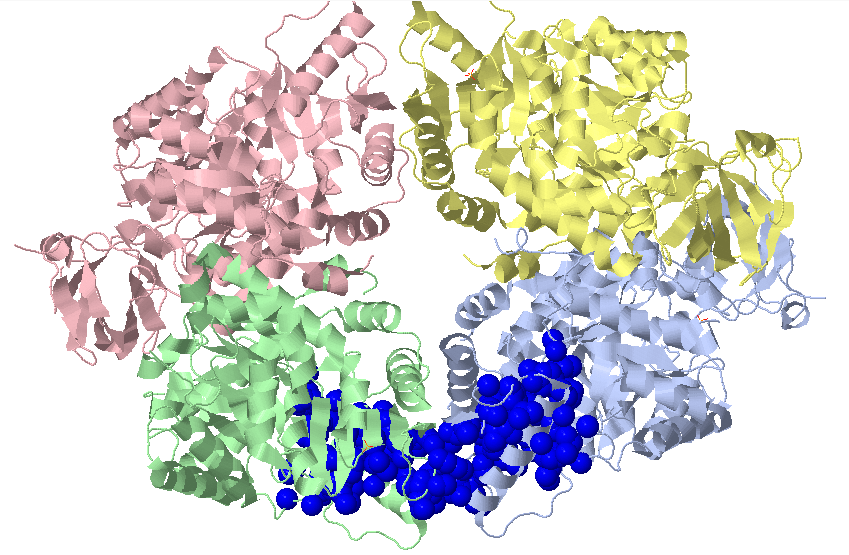

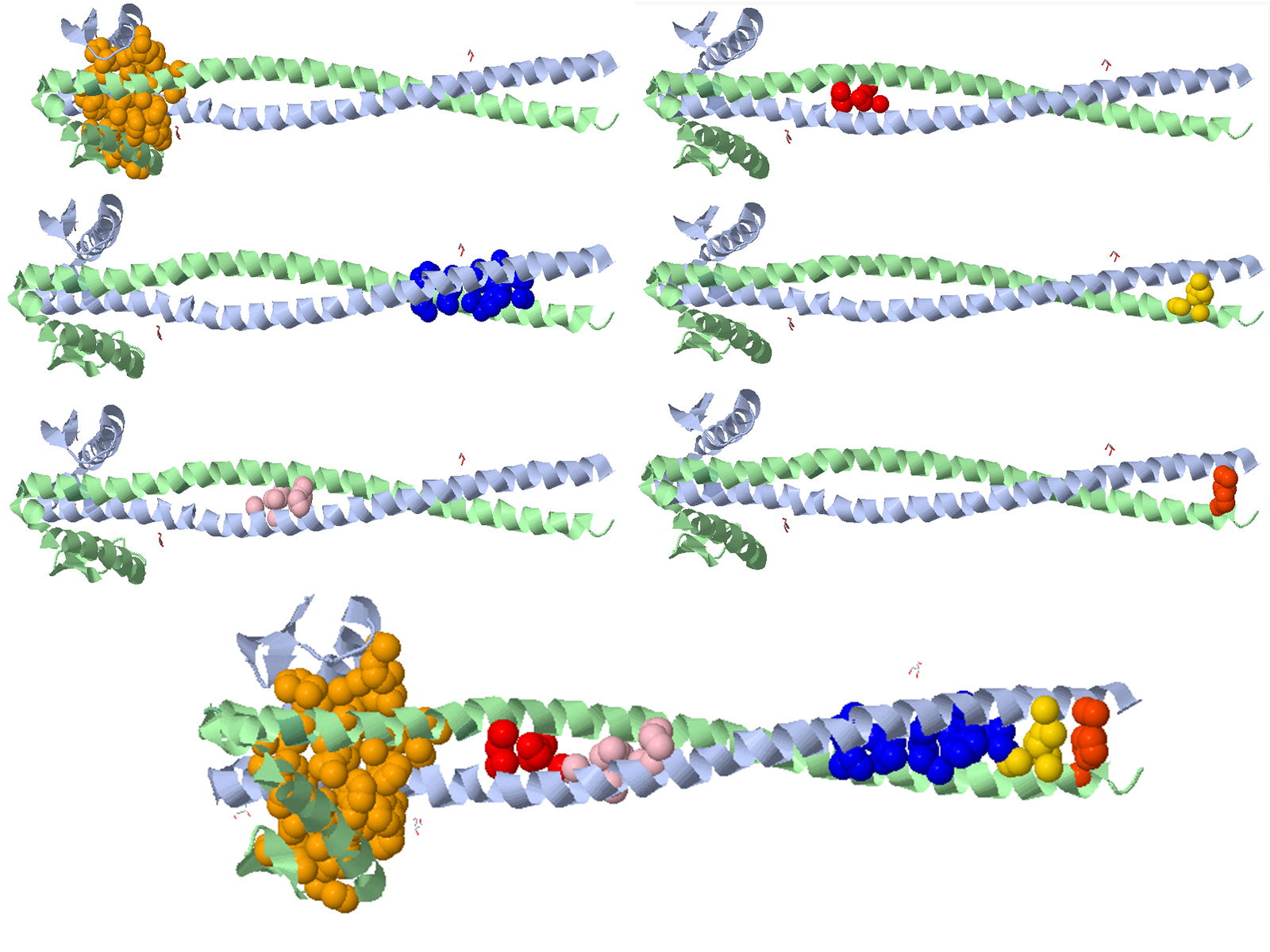
Поиск гидрофобных кластеров в димере белка 3HNW (цепях A и B структуры с мотивом coiled coil) позволяет различить гидрофобные кластеры в участках контакта двух спиралей (значения для порога расстояния и размера кластера – 4.5 и 5, соответственно) (рис. 3.4).
Для комплекса димера пуринового репрессора с ДНК (биологическая единица 1JH9) с использованием средств программы PyMol были созданы изображения, демонстрирующие контакт мономеров белка и между собой и контакт белка с ДНК. Контакты поверхности мономера белка с симметричным мономером и с двойной спиралью ДНК представлены на рисунках 4.1-4.3.

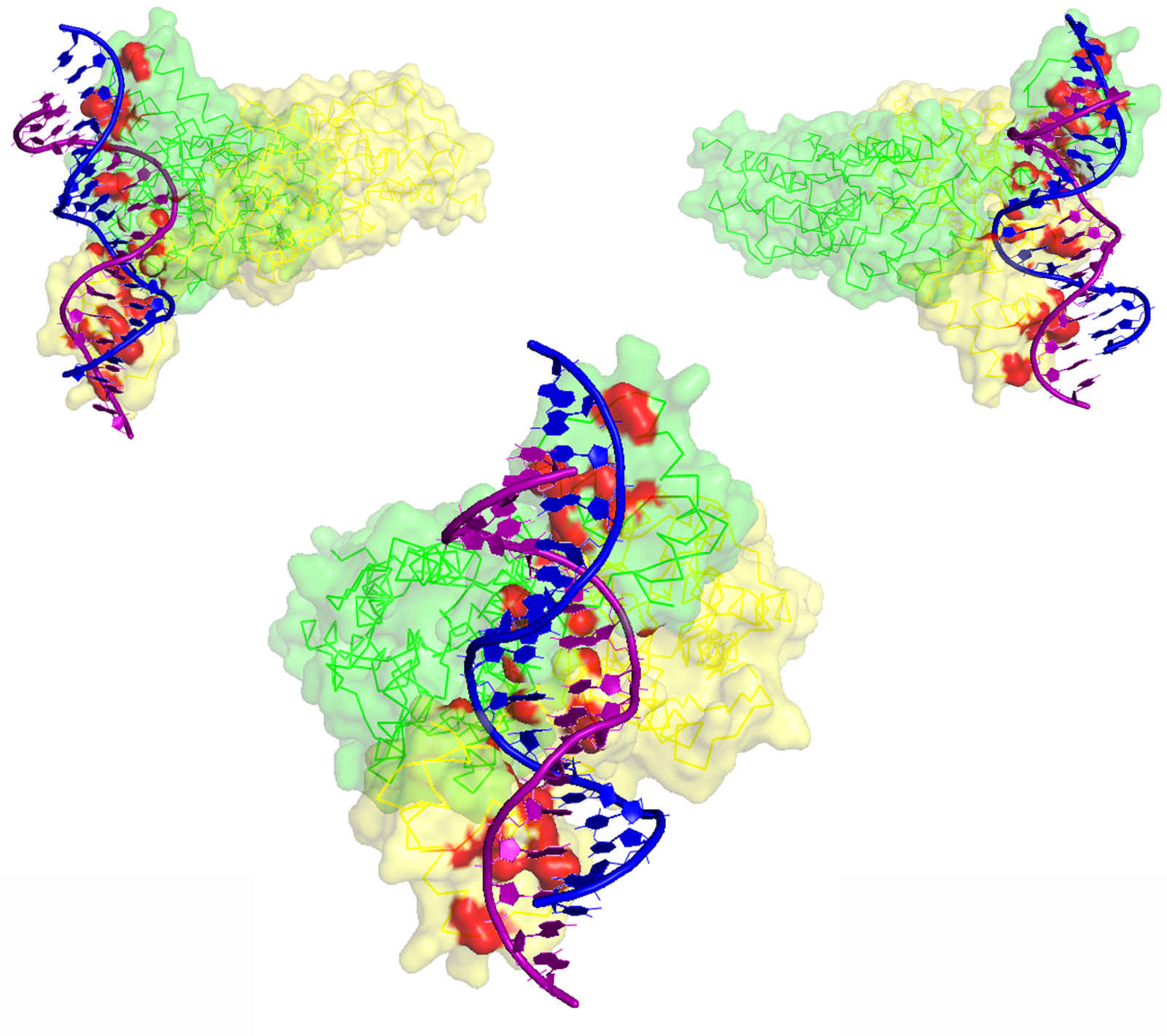
С помощью сервиса Clud в структуре 1JH9 были определены гидрофобные кластеры объёмом не менее 10 атомов на интерфейсе мономеров белка (distance threshold – 5 Å) (рис. 4.4).
Для структуры 1E0T был произведен поиск доменов в Pfam, SCOPe, ECOD и CATH.
Структура 1E0T - R292D мутантная пируват киназа E. coli, гомотетрамер, сотоящий из четырех одинаковых субъединиц (A, B, C и D). Указанные ниже домены найдены для цепи A, и совпадают с доменами для остальных цепей.
Домены, найденные Pfam: домен PK с координатами 1-345 и домен PK_C с координатами 356-468.
Домены, найденные SCOPe: домен d1e0ta1 с координатами 70-167, домен d1e0ta2 с координатами 1-69 и 168-344 и домен d1e0ta3 с координатами 454-470.
Домены, найденные CATH: домен 1e0tA01 с координатами 336-467, домен 1e0tA02 с координатами 2-69 и 169-335 и домен 1e0tA03 с координатами 70-168.
Домены, найденные ECOD: домен e1e0tA4 с координатами 1-70 и 169-344, домен e1e0tA1 с координатами 70-167 и домен e1e0tA3 с координатами 354-470.

Используя Advanced search были заданы параметры, а именно нужный нам метод ELECTRON MICROSCOPY. Было найдено 917 структур, определенных этим методом.
Информацию о последовательностях белков можно найти тут.
С помощью сервиса PDBeFold была получена информация о всех структурных гомологах белка 2QCX; таких гомологов оказалось всего 137 для всех цепей белка. Информация о гомологах.
Также был получен список гомологов 2QCX с помощью программы jFATCAT, которая доступна со страницы PDB. Далее возникли некоторые сложности с сохранением результатов из FATCAT. В итоге выдача была сохранена в формате .xlsx. Следует отметить, что на странице PDB выдача FATCAT представляется не в лучшем виде, абсолютно не читаемые IDs. Поэтому по средствам Ecel эти IDs были приведены к нормалному виду. После этого было проведено сравние результатов, полученных двумя сервисами. Было обнаружено всего 5 совпадений.