
Рисунок 1. Различные уровни подрезки электронной плотности для всей полипептидной цепи.
Я выбрала белок 2QCX - тиаминазу 2. Это довольно хорошо изученный и описанный белок, состоящий из 236 аминокислотных остатков, с которым мне уже приходилось работать в первых семестрах.
Данная структура удовлетворяет набору требований, необходимых для дальнейшей работы, а именно:
| Название | RMSD | N_align | PDB ID |
| TENA HOMOLOGUE PROTEIN FROM P.HORIKOSHII OT3 | 1.47 | 207 | 1udd |
| CRYSTAL STRUCTURE OF TENA HOMOLOGUE (HP1287) FROM HELICOBACTER PYLORI | 1.41 | 206 | 2rd3 |
| CRYSTAL STRUCTURE OF THI-4 PROTEIN FROM BACILLUS SUBTILIS | 0.81 | 211 | 1to9 |
| STRUCTURE OF A PUTATIVE TRANSCRIPTIONAL REGULATOR FROM STREPTOCOCCUS PNEUMONIAE | 1.98 | 203 | 1z72 |
| X-RAY STRUCTURE OF GENE PRODUCT FROM ARABIDOPSIS THALIANA AT3G16990 | 1.87 | 205 | 2f2g |
На сайте PDB были получены PDB модель выбранной структуры, файл mmCIP и файл структурных факторов. На сайте EDS в разделе Download => Maps была получена карта электронной плотности для выбранной структуры (параметры получения использовались по умолчанию).
Разрешение для полученной структуры 2.20 Å:
REMARK 2 RESOLUTION. 2.20 ANGSTROMS.В .pdb-файле находится информация об атомах белка, каждому атому соотвествует строчка ATOM. В файле со структурными данными находится информация о всех структурных факторах, полученных в результате эксперимента. Ниже представлен фрагмент файла структурных факторов:
_refln.crystal_id _refln.wavelength_id _refln.scale_group_code _refln.index_h _refln.index_k _refln.index_l _refln.status _refln.F_meas_au _refln.F_meas_sigma_au _refln.F_calc _refln.phase_calc _refln.fomСтрочки _refln соответствуют столбцам данных, которые далее приведены в файле. Информация о каждом структурном факторе указана в отдельной строчке.1 1 1 11 1 90 f 240.8 3.2
...
Для того, чтобы оценить качество модели выбранной структуры, на нее в программе PyMOL была наложена карта электронной плотности. На рисунке 1 изображены различные уровни подрезки электронной плотности (1σ, 1.5σ и 2σ) для всей полипептидной цепи. Для того, чтобы отсечь сигнал от соседних молекул, учитывалась только электронная плотность на расстоянии не больше 2.5 Å от молекулы.

На рисунке видно, что уже при уровне подрезки 2σ для некоторых участков молекулы пропадает электронная плотность.
Далее для оценки качества модели, были получены изображения электронной плотности с различным уровнем подрезки для отдельных аминокислот.
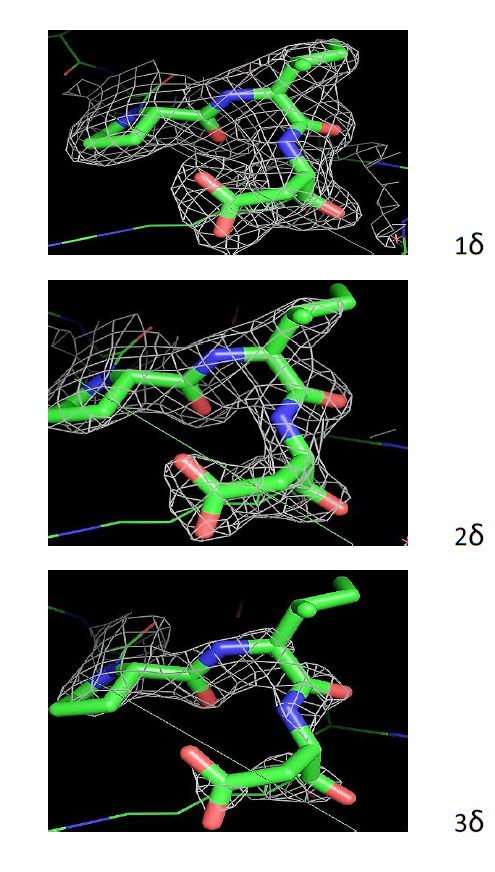
На рисунке 2 видно, что электронная плотность достаточно хорошо соответствует модели. Но если посмотреть на изображение при подрезке 3σ, можно заметить, что она слишком сильная, так как в некоторых местах у боковых радикалов аминокислот электронной плотности не наблюдается.
Чтобы, убедиться в своих предположениях, проделала теже операции с другими аминокислотами в другой цепи белка.
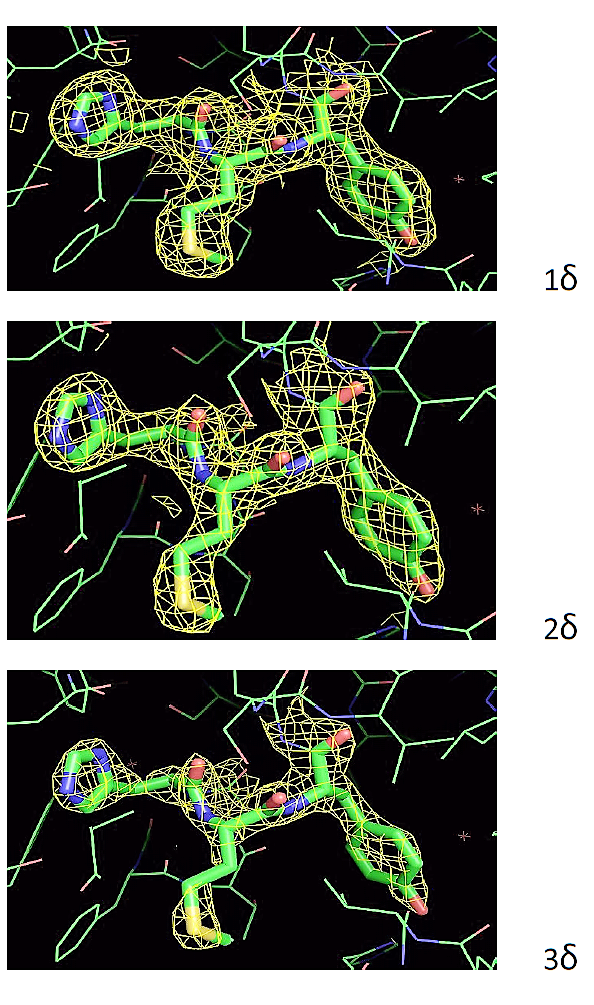
Разрешение структуры 2QCX не является достаточным для того, чтобы наблюдать отдельные атомы на изображении карты электронной плотности, однако по этой карте виден ход полипептидной цепи и уже угадывается положение боковых групп аминокислот.