Практикум 12
1. Программа сравнения выравниваний.
2. Сравнение выравниваний разными программами.
Для выравнивания были выбраны белки с полюбившейся мне в практикуме 9 мнемоникой ARAB:
ARAB_ECOLI - Escherichia coli (strain K12)
ARAB_BACSU - Bacillus subtilis (strain 168)
ARAB_SALTY - Salmonella typhimurium (strain LT2 / SGSC1412 / ATCC 700720)
ARAB_YERPE - Yersinia pestis
ARAB_ENT38 - Enterobacter sp. (strain 638)
ARAB_ECOHS - Escherichia coli O9:H4 (strain HS)
ARAB_SALG2 - Salmonella gallinarum (strain 287/91 / NCTC 13346)
Все белки имеют 3D структуры. По результатам выравнивания в практикуме 9 был сделан вывод, что все выбранные белки гомологичны.
Для проведения выравнивания был использован Jalview, программы MAFFT with Defaults, Muscle with Defaults, Tcoffee with Defaults.
Результаты
По результатам работы программы python можно сделать вывод, что выравнивания Muscle with Defaults и Tcoffee with Defaults немного более схожи, чем Muscle with Defaults и MAFFT with Defaults. В первом сравнении процент выровненных колонок для обоих выравниваний выше и на один блок больше.
| Выравнивание | Tcoffee with Defaults | MAFFT with Defaults |
|---|---|---|
| Длины | Muscle: 588 Tcoffe: 586 |
Muscle: 588 Mafft: 590 |
| Процент выровненных колонок | Muscle: 86.05442176870748 % Tcoffe: 86.3481228668942 % |
Mucle: 85.20408163265306 % Mafft: 84.91525423728814 % |
| Блоки | 7 - 42 56 - 88 91 - 158 160 - 203 209 - 225 227 - 295 297 - 321 326 - 360 391 - 392 395 - 396 402 - 403 405 - 515 521 - 561 564 - 576 |
6 - 39 58 - 89 91 - 158 160 - 203 209 - 225 227 - 295 297 - 321 326 - 360 391 - 392 402 - 403 405 - 516 521 - 561 564 - 574 |
3.Выравнивание по совмещению структур.
Для построения выравнивание 3D структур были выбраны 3 белка из случайно выбранного семейства PF03066 (Nucleoplasmin/nucleophosmin domain). Белки:
1XEO - NPM_XENLA, cтруктура и функция ядра Xenopus NO38, гистон-связывающего шаперона в ядрышке
1K5J - NUPL_XENLA, кристаллическая структура ядра нуклеоплазмина
1NLQ - NLP_DROME, кристаллическая структура ядра NLP дрозофилы, дающая представление об образовании пентамера и связывании гистонов
Структуры были загружены в PDB, где было получено выравнивание и визуализация. Также совмещение структур было выполнено в PyMol с помощью функции "align".


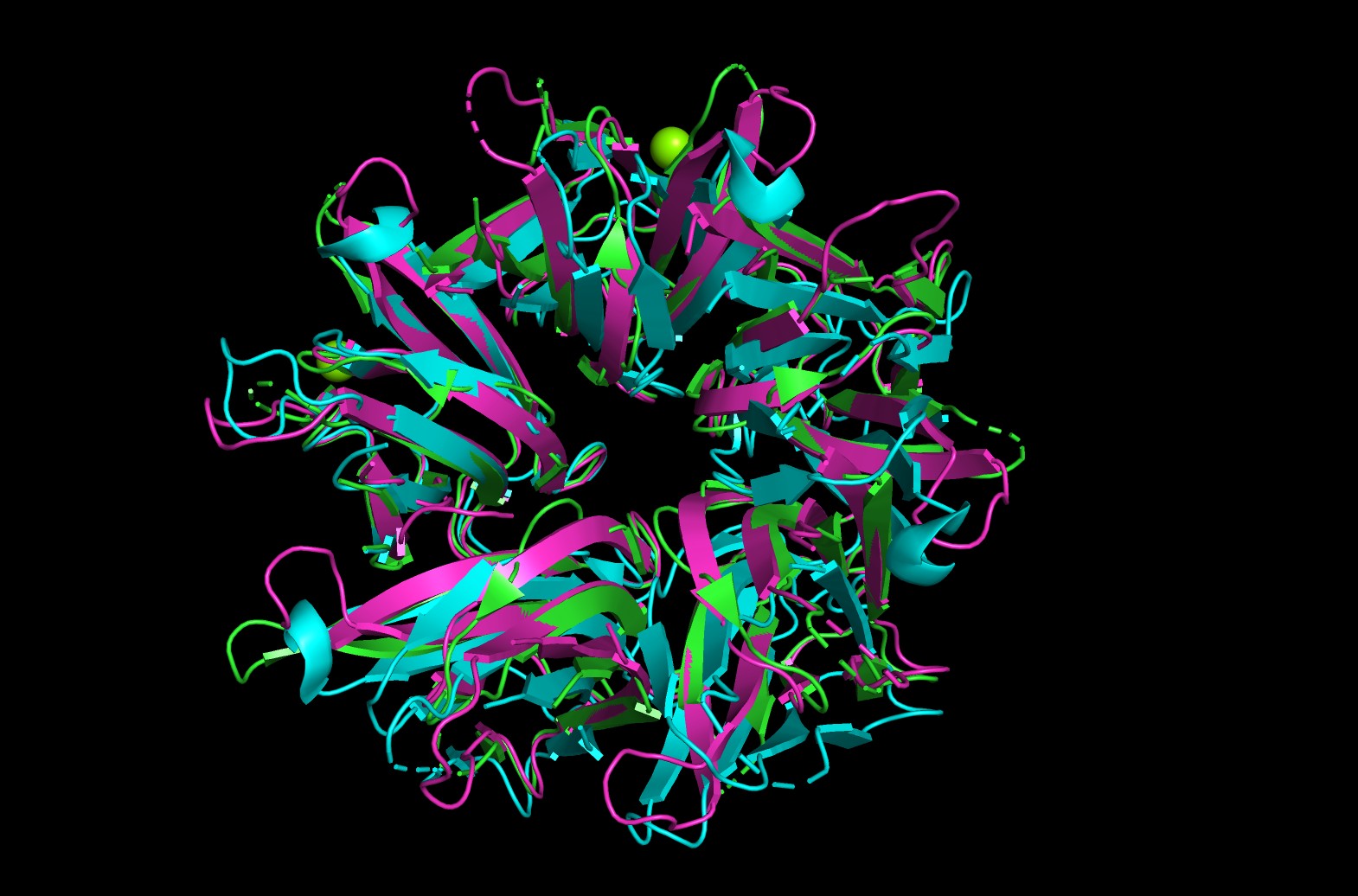



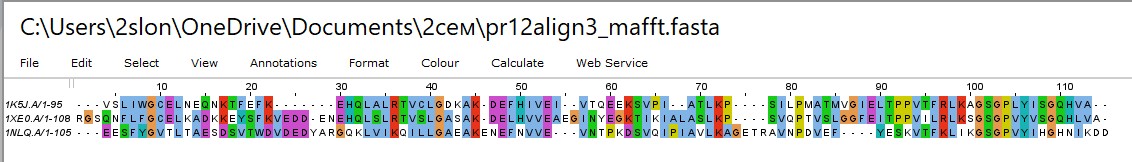

Из результатов PDB, визуализации структур и выравнивания, полученного совмещением парных выравниваний, видно, что в последовательностях есть консервативные блоки, например, если рассматривать совмещение парных выравниваний, то это блоки 40-41, 44-45, 52-53, 72-73, 99-102. Поэтому можно сделать вывод, что белки гомологичны, хотя есть некоторые сомнения.
Затем данные последовательности были выравнены с помощью программы Mafft with Defaults. Результаты сравнения выравниваний представлены ниже:
| Блоки: | 9 - 12; 17 - 18; 21 - 22; 30 - 33; 35 - 38; 40 - 41; 44 - 45; 52 - 53; 71 - 73; 92 - 94; 97 - 99; 101 - 104; 106 - 111 |
|---|---|
| Длинна выравнивания Mafft: | 115 |
| Длинна выравнивания PDB: | 115 |
| Процент выровненных коллонок 1: | 42.608695652173914 % |
| Процент выровненных коллонок 2: | 43.36283185840708 % |
Из работы программы сравнения видно, что выравнивания PBD и Mafft мало совпадают. Однако есть достаточно много блоков одинаковых коллонок.
4.Описание работы программы Mafft.
MAFFT - Multiple Alignment using Fast Fourier Transform (множественное выравнивание с использованием быстрого преобразования Фурье).
Подходит для средних и больших выравниваний.
Алгоритм работы (итеративный):
1) построение матрицы расстояний, основанной на количестве общих шестиричных кортежей
2) построение направляющего дерева (дерево родственности всех последовательностей)
• Для всех пар последовательностей строится парное выравнивание
• Вес парного выравнивания пересчитывается в расстояние между последовательностями
• Составляется матрица расстояний
• По матрице строится направляющее дерево
3) прогрессивное выравнивание (с оценкой логарифмического ожидания, выполняется от листьев к корню направляющего дерева)
4) перестройка направляющего дерева
5) перевыравнивание
MAFFT использует следующий метод для ускорения первоначального расчета матрицы расстояний:
• Аминокислотный алфавит из 20 символов сжимается до алфавита из 6 символов.
• Затем выполняется первоначальное прогрессивное выравнивание последовательностей, используя сжатый 6-символьный алфавит. Это значительно ускоряет расчет матрицы расстояний.
• После получения первоначального выравнения, MAFFT выполняет второе прогрессивное выравнивание, используя полный 20-символьный аминокислотный алфавит. Это позволяет достичь высокой точности финального выравнивания.
Сжатие алфавита до 6 символов на первом этапе ускоряет расчет матрицы расстояний, а второе прогрессивное выравнивание с полным алфавитом в конце обеспечивает высокую точность результата. Этот метод называется "быстрым деревом" и аналогичен опции быстрого дерева в ClustalW.
Таким образом, в Mafft используется 2 эвристических алгоритма выравнивания, что позволяет выравнивать последовательности большей длина с большей точность и скоростью, чем другие программы(например, T-coffee).[3]

Источники: