Практикум 14
Задание 1
С помощью команды
wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/008/SRR4240358/SRR4240358.fastq.gz
был скачан архив с чтениями проекта по секвенированию бактерии Buchnera aphidicola str. Tuc7.
Далее с помощью команды:
cat /mnt/scratch/NGS/adapters/* > adapters.fasta
был создан файл со всеми адаптерами для Illumina.
Удаление возможных остатков адаптеров:
TrimmomaticSE -phred33 SRR4240358.fastq.gz SRR4240358_trim1.fastq.gz ILLUMINACLIP:adapters.fasta:2:7:7 -trimlog trimmomatic1.log
В результате 1,66% (174955 чтений) оказались остатками адаптеров, соответственно 98,34% (10368884 чтения) "выжило".
Удаление с правых концов чтений нуклеотиды с качеством ниже 20, и удаление чтений, длина которых меньше 32 нуклеотидов:
TrimmomaticSE -phred33 SRR4240358_trim1.fastq.gz SRR4240358_trim2.fastq.gz TRAILING:20 MINLEN:32 -trimlog trimmomatic2.log
В результате 22,69% (2352447 чтений) оказались неподходящими, соответственно 77,31% (8016437 чтений) "выжило".
Изнчальный размер gz файла составлял 470М, после первого триммирования - 463М, после второго - 341М (суммарная разница 129).
Задание 2
Запуск программы velveth для коротких и не парных чтений для подготовки k-меров длиной 31:
velveth velv 31 -short -fastq.gz SRR4240358_trim2.fastq.gz
Результат запуска: директория с тремя файлами : Log, Roadmaps, (сопроводительная информация) Sequences (целевой файл)
Задание 3
Сборка k-меров с помощью velvetg:
velvetg velv
В результате в директорию добавилось 5 файлов.
N50 = 8600
С помощью конвейера:
grep -e '>' contigs.fa | tr '_' ' ' | sort -k4,4 -t ' ' -n -r | head -n 4 | less
были найдены 3 самых длинных континга и их покрытие:
>NODE 56 length 19821 cov 29.475859
>NODE 34 length 18714 cov 29.922678
>NODE 40 length 16436 cov 30.793623
Для анализа аномальных покрытий были использованы команды:
grep -e '>' contigs.fa | tr '_' ' ' | sort -k6,6 -t ' ' -n -r | less
grep -e '>' contigs.fa | tr '_' ' ' | sort -k6,6 -t ' ' -n | less
Медиана: >NODE_102_length_1717_cov_29.333721
В результате было найдено 13 аномально больших покрытий, максимальное превышает медиану в 14 раз, минимальное из аномальных практически в 5 раз. При этом длина контигов с аномально большими покрытиями варьируется от 53 до 949.
Контигов с аномально малыми покрытиями также 12. Минимальное - 1,7, максимальное аномальное - 5,75. Длина контигов варьируется от 31 до 115, т.е. вряд ли они могут являться шумом.
Задание 4
Сравнение программой megablast самых длинных контигов с хромосомой Buchnera aphidicola.
Для контига 34 было построено 6 выравниваний:
Таблица 1
| Участок | Процент идентичности | Число гэпов | Вес выравнивания |
|---|---|---|---|
| 9387-11586 | 1896/2220 (85%) | 30/2220 (1%) | 2278 |
| 15025-18744 | 2935/3781 (78%) | 144/3781 (3%) | 2163 |
| 6139-9309 | 2453/3228 (76%) | 92/3228 (2%) | 1583 |
| 1-2495 | 1982/2530 (78%) | 60/2530 (2%) | 1581 |
| 12176-14000 | 1508/1850 (82%) | 49/1850 (2%) | 1476 |
| 5505-5979 | 392/478 (82%) | 9/478 (1%) | 398 |
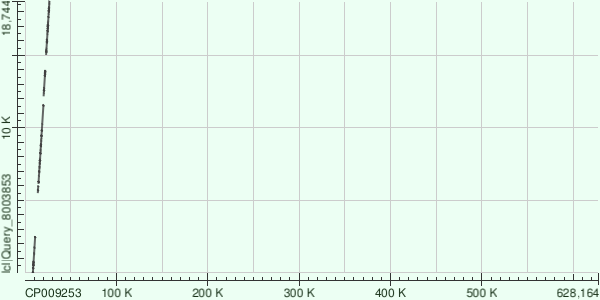
Из DotPlotа видно крупные делеции, и что контиг "ложится" на начало хромосомы.
Для континга 40 было построено 2 выравнивания:
Таблица 2
| Участок | Процент идентичности | Число гэпов | Вес выравнивания |
|---|---|---|---|
| 3-6889 | 5344/6962 (77%) | 206/6962 (2%) | 3703 |
| 6916-11860 | 3864/5019 (77%) | 164/5019(3%) | 2719 |

Делеций не видно, контиг ложится ближе к концу. Также можно заметить, что последовательность контига обратна хромосоме.
Для контига 54 было построено 3 выравнивания:
Таблица 3
| Участок | Процент идентичности | Число гэпов | Вес выравнивания |
|---|---|---|---|
| 5342-13787 | 6513/8614 (76%) | 345/8614 (4%) | 3949 |
| 15478-18851 | 3580/4396 (81%) | 83/4396 (1%) | 3520 |
| 948-5226 | 3257/4325 (75%) | 156/4325 (3%) | 1927 |
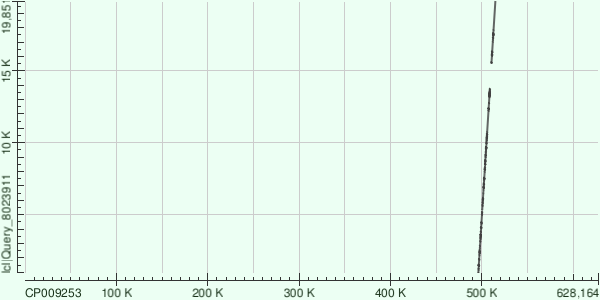
Контиг ложится ближе к концу хромосомы, чем 40 контиг. Видна 1 делеция.