Практикум 3
Задание 1
Для работы были использованы данные первого практикума. Деревья, построенные в рамках практикумов 1 и 2 считаются референсными. Для построения следующего дерева было сделано выравнивание по последовательностям малой РНК митохондриальных рибосом (12S rRNA), по которому с помощью программы fastme было построено следующее дерево (рис.1). Последовательности 12S РНК для ELIGR и MACBA не были найдены, как и для их близких родственников. Последовательности GEOTO, HETAO и APOIN были заменены на близкородственные (GEOPY, DIPMR, RAT соответсвенно) по тем же причинам.

Полученное дерево практически ничем не отличается от дерева, полученного в рамках первого практикума, разве что отсутствием некоторых видов).
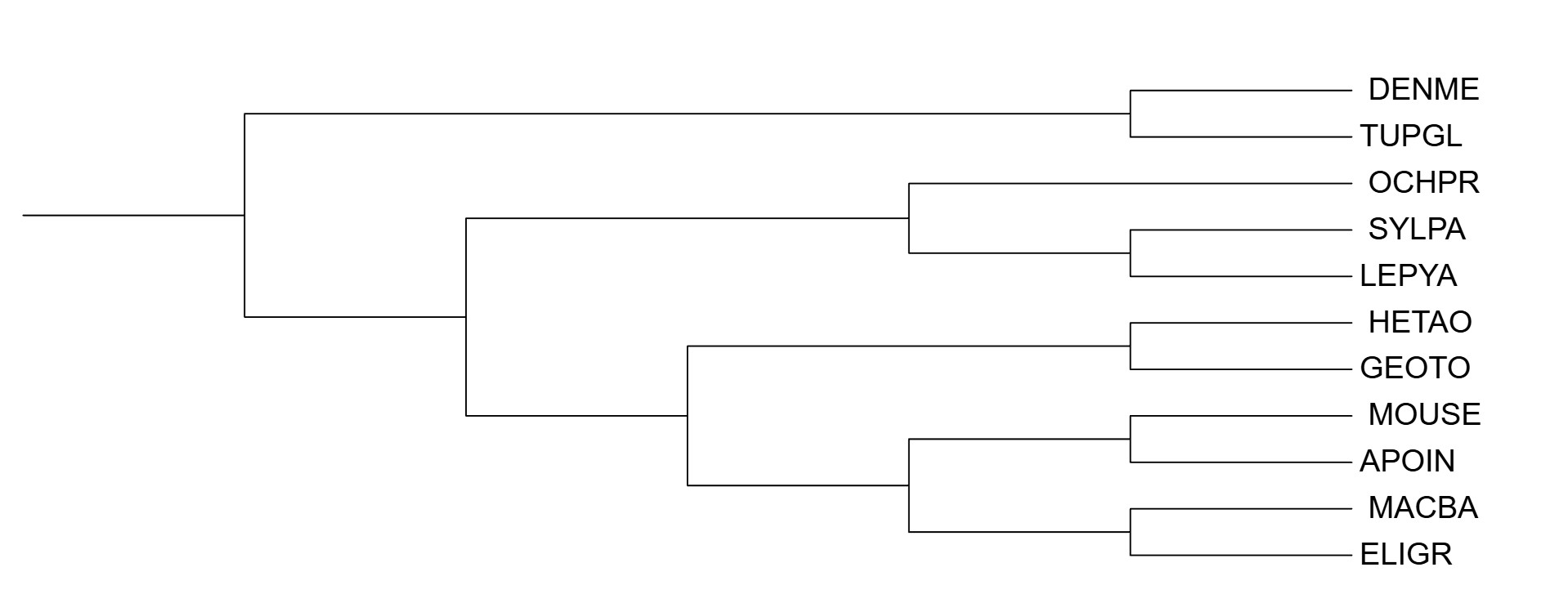
Так как дерево, построенное по rRNA, очень схоже с референcом, то и расхождения с деревьями из практикума 2 у него идентичны. Их можно увидеть по ссылке.
Задание 2
Далее было построено дерево, укорененное во внешнюю группу. Для этого был взята последовательность гена цитохрома В Ursus arctos (бурый медведь).
Для анализа дерева с укоренением во внешнюю было построено такое же дерево по последовательностям цитохрома B, но без внешней группы. При анализе можно заметить, что все клады сохранились, однако (TUPGL, DENME) и (OCHPR,(GEOTO, HETAO)) стали отделяться позднее, а (GEOTO, HETAO), наоборот, отделяется раньше.
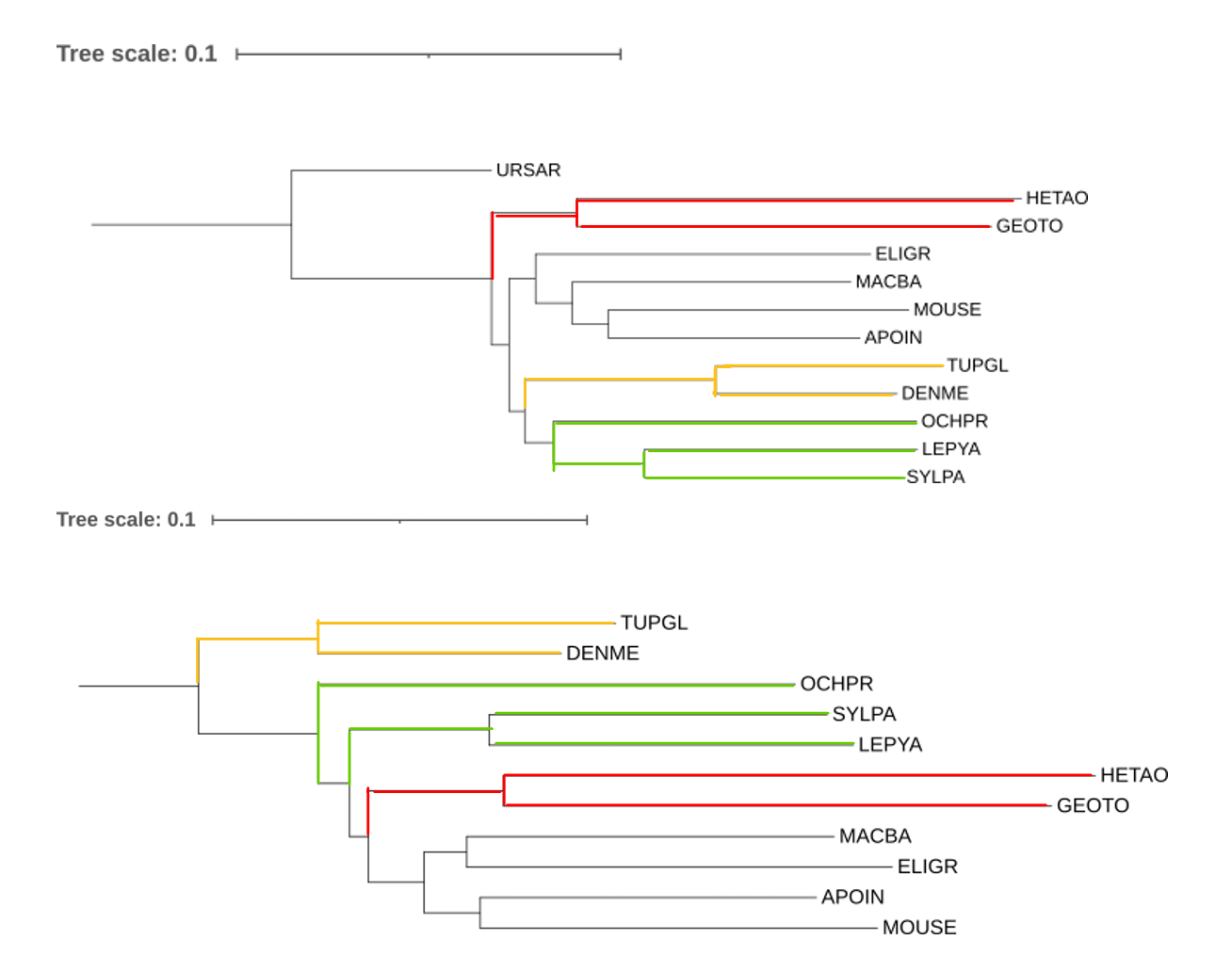
Сравнивая данное дерево с референсным (построенным по таксономии), можно заметить, что клада (TUPGL, DENME) оказалась эволюционно более поздней, в отличие от остальных деревьев из практикумов 1 и 2. Клада (GEOTO, HETAO), наоборот, переместилась ближе к корню дерева.
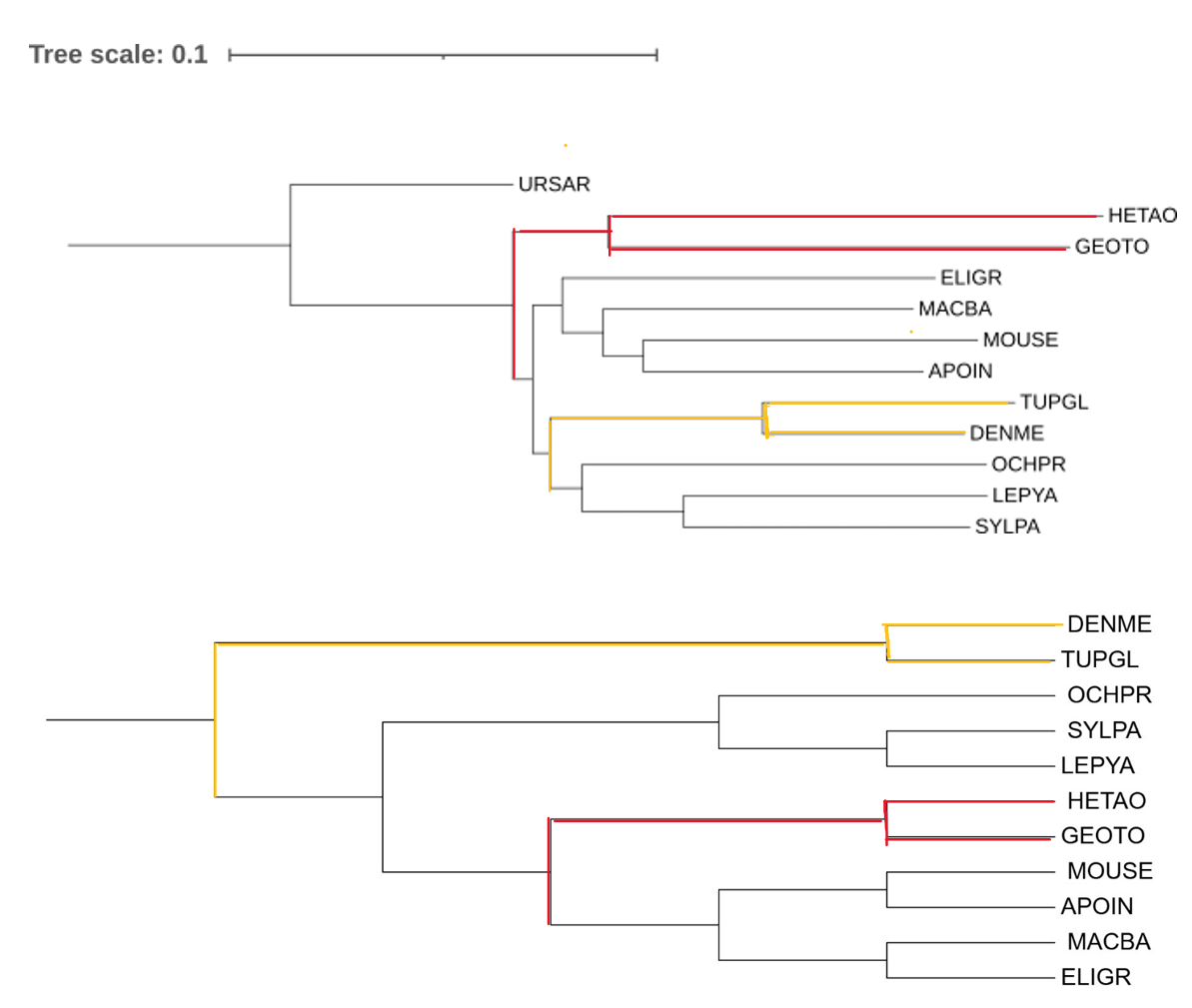
Задание 3

Ветвь, отделяющая кладу (TUPGL, DENME) от остальных, имеет наибольшую поддержку, что логично, так как на всех деревьях, кроме дерево с внешней группой, эта клада была отдельной. Так же клада (GEOTO, HETAO) имеет поддержку 100, что логично, так как ее положение изменяется только при построении дерева с iqtree. Наименьшую поддержку имеет клада ((GEOTO,HETAO),((MACBA, ELIGR), (APOIN, MOUSE))), что странно, так как она полностью повторяет кладу дерева, построенного по таксономии, хотя и имеет отличия от деревьев, построенных fastme и iqtree. Еще низкую поддержку имеет клада (MACBA, ELIGR), что не удивительно, так как при использовании разных программ положение этой клады было достаточно изменчивым.