Практикум 4
Составление списка гомологичных белков, включающих паралоги
Для выполнения задания были выбраны бактерии:
AROAE (Aromatoleum aromaticum)
BRUSU (Brucella suis biovar)
HAEIN (Haemophilus influenzae)
PASMU (Pasteurella multocida)
PSEAE (Pseudomonas aeruginosa)
ROSDO (Roseobacter denitrificans)
SHEDO (Shewanella denitrifican)
THIDA (Thiobacillus denitrificans).
Для получения списка гомологов был создан список последовательностей proteomes.fasta с помощью команды cat. Далее файл proteomes был проиндексирован с помощью команды makeblastdb с параметром -dbtype prot. После этого был запущен поиск гомологов по базе данных с помощью команды blastp с параметром -evalue 0.001.
Из содержимого файлов была создана база данных proteomes и в ней были найдены достоверные гомологи белка CLPX_ECOLI:
sp|Q12LA2|CLPX_SHEDO ATP-dependent Clp protease ATP-binding subun... 712 0.0
sp|Q9I2U0|CLPX_PSEAE ATP-dependent Clp protease ATP-binding subun... 654 0.0
sp|Q3SI99|CLPX_THIDA ATP-dependent Clp protease ATP-binding subun... 642 0.0
sp|P57981|CLPX_PASMU ATP-dependent Clp protease ATP-binding subun... 612 0.0
sp|P44838|CLPX_HAEIN ATP-dependent Clp protease ATP-binding subun... 587 0.0
sp|Q165G0|CLPX_ROSDO ATP-dependent Clp protease ATP-binding subun... 582 0.0
sp|Q9HUC5|HSLU_PSEAE ATP-dependent protease ATPase subunit HslU O... 95.9 1e-21
sp|Q12IT8|HSLU_SHEDO ATP-dependent protease ATPase subunit HslU O... 94.0 4e-21
sp|Q16CY2|HSLU_ROSDO ATP-dependent protease ATPase subunit HslU O... 93.6 6e-21
sp|P57968|HSLU_PASMU ATP-dependent protease ATPase subunit HslU O... 93.2 8e-21
sp|P43773|HSLU_HAEIN ATP-dependent protease ATPase subunit HslU O... 92.0 2e-20
tr|Q3SFW1|Q3SFW1_THIDA ATP-dependent protease ATPase subunit HslU... 86.7 1e-18
sp|Q168A2|RUVB_ROSDO Holliday junction branch migration complex s... 45.4 2e-05
tr|Q3SJR4|Q3SJR4_THIDA ATP-dependent zinc metalloprotease FtsH OS... 43.5 1e-04
Реконструкция и визуализация
Для построения дерева последовательности был использован алгоритм iqtree.
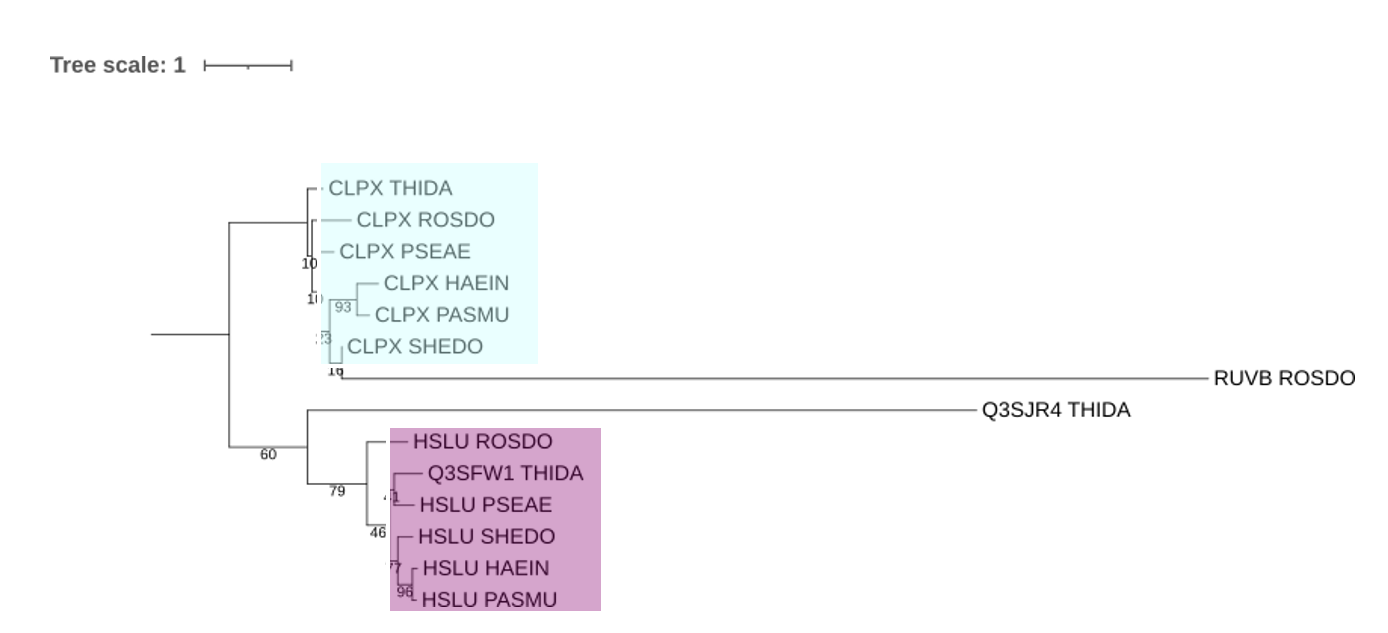
Ортологи:
Q3SFW1 THIDA и HSLU PSEAE
CLPX_HAEIN и CLPX_PASMU
HSLU HAEIN и HSLU_PASMU
Паралоги:
CLPX_THIDA и Q3SFW1_THIDA
CLPX_ROSDO и RUVB_ROSDO
CLPX_HAEIN и HSLU_HAEIN
Описание ортологических групп
В состав ортологической группы, покрашенной голубым на рисунке 1 входят белки CLPX из бактерий SHEDO, PSEAE, THIDA, PASMU, HAEIN, ROSDO.
Этот белок представляет собой АТФ-зависимую субъединицу протеазы Clp, связывающую АТФ. АТФ-зависимая субъединица протеазы Clp является частью системы протеазы Clp, которая играет важную роль в деградации белков в клетках. Система Clp состоит из нескольких субъединиц, включая АТФ-связывающие субъединицы (ClpX, ClpC) и протеолитические субъединицы (ClpP). АТФ-связывающие субъединицы отвечают за распознавание и связывание целевых белков, а также за обеспечение энергии для их деградации посредством гидролиза АТФ.
При сравнении с таксономическим деревом видно, что клада (SHEDO, (HAEIN, PASMU)) построена верно, а бактерии THIDA, PSEAE, ROSDO не являются близкородственными.
В состав ортологической группы, покрашенной розовым на рисунке 1 входят белки HSLU из бактерий PSEAE, SHEDO, ROSDO, PASMU, HAEIN и белок Q3SFW1 из бактерии THIDA.
HslU является АТФазной субъединицей бактериальной протеазы HslUV, которая представляет собой АТФ-зависимый протеолитический комплекс. Этот комплекс состоит из двух основных компонентов: HslU, который является АТФазной субъединицей, и HslV, являющейся протеолитической субъединицей. Функции белка: распознавание и транслокация субстратов, активация протеазы, структурная организация. Белок Q3SFW1 также является АТФазной субъединицей бактериальной протеазы HslU.
При сравнении с таксономическим деревом видно, что клада (SHEDO, (HAEIN, PASMU)) построена верно, ROSDO также отделена от остальных бактерий, а клада (THIDA, PSEAE) на таксономическом дереве отсутствует и эти бактерии не являются близкородственными.