Практикум 8
Описание сигнала
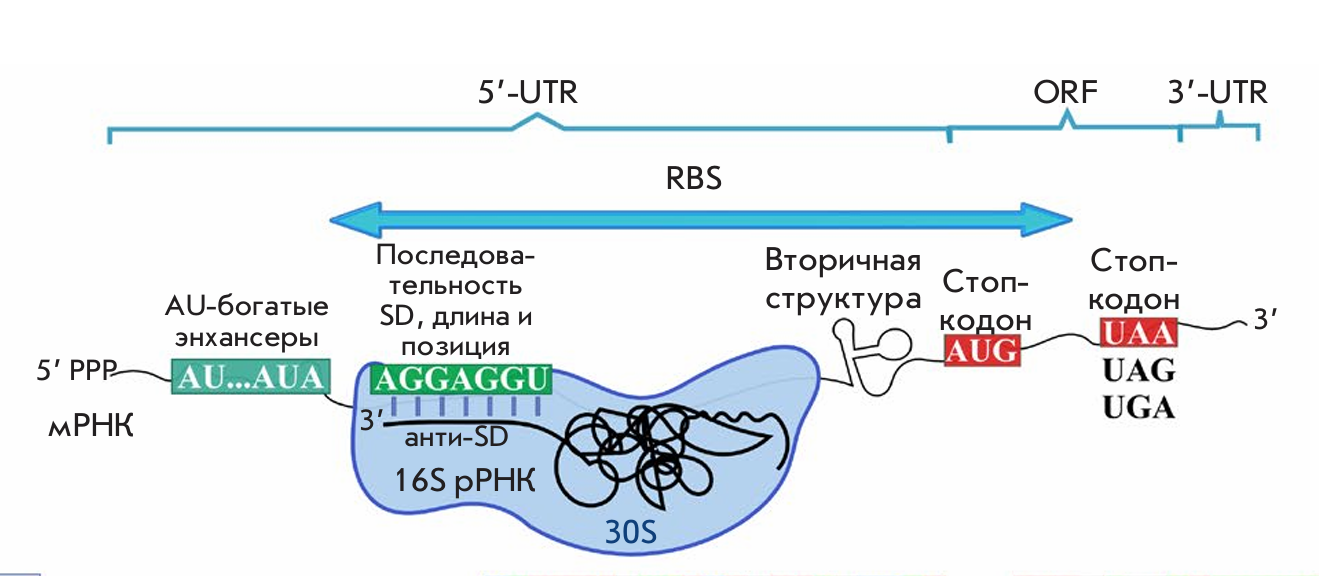
Название сигнала: Последовательность Шайна–Дальгарно (SD) — это консервативный регуляторный элемент в мРНК бактерий и архей, обеспечивающий связывание рибосомы с матрицей.
Носитель сигнала: Молекула мРНК прокариот. Сигнал представлен короткой нуклеотидной последовательностью, расположенной в 5'-нетранслируемой области перед стартовым кодоном AUG.
Кому адресован: Малая субъединица рибосомы (30S у бактерий). Анти-SD-последовательность на 3'-конце 16S рРНК рибосомы комплементарна SD-сайту.
Предназначение: Обеспечить точное связывание рибосомы с мРНК и позиционирование стартового кодона в P-сайте. Это необходимо для инициации трансляции, регуляции уровня экспрессии генов (сила взаимодействия SD-анти-SD влияет на эффективность трансляции), независимой инициации трансляции в полицистронных мРНК.
Сила сигнала определяется:
Степенью комплементарности с анти-SD-последовательностью 16S рРНК;
Расстоянием до стартового кодона (5–8 нуклеотидов);
Отсутствием вторичных структур в 5'-UTR, которые могут блокировать доступ к SD-сайту.
Мутации в SD-сайте снижают трансляцию в 10–100 раз.
Из вышеперечисленных данных можно сделать вывод, что сигнал обладает высокой эффективностью.
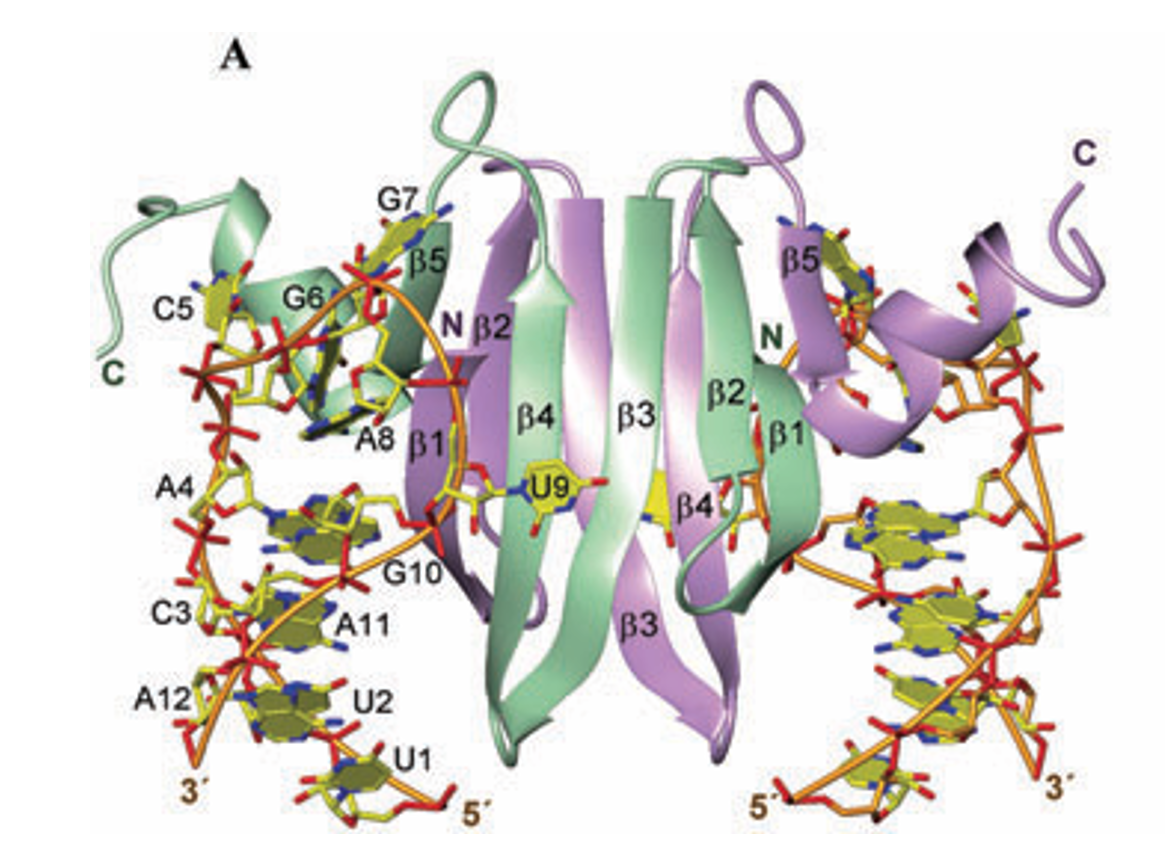
С эффективностью также связаны белки RsmA/CsrA (и их гомологи, такие как RsmE). Они участвуют в регуляции трансляции через связывание с малими РНК (sRNA), которые имеют повторяющиеся GGA мотивы. Эти мотивы часто локализуются в структурах петли или между стеблями в структуре stem-loop мРНК.
Такие мотивы GGA в 5' нетранслируемом участке мРНК, например у hcnA гена, перекрываются с SD последовательностью. Это препятствует связыванию рибосомы с мРНК и ингибирует начало трансляции. Таким образом, белки RsmA и RsmE блокируют доступ рибосомы к SD-последовательности. Это снижает или полностью блокирует инициацию трансляции на этой мРНК.
Малая РНК (например, RsmY) с многочисленными GGA мотивами может освобождать мРНК от этого регуляторного блока, улучшая доступ рибосомы к SD-последовательности, тем самым снимая репрессии и способствуя трансляции.
Примеры сигнала:
1. В геноме E. coli:
Каноническая последовательность — AGGAGGU
2. В экспрессионных векторах используется SD-сайт гена 10 бактериофага T7 для усиления трансляции.
3. У цианобактерии Nostoc sp.: SD-мотив GGAGG с вариациями в позициях.
4. В полицистронных мРНК: перед каждым ORF расположены отдельные SD-последовательности (например, GGAGGA перед ORF1, ORF2 и ORF3).
Ссылки на литературу:
Сервисы для поиска выбранного сигнала
Как таковых сервисов для поиска последовательностей Шайна-Далгарно к сожалению найти не удалось :(
1) ProPr: Prokaryote Promoter Prediction v2.0
Веб-сервер для предсказания промоторных элементов и регулонов прокариот
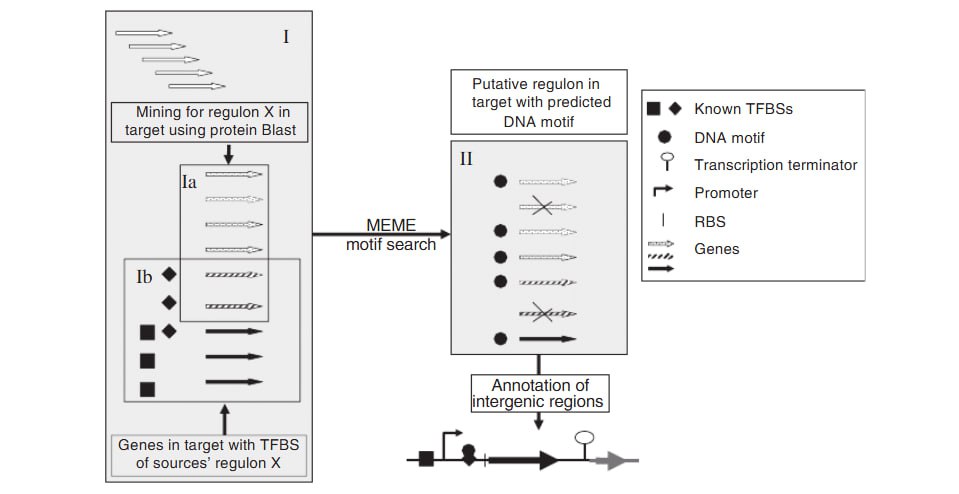
Алгоритм: Первым шагом PePPER all-in-one является отбор набора генов, которые предположительно принадлежат к определенному регулятору в одном организме (целевом; в рамке I представлены все гены целевого организма), путем сравнения с соответствующими регуляторами во всех других организмах (исходных) с помощью белкового Blast (гены в рамке Ia). Параллельно с этим известные TFBS этих регулонов используются для поиска генов в целевом организме, которые несут этот мотив ДНК в своих апстрим регионах (гены во врезке Ib). Затем проводится MEME-поиск в регионах верхнего течения генов в обоих независимо полученных пулах генов. В результате получается набор генов, представляющий предполагаемый регулон в целевом организме с его предсказанными TFBS (гены в блоке II). Наконец, добавляются такие характеристики, как RBSs, промотор и терминаторы транскрипции, после чего результат представляется в графическом виде. Информация может быть доступна и просмотрена отдельно по каждому гену.
В выдаче получаем таблицы с последовательностями промоторов, терминаторов и их координат, а также номера хромосом
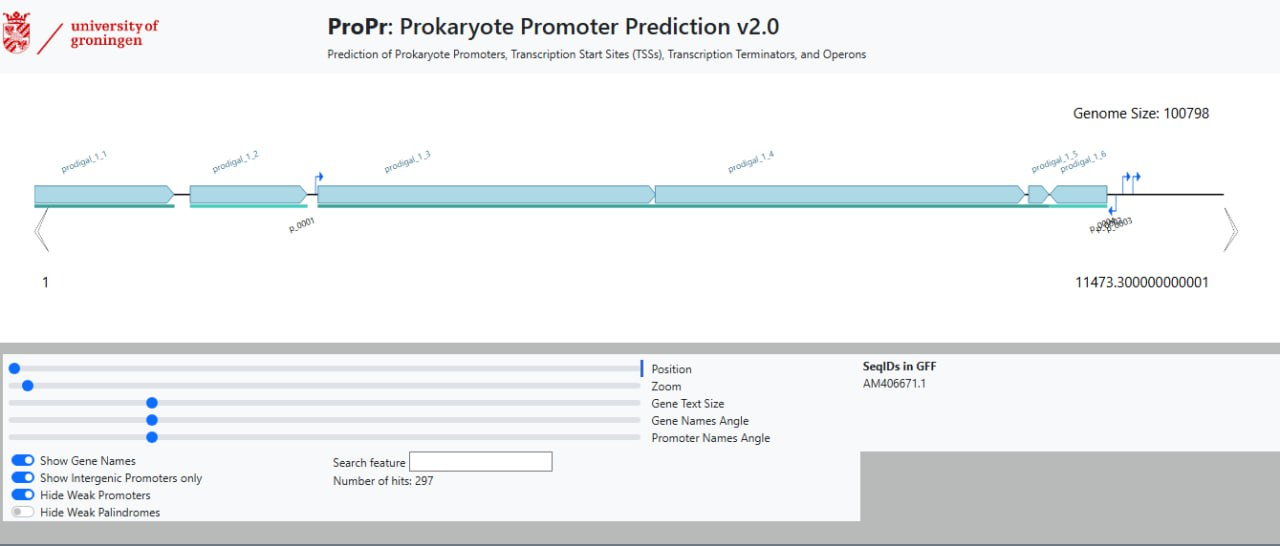
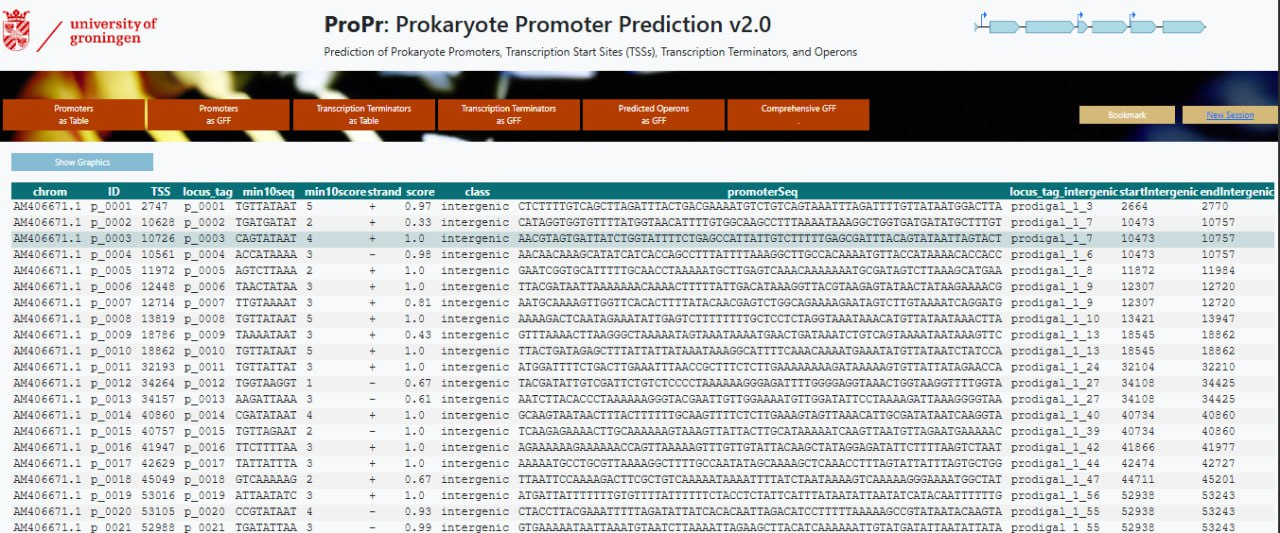
Однако, этот сервис ищет именно промоторные последовательности все какие есть, а не целенаправленно последовательность Шайна-Дальгарно.
2) uorf4u
Биоинформационный инструмент для аннотации консервативных up-stream ORF.
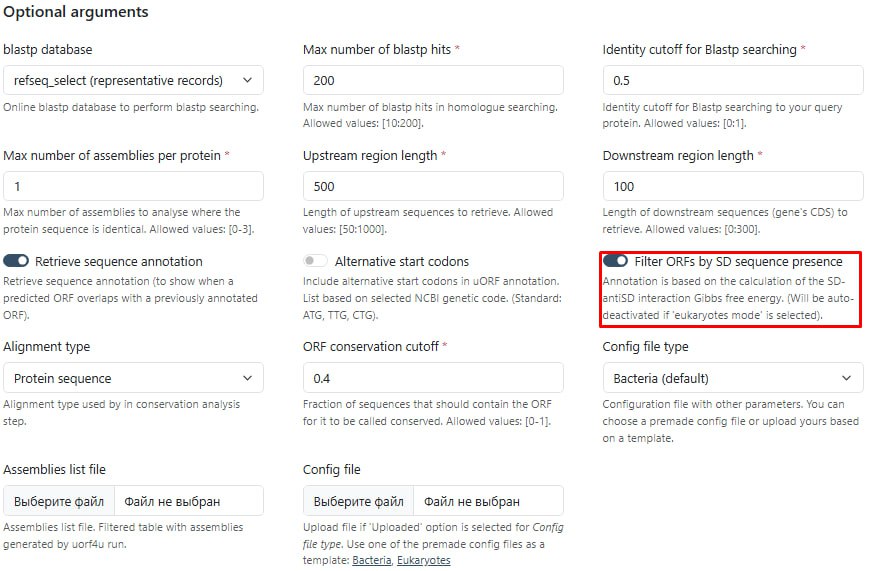
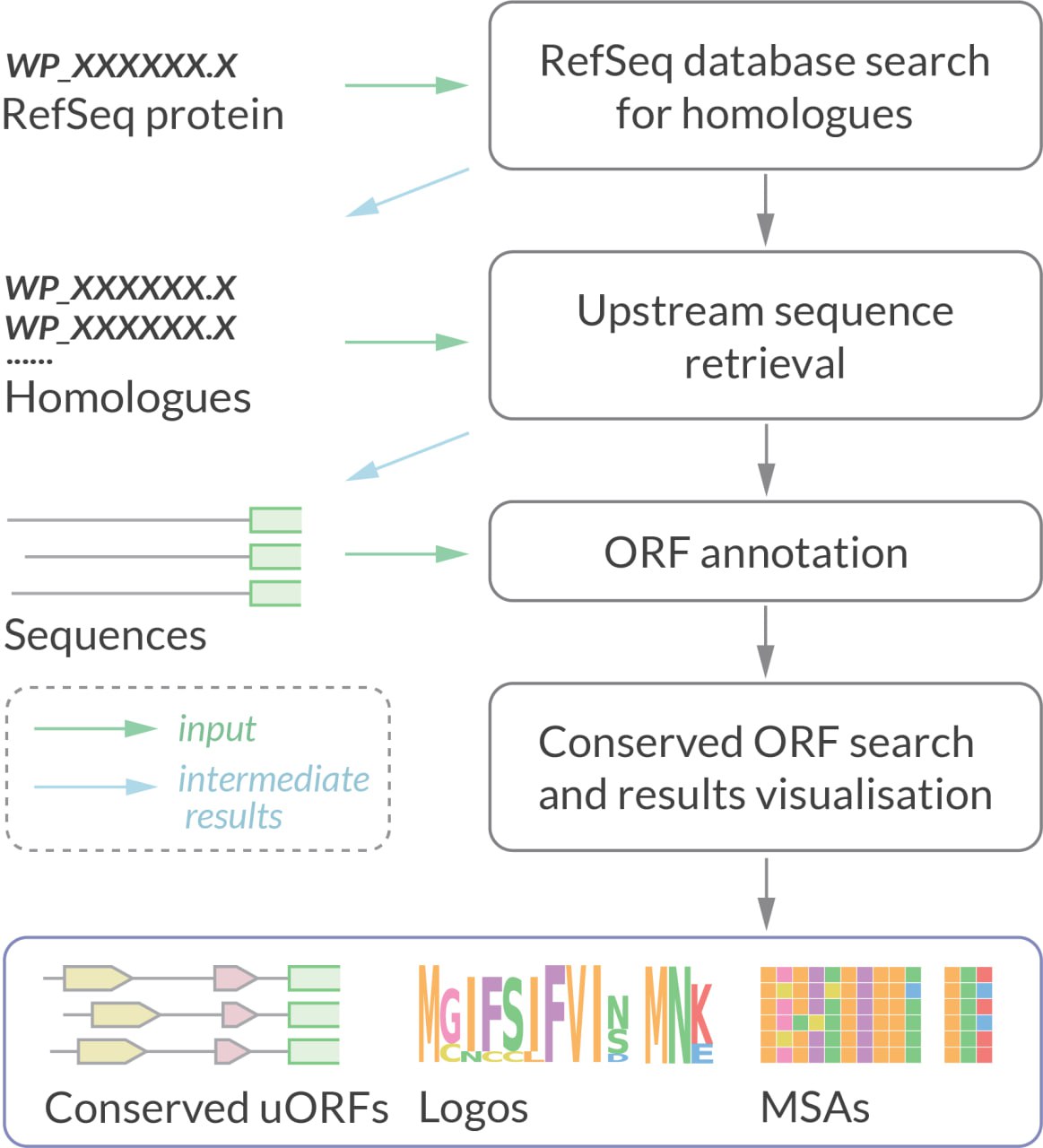
Алгоритм: Самый простой входной файл - это заданный пользователем номер доступа к белку в NCBI RefSeq, но также это может быть список гомологов (список номеров доступа RefSeq) или файл последовательностей в формате FASTA. В зависимости от исходных данных uORF4u состоит из нескольких этапов: поиск гомологов по базе данных RefSeq с помощью BlastP [запись промежуточных результатов (синие стрелки) в выходной файл], поиск последовательности выше по течению с записью результатов в выходной файл FASTA, аннотация ORF, и, наконец, анализ сохранности и визуализация результатов. ORF определяется как область между стартовым кодоном (могут быть включены и альтернативные стартовые кодоны) и стоп-кодоном, расположенным down-stream. Минимальная длина, установленная по умолчанию, составляет девять нуклеотидов (три кодона). Для прокариот этот шаг также включает поиск последовательности Шайна-Дальгарно (SD) в 20-нуклеотидном окне перед стартовым кодоном. Аннотация SD-последовательности основана на расчете свободной энергии Гиббса SD-антиSD взаимодействия.
Опять же она аннотирует, но не ищет послдовательность Шайна-Дальгарно.

3) FIMO (Find Individual Motif Occurrences)
FIMO сканирует набор последовательностей в поисках индивидуальных совпадений с каждым из предоставленных вами мотивов.
Алгоритм: Программа ищет в наборе последовательностей вхождения известных мотивов, рассматривая каждый мотив независимо. Мотивы должны быть в формате MEME Motif Format. Веб-версия FIMO также позволяет вводить мотивы в дополнительных форматах.
Авторы отчета:
Аристов Иван
Джолос Ксения
Чистякова Екатерина