8-916-939-49-78
jiabicht@rambler.ru
vk.com/allweiss
Программа getorf извлекает из нуклеотидной последовательности открытые рамки считывания. С помощью команды entret была получена запись D89965 из банка EMBL. Для получения интересующих нас рамок считывания getorf использовался со следующими параметрами:
getorf -find 1 -maxsize 90 -outfile D89965.orf D89965.entret
Было обнаружено 5 последовательностей, причем одна из них - D89965_3 - частично соответствует приведённой в поле FT кодирующей последовательности (CDS): координата последнего нк в найденной с помощью программы рамки меньше, чем у исходной на 3, т. к. в CDS рисутствует стоп-кодон.
Запись D89965 ссылается на последовательность hslv_ecoli.Для того, чтобы узнать, какой рамке соответствует эта последовательности, применили программу blastp:
blastp -query hslv_ecoli.fasta -subject d89965.orf -out blastp.out
Получили, что пятая рамка D89965_5, соответствующая обратной цепи, лучше всех выравнивается с hslv_ecoli
Query= HSLV_ECOLI P0A7B8 ATP-dependent protease subunit HslV (3.4.25.2)
(Heat shock protein HslV)
Length=176
Subject= D89965_5 [294 - 1] (REVERSE SENSE) Rattus norvegicus mRNA for RSS,
complete cds.
Length=98
Score = 200 bits (509), Expect = 4e-71, Method: Compositional matrix adjust.
Identities = 98/98 (100%), Positives = 98/98 (100%), Gaps = 0/98 (0%)
Query 28 MKGNVKKVRRLYNDKVIAGFAGGTADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDR 87
MKGNVKKVRRLYNDKVIAGFAGGTADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDR
Sbjct 1 MKGNVKKVRRLYNDKVIAGFAGGTADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDR 60
Query 88 MLRKLEALLAVADETASLIITGNGDVVQPENDLIAIGS 125
MLRKLEALLAVADETASLIITGNGDVVQPENDLIAIGS
Sbjct 61 MLRKLEALLAVADETASLIITGNGDVVQPENDLIAIGS 98
Чтобы получить искомы дегидрогеназы использовалась команда:
seqret sw:adh*_* adh.fasta
В итоге были получены все доступные в Swissprot последовательности алкогольдегидрогеназ. Для того, чтобы получить файл с универсальными адресами (USA) этих последовательностей использовалась программа infoseq -only и -usa:
infoseq adh.fasta -only -usa > list.txt
Из этого файла-списка был создан меньший файл, с адресами только тех последовательностей, которые взяты из некоторых организмов. Для этого использовалась команда grep с опцией -f:
grep -f pattern.txt list.txt > list2.txt
Fasta-файл с последовательностями дегидрогеназ моих организмов был получен с помощью команды seqret:
seqret @list2.txt adh_n.fasta
Файлы, полученные в этом задании, можно посмотреть здесь:adh.fasta,list.txt, list2.txt,adh_n.fasta
Идентификатор белка по online BLAST: ANGE1. Последовательность гена: angel1.fasta. Для начала искали ген в человеческом геноме сервисом "BLAST/BLAT".
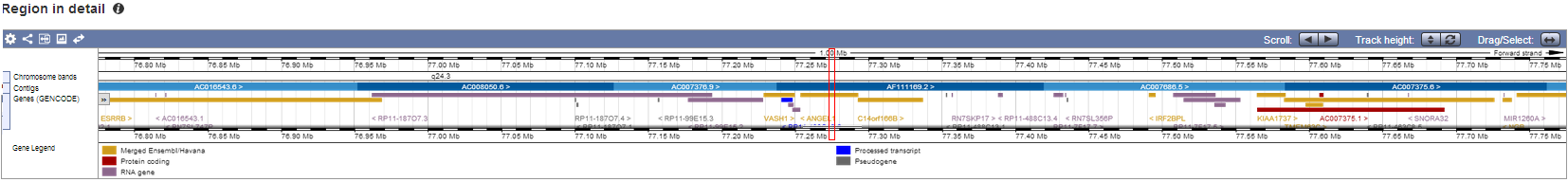
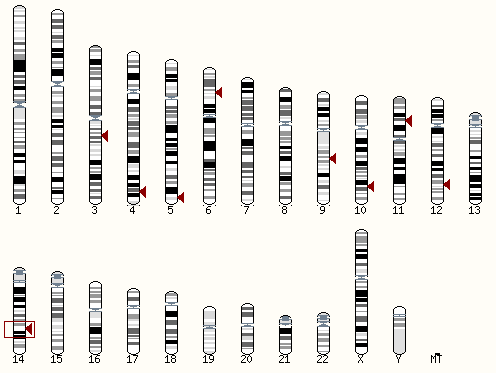
В блоке "Alignment Locations vs. Karyotype" можно увидеть расположение участка генома, который выравнялся с данным геном. Наш ген находится в большом плече 14 хромосомы, это видно потому, что участок выделен. Все отстальное - артефактные выравнивания.
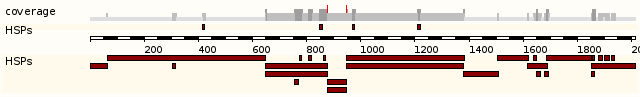
В следующем разделе "Alignment Locations vs. Query" информация о выравнивании приведена в графическом виде:
В последнем блоке "Alignment Summary" дана информация о последовательности, позиции на гене хромосоме, score, e-value, проценте идентичности, длине выравнивания, а так же ссылки (Links).
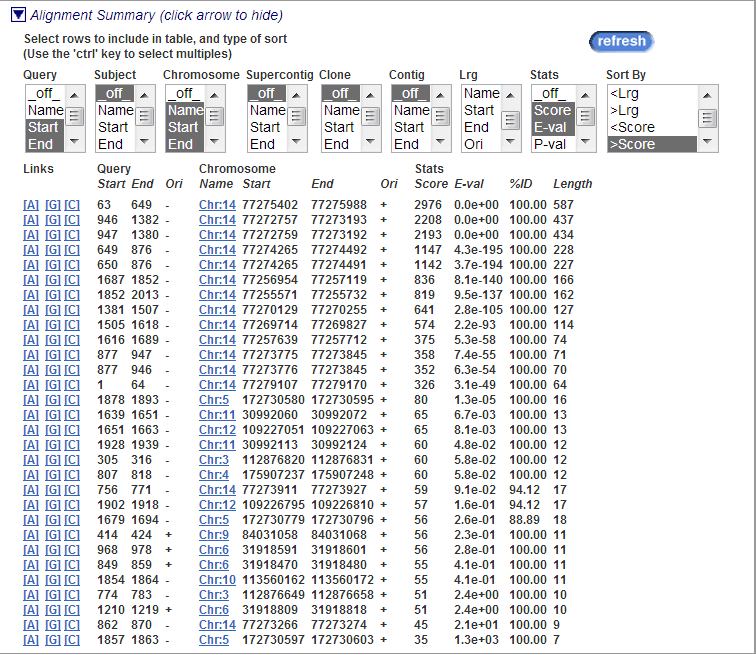
В "Links" есть 3 ссылки: [A] - Alignment, [G] - Genome Sequence, [C] - ContigView.
Сылка [A] приводит к подробному описанию выравнивания:

Сылка [G] показывает последовательность хромосомы. Красным цветом отмечено выбранное выравнивание, желтым - экзоны (здесь нет), синим - другие выравнивания (здесь нет):

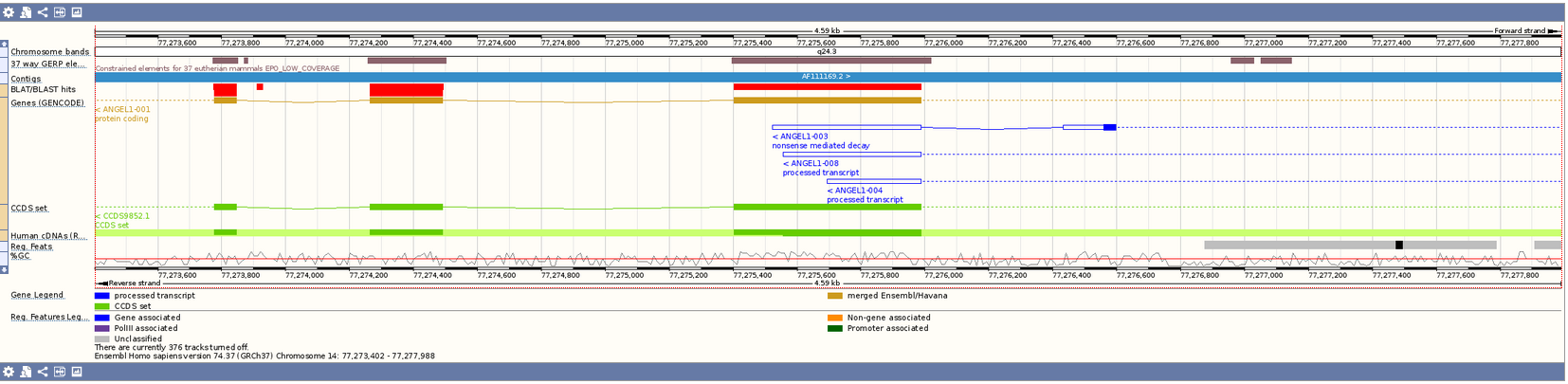
Ссылка [C] дает информацию о расположенных на участке хромосомы генах, о экзонах и интронах. В общем, есть 3 раздела: