8-916-939-49-78
jiabicht@rambler.ru
vk.com/allweiss
Для работы был выбран домен Integrin_alpha2 (Pfam: PF08441). Этот домен часто входит в состав белков, участвующих в клеточной адгезии либо во взаимодействии клеток с внеклеточным матриксом. Также домен входит в суперсемейство IG-like доменов, третичная структура которых похожа на иммуноглобулин.
Выбранный домен входит в состав 51 архитектуры. В 29 архитектурах из 51 помимо Integrin_alpha2 присутствует домен FG_GAP (часто в повторах). Ещё один домен, часто встречающийся в архитектурах вместе с выбранным доменом (17 из 51) - это VWA (von Willebrand factor type A domain). VWA домены в интегринах опосредуют адгезию, контролируемую ионами металлов. FG-GAP мотивы часто присутствуют на N-концах интегринов, формируя структуру бета-пропеллера. Их предполагаемая функция - связь с ионами кальция.
Было выбрано 3 архитектуры:



Сравнение будет проводиться внутри таксона Metazoa (царство животных). В этом таксоне были выбраны подтаксоны Chordata и Arthropoda.
Была получена таблица, где для каждого домена (первая строка с PFAM ID) была показана встречаемость в конкретной последовательности (AC белков в первом столбце) с указанием длины домена в скобках. Далее был осуществлён поиск последовательностей в Uniprot по их AC. На вход было подано 1448 AC, однако активных записей оказалось меньше - 1304. Остальные записи были удалены. 1304 записей были скачаны из Uniprot в формате Text (файл) и поданы на вход скрипту uniprot_to_taxonomy.py. Команда
python swisspfam-to-xls.py -z /srv/databases/pfam/swissprot.gz -p PF08441 -o pfam.xls
Была получена таблица с полными названиями организмов, к которым относятся Uniprot AC. Далее с помощью функции VLOOKUP в OpenOffice столбец с таксонами был совмещён со столбцом Uniprot AC и Pfam ID на первом листе. Финальныый вариант таблицы можно скачать здесь.
python uniprot_to_taxonomy.py -i seqtext -o taxonomy
Получение полного выравнивания для домена и формирование выборки для архитектур
Выравнивание всех последовательностей, содержащих домен (1499) было получено в Jalview путём импорта из PFAM (Fetch sequences -> PFAM -> вводится PFAM ID домена). Файл с проектом - выравнивание в Jalview. Раскрашивание было сделано по аннотации вторичной структуры, поскольку последовательности слишком плохо выравниваются между собой (структурное сходство есть, по последовательности - не очень) и раскраска ClustalX ничего не давала. Для двух интегринов человека - ITA2B и ITAV - с выравниванием были проассоциированы 3D структуры из банка PDB. Также был получен файл с выравниванием в формате .fasta.
С сайта PFAM были вручную получены последовательности доменов из разных организмов (скачать). Выборка была сделана среди доменов интегрина из 3х выбранных архитектур. Все последовательности относятся к одному из двух ранее выбранных таксонов (Arthropoda и Chordata). Они были закодированы идентификаторами вида NumberTaxon_organism. 1, 2, 3 соответствуют номерам архитектур в списке из первого задания, таксоны закодированы буквами A - Arthropoda и C-Chordata. Для 1 и 3 архитектуры были найдены последовательности из обоих таксонов, для 2 архитектуры - только для Chordata. Для второй архитектуры была взята 1 последовательность домена беспозвоночного (не Arthropoda, так как такая структура не представлена у Arthropoda), на дереве она обозначена как 2N_seasqirt. Далее полученные последовательности были выровнены при помощи MUSCLE (скачать выравнивание ). Затем в MEGA было построено дерево методом Maximum Likehood. На рисунке 1 представлен проект Jalview с выровненными доменами, который также можно скачать здесь. Дерево в Newick-формате можно скачать по ссылке.
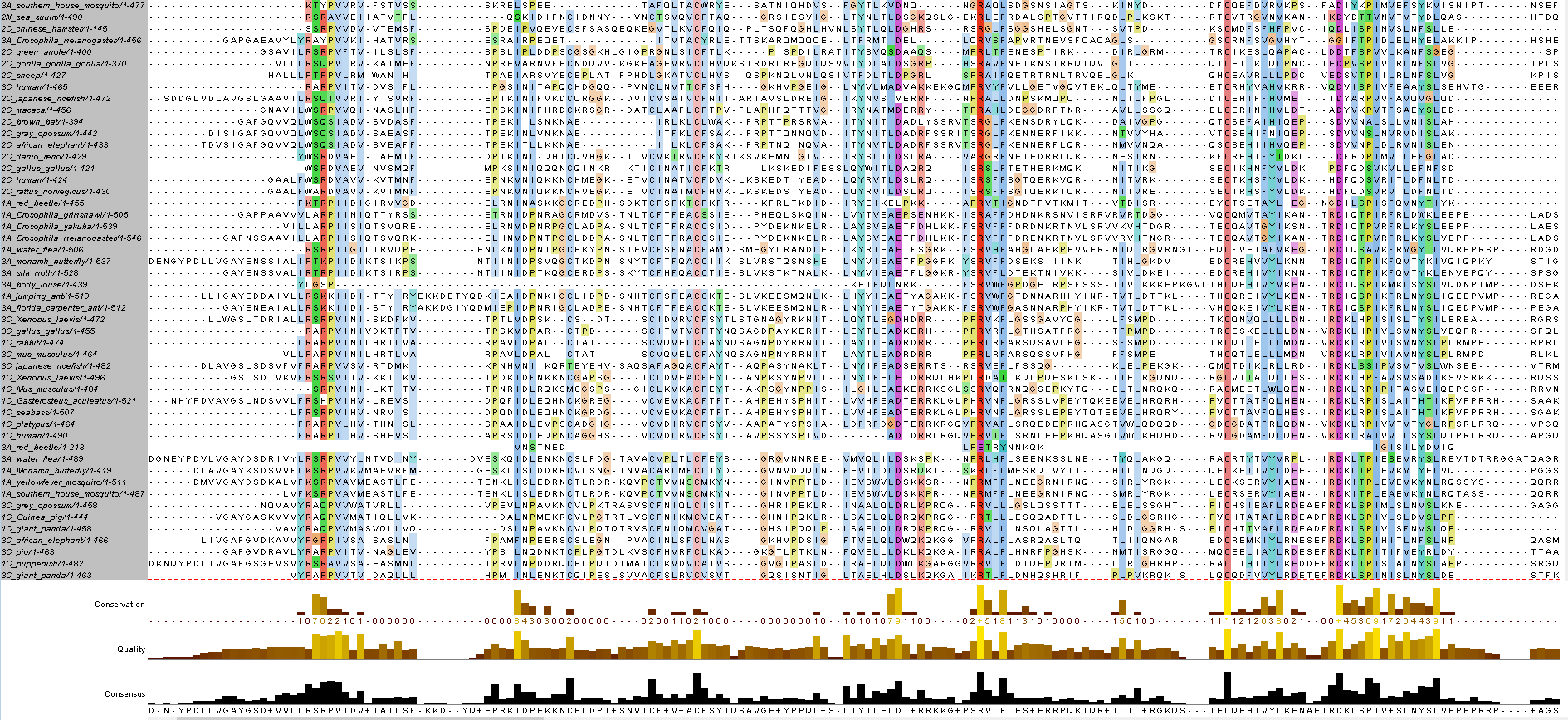
Рис.1. Общий вид проекта Jalview с раскраской ClustalX по консервативности (порог - 10%)
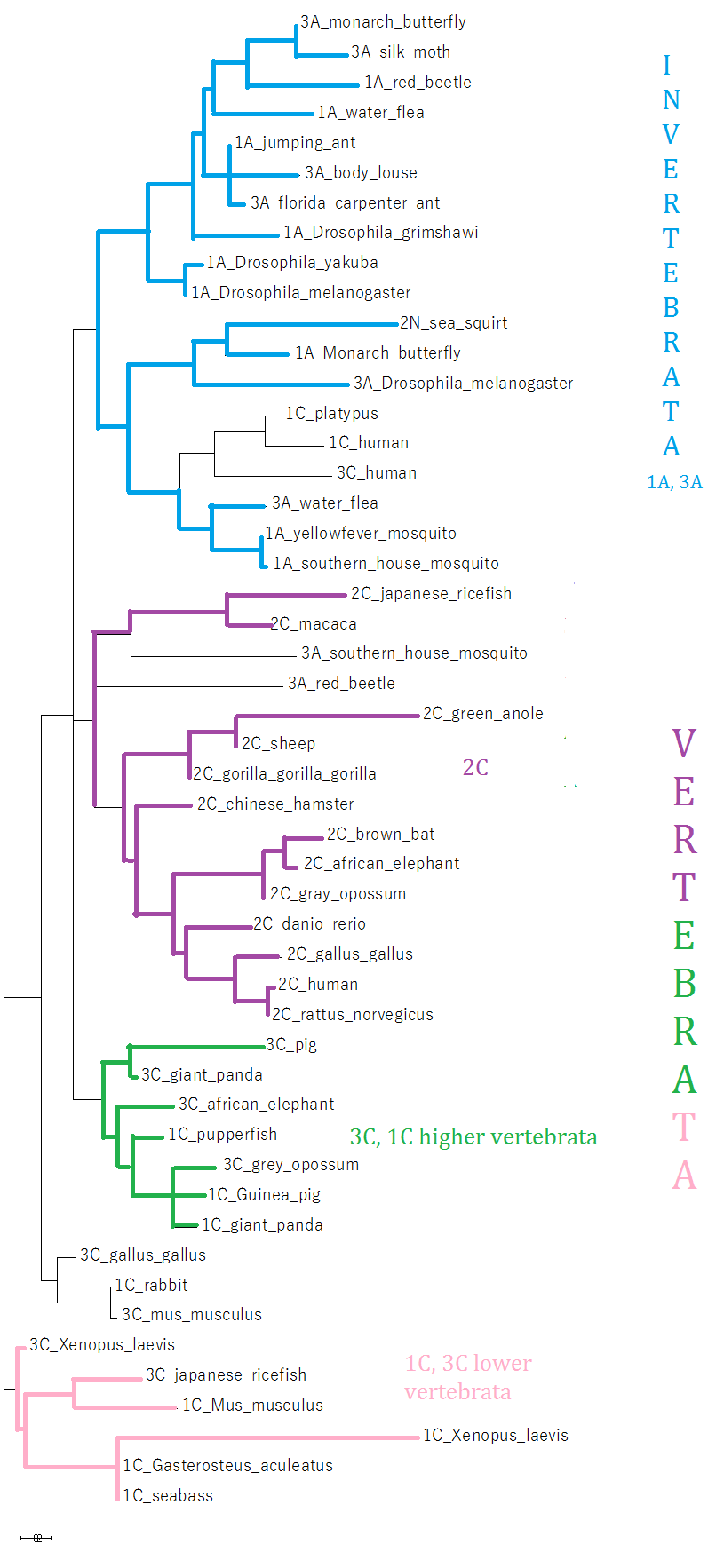
Рис.2. Дерево, построенное по полученному выравниванию.
На дереве чётко выделяется клада, включающая последовательности доменов из одинаковых архитектур. Эта клада показана фиолетовым. Для остальных двух архитектур не наблюдается группировки по архитектурам, но есть выделение общепризнанных таксонов (Invertebrata, Vertebrata). Также можно выделить группу низших и высших эукариот, хотя в этих кладах на дереве есть очевидно не подходящие организмы.
Для дальнейшего анализа возьмём группу доменов из второй архитектуры. Файл с выравниваниями выбранных последовательностей можно скачать здесь. Теперь с помощью пакета HMMER на kodomo построим скрытую марковскую модель с помощью программ:
hmm2build hmm.out 2c_alignment.fasta hmm2calibrate hmm.out hmm2search hmm.out all_seq.fasta > hmm2search
В файле hmm2search находятся результаты поиска по всем последовательностям, содержащим этот домен, которые входят в выбранное семейство. С помощью скрипта Дмитрия Пензара построим ROC-кривую. Его скрипту на вход подаём результат работы hmm2search, файл с информацией о доменной архитектуре и таксономической принадлежности, файл, с критериями отношения к нашему семейству. Заведомо положительными мы считаем результаты с выбранной архитекрутой (2с) и таксономией. Данная архитектура включает в себя 4 домена (PF08441, PF00092, PF01839, PF00357). ROC-кривая показана на рисунках 3 и 4.
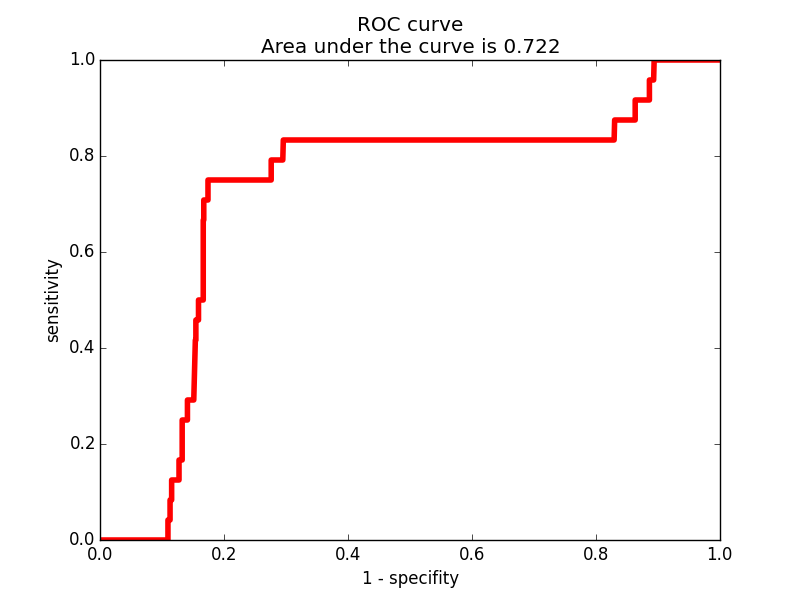
Рис.3. ROC-кривая 1
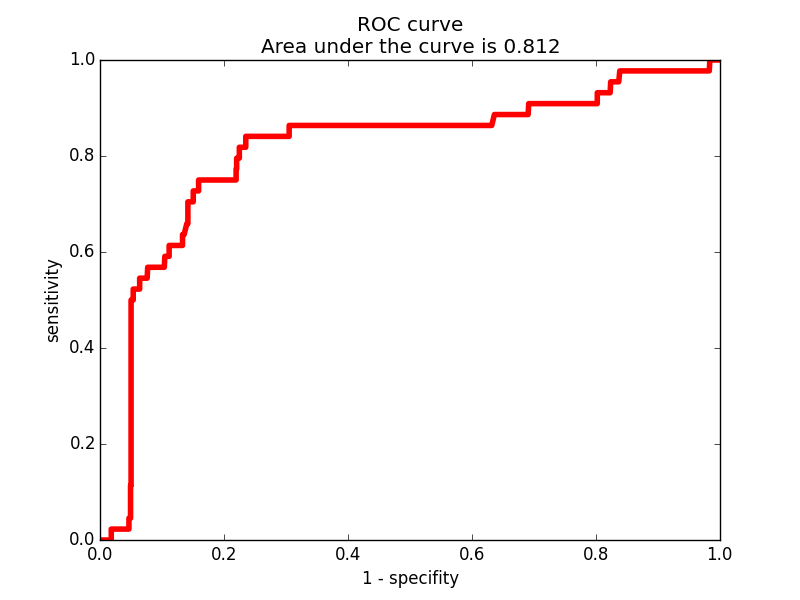
Рис.4. ROC-кривая 2