Учебный сайт Ксении Худяковой
Задание 1.
В программе BLAST был проведен поиск гомологов белка YP_004071041.1 (цистеиновая десульфираза археи Thermococcus barophilus MP). Сначала был проведён поиск в БД RefSeq, но нашлось больше 20 000 последовательностей с очень низким e-value, что не поддаётся анализу. Поэтому второй поиск был проведён в БД Swissprot.
Ссылка на поисковый запрос в БД RefSeq
Ссылка на поисковый запрос в БД SwissProt
В банке Swissprot было найдено 184 последовательностей (при выставленном максимуме 20000, т.е. это все), из них 5 относятся к таксону Archaea, 203 - Eubacteria, 26 - Eucariotae. В Таблице 1 представлены данные по нескольким находкам в базе данных RefSeq, представленным BLAST-ом.
Таблица 1. Данные о находках в БД RefSeq
| Находка | Accession | Длина выравнивания | Bit score | % Identities | % Positives | E-value |
|---|---|---|---|---|---|---|
| Первое (сама послед-ть) | WP_013467116.1 | 382 | 770 | 100% | 100% | 0.0 |
| Лучшее (второе) выравнивание | WP_056933736.1 | 382 | 768 | 99% | 100% | 0.0 |
| Выравнивание из середины списка | WP_047390110.1 | 383 | 191 | 34% | 56% | 7e-53 |
| Худшее выравнивание | WP_059569933.1 | 666 | 90.5 | 26% | 44% | 2e-16 |
В Таблице 2 представлены данные по нескольким находкам в базе данных SwissProt, представленным BLAST-ом.
Таблица 2. Данные о находках в БД Swissprot
| Находка | Accession | Длина выравнивания | Bit score | % Identities | % Positives | E-value |
|---|---|---|---|---|---|---|
| Первое | P57795.1 | 404 | 300 | 45% | 60% | 2e-96 |
| Выравнивание из середины списка | B8E9D2.1 | 404 | 257 | 42% | 60% | 7e-80 |
| Худшее выравнивание | Q9M1R1.1 | 33.9 | 454 | 34% | 52% | 2.1 |
Далее требовалось выделить среди находок всех гомологов моего белка. Я решила, что предложенный критерий: E-value < 1e-3 и не менее 70% запроса вошло в полученное выравнивание (Query cover) в моём случае подходит, и определила, что есть 163 гомолога. Ссылка на графическое представление выравнивания гомологов
Задание 2.
Ссылка на поисковый запрос по таксону в БД SwissProt
Был проведен поиск по таксону Archaea. Результаты получились иными, нежели в поиске по всем таксонам: среди архей было найдено 11 последовательностей, тогда как среди всех таксонов нашлось лишь 5 последовательностей, принадлежащих археям. Впрочем, среди дополнительных находок 5 последовательностей имеют E-value больше 0.001, а значит, гомологами их назвать нельзя. Данные о выравниваниях с одной и той же последовательностью из двух разных запросов в Таблице 3. Score не изменился (так как использовалась одна и та же матрица и одни и те же 2 последовательности). Изменилось E-value: оно немного уменьшилось вследствие сужения поля поиска (т.е. размеров банка).
Таблица 3. Сравнение выравниваний при разных поисковых запросах
| Вариант поиска | Accession | Длина выравнивания | Bit score | % Identities | % Positives | E-value |
|---|---|---|---|---|---|---|
| По всем таксонам | P57795.1 | 404 | 300 | 45% | 60% | 2e-96 |
| По Archaea | P57795.1 | 404 | 300 | 45% | 60% | 7e-98 |
Задание 3.
Карта локального сходства двух последовательностей: исходной (YP_004071041.1) и выбранной из списка найденных (идентификатор в Swissprot P57795.1) представлена на Рис.2.
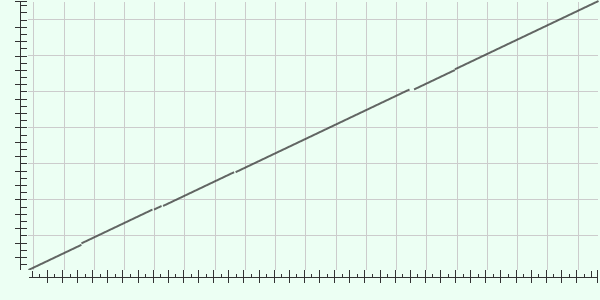
По этой карте видно, что выравнивание покрывает практически всю заданную последовательность (93% query cover). Прерывистость линии обозначает гепы в парном выравнивании. Первый геп двойной, и на рис.2 он больше, чем три последующих, потому что они одинарные. Четвёртый геп тройной, поэтому разрыв линии в этом месте самый большой.

Задание 4.
База данных my_db была создана на основе множественного выравнивания align_06.fasta. в результате поиска в ней гомологов моего белка YP_004071041.1 получено две находки: участок в последовательностях LACLK и DESOD(см. Рис.3).
Оба участка маленькие (8 и 13 позиций, то есть примерно 2,6% общей длины заданной последовательности).
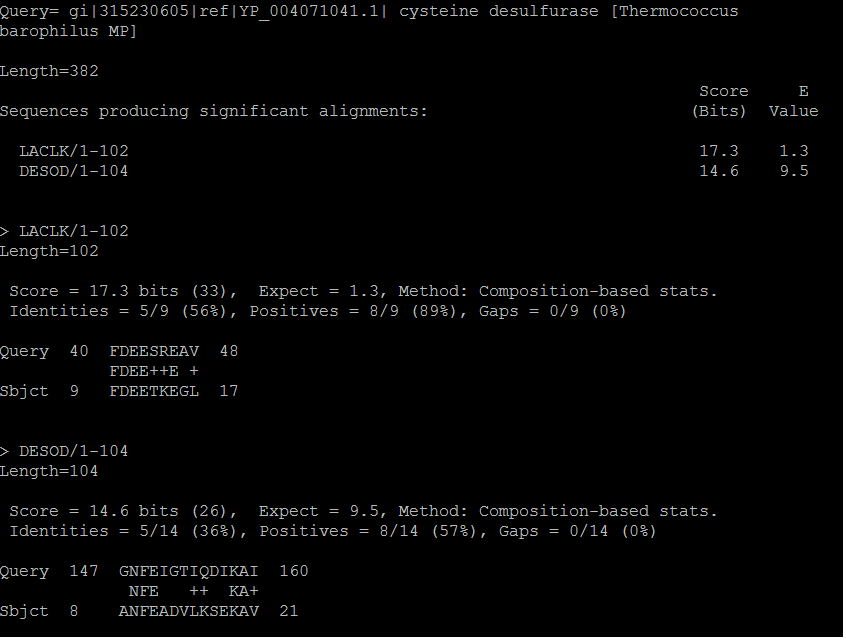
Их характеристики представлены в Таблице 4.
Таблица 4. Данные о находке в БД align_06.fasta
| Находка | LACLK | DESOD |
|---|---|---|
| Длина выравнивания | 18 | 13 |
| Bit score | 17.3 | 14.6 |
| Процент сходных колонок | 89% | 57% |
| Процент идентичных колонок | 56% | 36% |
| Gaps | 0% | 0% |
| E-value | 1.3 | 9.5 |
Приведенные в Таблице 4 параметры дают понять, что эти последовательности не является гомологом моего белка: слишком высокий E-value, низкий bit score. Bit score самой худшей находки в БД Swissprot примерно в двадцать раз больше. Количество идентичных и сходных колонок велико, но лишь относительно длины выровненного участка.