Мини-обзор генома и протеома актинобактерии Saccharopolyspora spinosa
Резюме
Данный мини-обзор представляет из себя работу с геномом и протеомом актинобактерии Saccharopolyspora spinosa с использованием программирования на языке Python и функционала электронных таблиц. Полученные данные по возможности объясняются различными эмпирически выявленными биоинформатиками закономерностями.
Ключевые слова: Актинобактерии, инсектициды, токсины, геном, протеом.
Введение
Целью данного обзора является изучение генома и протеома актинобактерии Saccharopolyspora spinosa из семейства Pseudonocardiaceae. Бактерия принадлежит к роду Saccharopolyspora, первоначально выделенному из образца почвы, собранного с Карибских островов в 1982 году (1). Морфология вида представлена бледно-жёлтым мицелием, несущим цепочки “колючих” спор (1).
Несколько позже было выяснено, что Saccharopolyspora spinosa способна синтезировать инсектицидную смесь спиносад (2). Смесь содержит химические вещества из семейства спинозинов: спинозин A (основной компонент) и спинозин D (второстепенный компонент) (1, 3). Спинозины направлены на перевозбуждение нервной системы насекомых, что приводит к параличу и, в конце концов, смерти (3). При этом он не вызывает перекрёстной устойчивости к другим инсектицидам (по крайней мере это до сих пор не было обнаружено), поскольку действует по уникальным механизмам (4). Спиносад действует на широкий спектр насекомых-вредителей и обладает низкой токсичностью для млекопитающих, что делает его одним из самых эффективных средств, применяющихся в сельском хозяйстве (5).
У Saccharopolyspora spinosa также был найден фермент SpnF, участвующий в синтезе спинозина A и катализирующий в этом процессе реакцию внутримолекулярного циклоприсоединения (реакция Дильса-Альдера) при комнатной температуре (6). Эта интересная особенность - дополнительный стимул к изучению данной бактерии, поскольку такого рода ферменты могут оказаться полезными при изучении механизмов биологического катализа.
Материалы и методы
Используемые данные, скачанные по ссылке [9]: 1) таблица локальных особенностей, Полный геном (fasta-формат) и файл с Cds-последовательностями (fasta-формат).
1. Для построения столбчатых диаграмм №1, №2 и №3 использовался онлайн-офис google sheets. Для построения каждой из них я создавал две колонки: одна из них содержала последовательности k-меров, а другая - соответствующие им cb (объяснения в разделе “результаты и обсуждения”). Далее на другом “листе” я заполнил один столбец арифметической прогрессией с шагом гистограммы (равным 0,2), а в другом столбце посчитал командой ‘счетесли’ встречаемость k-меров с соответствующим значением cb. В программе №1 (python) приводится расчёт cb k-меров любого введённого натурального k. Все комментарии непосредственно в программе.
2. Программа №2 рассчитывает встречаемость всех стоп-кодонов в CDS бактерии. Таблица №1 была сделана с помощью google sheets. Программа №2 параллельно с расчётом встречаемости стоп-кодонов также выводит в файл out.txt имена и описания тех последовательностей, которые содержат “странный” стоп-кодон. На основе этого файла и была заполнена данная таблица.
3. Программа №3 рассчитывает встречаемость кодонов, кодирующих различные аминокислоты: общее число кодонов, минимальную частоту кодонов и максимальную. Таблица №2, сделанная с помощью google sheets, содержит эту информацию в удобном виде (получена аналогично таблице №1).
4. Таблица №3 сделана в google sheets. Она содержит информацию о генах транспортных и рибосомальных РНК. Количество таких РНК я посчитал с помощью функции “статистика по столбцам” google sheets. Применяя эту функцию к первому столбцу ( # feature) таблицы “genome features”, можно узнать информацию о количестве различных “types”: CDS, gene, tRNA, rRNA и так далее.
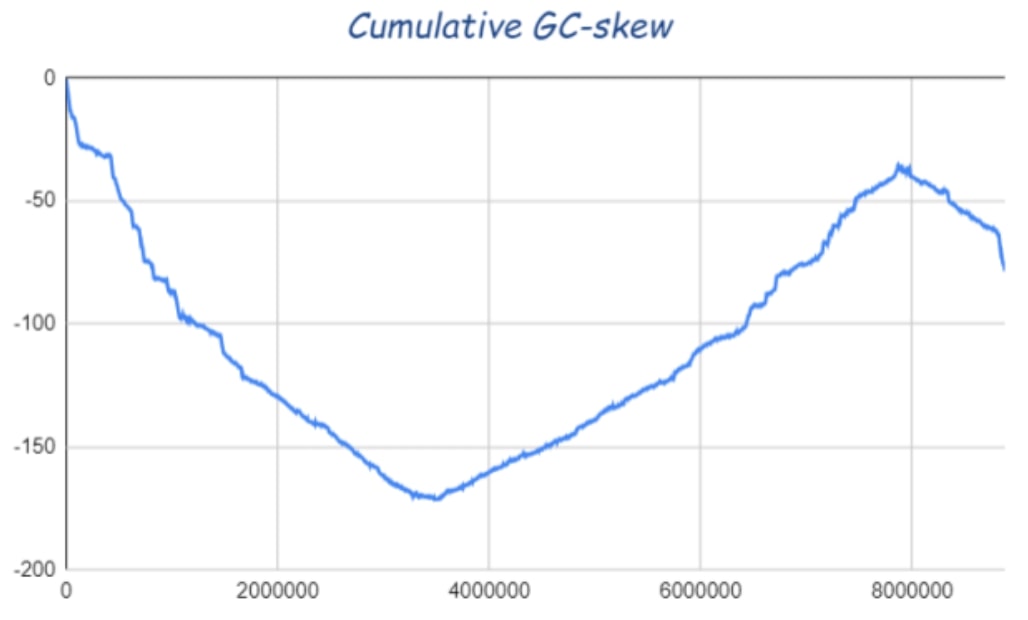
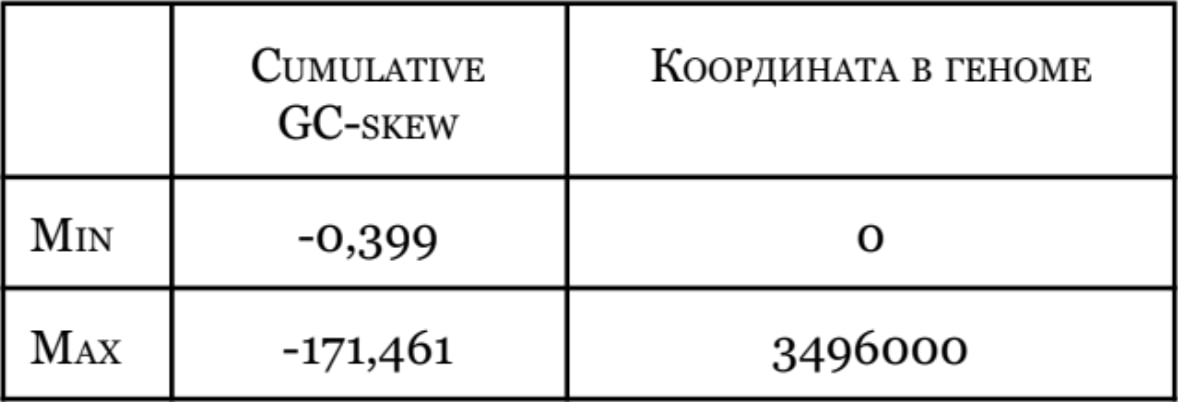
5. Программа №4 рассчитывает GC-skew и cumulative gc-skew, выводя результаты в файл ‘out.txt’. Из данного файла формируется таблица №4 (google sheets), в которой представлен график зависимости cumulative GC-skew от позиции в геноме. С помощью функции Max и Min я рассчитал соответственно максимальное и минимальное значение cumulative GC-skew, а после функцией Vlookup установил соответствующие им позиции в геноме.
Результаты и обсуждения
1.Анализ статистики k-меров в геноме для нескольких значений k.
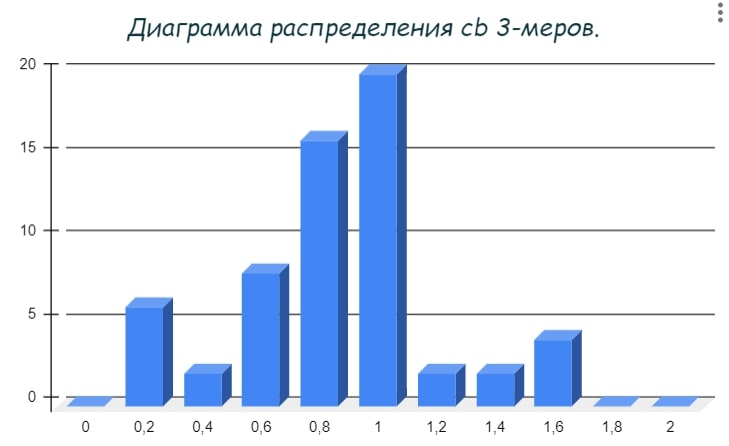
Я решил проверить распределение значений cb (o/e, observed/expected, compositional bias) для k = 3, 4 и 5.
На диаграмме №1 заметны два пика с cb, равными 0,2 и 1,6. Кроме того, в геноме в большей степени распространены недопредставленные 3-меры.
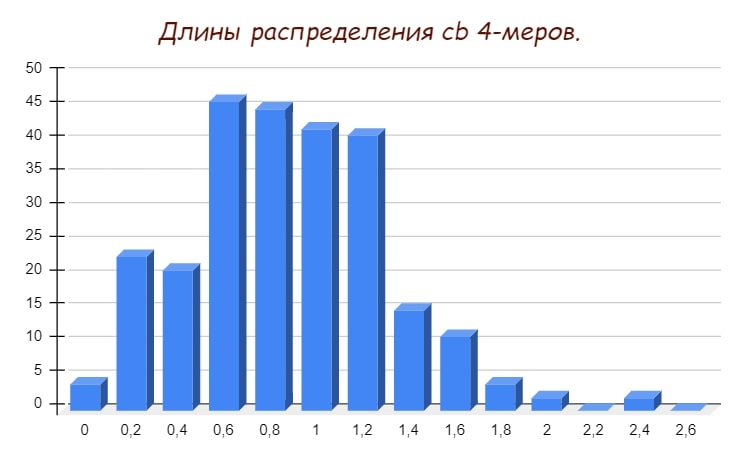
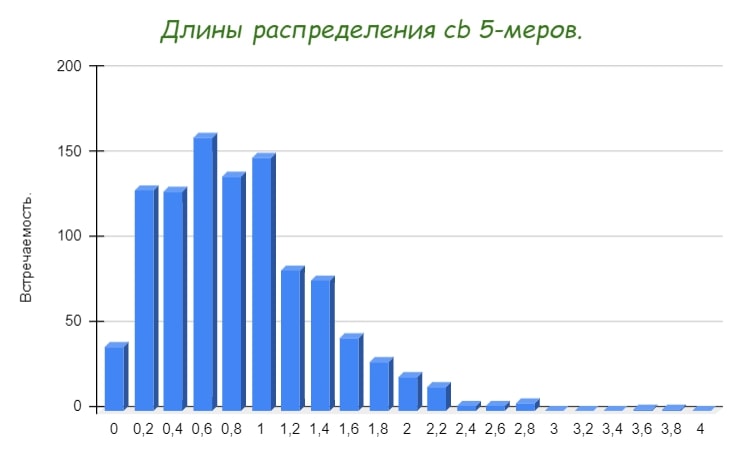
Как видно, с увеличением k распределение становится всё более интересным. Как для 4-меров, так и для 5-меров более распространено cb, отличное от единицы, что, конечно, может быть связано с неточностью метода вычисления ожидаемой встречаемость k-меров.
Из наиболее аномально встречающихся слов можно выделить: ‘TTA’ (cb = 0,267), ‘CTAG’ (cb = 0,153), ‘GATC’ (cb = 2,579), ‘UUAG’ (cb = 0,163), ‘AGATC’ (сb = 2,696), ‘ATCGA’ (cb = 2,833), ‘CCTAG’ (cb = 0,121), ‘TGATC’ (cb = 3,75 ). Заметно, что 5-меры, начинающиеся на CTA (‘CTA**’), аномально мало представлены в геноме. Все они имеют cb, меньший единицы. (например, cb ‘CTAGC’ равен 0,171, а cb ‘CTATC’ = 0,347).
Максимальный cb из группы CTA** имеет CTACG (cb = 0,753), а минимальный - CTAGG (cb = 0,135). Сложно сказать, с чем это связано.
2. Анализ встречаемости стоп-кодонов.
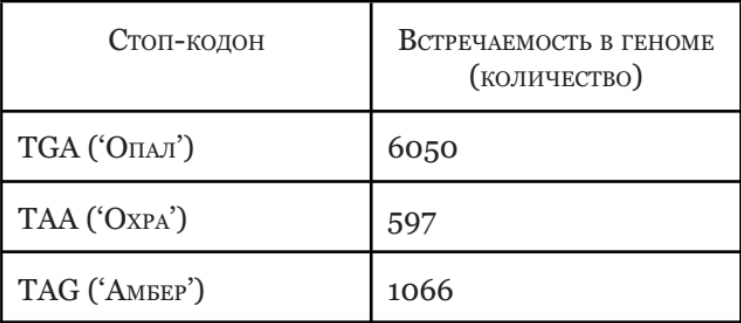
С помощью моей программы (программа №2) я посчитал встречаемость стоп кодонов в геноме Saccharopolyspora spinosa. Количество стоп-кодона “TGA”: 6050, количество стоп-кодона “TAA”: 597, количество стоп-кодона “TAG”: 1066, количество других стоп-кодонов: 212.
В таблице №1 я поместил названия и описания всех последовательностей, которые заканчивались на нестандартный стоп-кодон. Все они оказались псевдогенами. Видна заметная разница между встречаемостью “стандартных” стоп-кодонов.
Как оказалось, высокая встречаемость стоп-кодона “TGA” коррелирует с большим GC-составом генома, стоп-кодона “TAA” наоборот тем больше, тем GC-состав меньше [7]. В программе №2 я также высчитал GC-состав генома бактерии. Он оказался равным 68%. Как мы видим, данное эмпирически установленное правило действительно выполняется: в геноме много “TGA” стоп-кодонов и довольно мало “TAA”.
3. Анализ частоты использования кодонов, кодирующих разные аминокислоты.
Написанная мною программа (программа №2) рассчитывает количество каждого из кодонов, кодирующих определённую аминокислоту. На выходе каждой аминокислоте сопоставляются: общее число кодонов, кодирующих данную аминокислоту, минимальная частота кодона и максимальная частота кодона.
В таблице №2 эта информация содержится в удобном для анализа виде. Эмпирически установлено, что аминокислоты аланин (A), глицин (G), пролин (P) и аргинин (R) встречаются чаще в геномах с высоким GC-составом, в то время как аминокислоты фенилаланин (F), изолейцин (I), лизин (K), аспарагин (N) и тирозин (Y) встречаются реже в таких геномах [8].
Полученные данные согласуются с этим правилом. A, G, P, R встречаются как минимум на порядок чаще, чем F, I, K, N и Y.
Кроме того, можно заметить, что у бактерий сравнительно мало метионина (M), поскольку, в отличие от эукариот, у бактерий для инициации трансляции используется n-формилметионин.
4. Подсчёт количества рибосомальных и транспортных рнк в геноме.
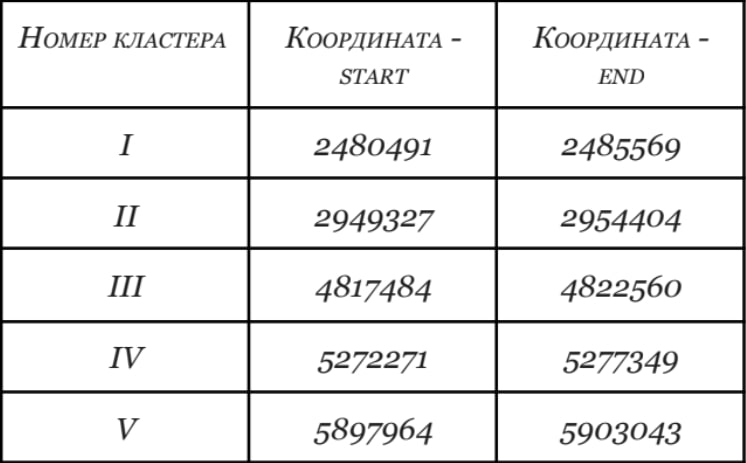
Как было выяснено, в геноме Saccharolyspora spinosa содержится 57 генов транспортных РНК и 15 рибосомальных РНК. информация об этих генах содержится в таблице №3. гены 5s-рибосомальной РНК встретились 5 раз (причём трое из них на “+”-цепи, а двое - на “-”). В таком же виде представлены гены 16s-рРНК и 23s-рРНК. все они собраны в пять кластеров:
Все кластеры содержат рибосомальные РНК и 5s, и 16s, и 23s типов. Примечательно, что каждый из кластеров имеет почти такую же длину, как и у других кластеров, но изменённую на 1-2 нуклеотида. Так, например, кластер I имеет длину в 5079 нуклеотидов, а кластер V - 5080.
5. Вычисление oriC и Ter с помощью параметра comulative GC-skew.
Cumulative GC-skew равен сумме GC-skew, посчитанных для всех предыдущих позиций. Установлено, что минимальному значению cumulative GC-skew соответствует Oric, а максимальному - Ter. Данные получились примерными и, быть может, не отражают реальную картину положения Oric и Ter.
Сопроводительные материалы
1. Программа №1: Анализ статистики k-меров в геноме.
2. Программа №2: Анализ статистики стоп-кодонов.
3. Программа №3: Частота использования кодонов.
5. Гистограммы (№1, №2, №3) распределение cb k-меров.
6. Таблица №1: Описания последовательстей со странными стоп-кодонами.
7. Таблица №2: Встречаемость кодонов аминокислот.
9. Таблица №4: Cumulative GC-skew.
Ссылки на источники
1. Mertz F. P., Yao R. C. 1990. Saccharopolyspora spinosa sp. nov. isolated from soil collected in a sugar mill rum still. Int. J. Syst. Bacteriol. 40:34-39 [https://www.microbiologyresearch.org/content/journal/ijsem/10.1099/00207713-40-1-34?crawler=true]
2. Sparks T. C., et al. 1998. Biological activity of the spinosyns, new fermentation derived insect control agents, on tobacco budworm (Lepidoptera: Noctuidae) larvae. J. Econ. Entomol. 91:1277-1283 [https://academic.oup.com/jee/article/91/6/1277/2216924?login=true]
3. Qiao, Meihua; Daniel E. Snyder; Jeffery Meyer; Alan G. Zimmerman; Meihau Qiao; Sonya J. Gissendanner; Larry R. Cruthers; Robyn L. Slone; Davide R. Young (12 September 2007). ''Preliminary Studies on the effectiveness of the novel pulicide, spinosad, for the treatment and control of fleas on dogs''. Veterinary Parasitology. 150 (4): 345–351. [https://www.sciencedirect.com/science/article/abs/pii/S0304401707004955?via%3Dihub]
4. Sparks, Thomas; Gary D crouse; Gregory Durst (30 March 2001). ''Natural products as insecticides: the biology, biochemistry and quantitative structure-activity relationships of spinosyns and spinosoids''. Pest Manag Sci. 57 (10): 896–905. [https://onlinelibrary.wiley.com/doi/10.1002/ps.358]
5. Hertlein, Mark; Gary D. Thompson; Bhadriraju Subramanyam; Christos G. Athanassiou (12 January 2011). "Spinosad: A new natural product for stored grain protection". Stored Products. 47 (3): 131-146. [https://www.sciencedirect.com/science/article/abs/pii/S0022474X11000142?via%3Dihub]
6. Evgeniy G. Gordeev, Valentine P. Ananikov. Computational Study of a Model System of Enzyme-Mediated [4+2] Cycloaddition Reaction // PLoS ONE. 2015. DOI: 10.1371/journal.pone.0119984 [https://journals.plos.org/plosone/article?id10.1371/journal.pone.0119984]
7. Alexander T Ho, Laurence D Hurst, Variation in Release Factor Abundance Is Not Needed to Explain Trends in Bacterial Stop Codon Usage, Molecular Biology and Evolution, 2021;, msab326, [https://doi.org/10.1093/molbev/msab326]
8. Besemer J, Borodovsky M. Heuristic approach to deriving models for gene finding. Nucleic Acids Res. 1999 Oct 1;27(19):3911-20. doi: 10.1093/nar/27.19.3911. PMID: 10481031; PMCID: PMC148655. [https://pubmed.ncbi.nlm.nih.gov/10481031/]
9. Данные о последовательностях бактерии [https://ftp.ncbi.nlm.nih.gov/genomes/all/GCF/014/490/055/GCF_014490055.1_ASM1449005v1/]
Кирилл Кузенков, студент второго курса ФББ