Практикум №11
Гомология и выравнивание
1.Поиск семейства доменов
Я решил выбрать семейство доменов случайным образом. Перейдя в раздел семейств, начинающихся с буквы ''E'', я нашёл семейство Ead_Ea22. Оно удовлетворяло всем критериям, которые были обозначены в задании.
2. Описание семейства доменов
a) Семейство, выбранное мной, называется Ead/Ea22-подобные белки (Ead/Ea22-like protein).
ID: PF13935.
Это семейство содержит фаговые белки и бактериальный белки, которые, скорее всего, являются встроенными фаговыми белками.
b) Размеры выборок:
Общий размер выборки (Full): 154
Число последовательностей в выравнивании (Seed): 29
c) Семейство содержит 5 доменных архитектур.
d) Я выбрал две наиболее представленные доменные архитектуры:
1) V5URV9_9CAUD, содержит 127 белков.
2) Q9XJM6_BP933, содержит 21 белок.
e) Ни для одного белка из данного семейства не известна трёхмерная структура.
f) Белки данного семейства встречаются в двух доменах жизни: в Viruses (117 белков) и в Bacteria (34 белков).
g) Дата создания HMM профиля выравнивания - 12.10.21 (Tue Oct 12 08:27:12 2021).
Число позиций - 139.
3. Построение карты локального сходства
Я построил карту локального сходства белков с разной доменной архитектурой (V5URV9_9CAUD и Q9XJM6_BP933 - наиболее представленные доменные архитектуры, которые я и выбрал).
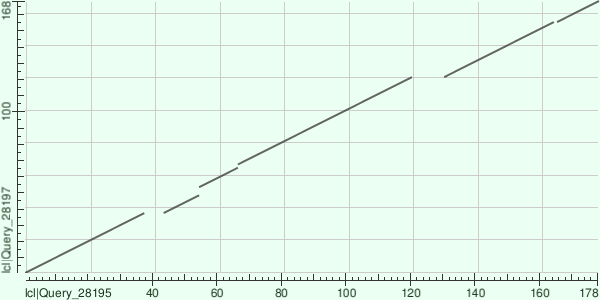
Видно, что произошёл ряд делеций/вставок, самая крупная из которых в 10 аминокислотных остатков.
4. Выделение двух подгрупп доменов Pfam на основании сходства
Я скачал файл всех выровненных последовательностей из выбранного семейства (full, всего их 154) в формате FASTA и в Jalview удалил с помощью Remove Redundancy удалил последовательности с порогом идентичности в 97%, после чего количество последовательностий сократилось до 67. Я был уверен, что искомые мной подгруппы найдутся, поэтому ''без сожаления'' удалил такое большое количество последовательностей.
Я построил филогенетическое древо и на его основании определил две крупные и более или менее одинаковые по представленности подгруппы. Первая - жёлтого цвета (в проекте Jalview), вторая - зелёного.
| Положение | Различие |
|---|---|
| 73 | Во второй подгруппе гораздо больше пролина |
| 75 | Аналогично положению 73. |
| 226 | В первой подгруппе гораздо больше глутамина. |
| 232 | Во второй подгруппе гораздо больше гидрофобных аминокислот. |
| 255 | В первой подгруппе очень много изолейцина. Во второй подгруппе тоже много гидрофобных аминокислот, но помимо изолейцина есть заметное количество лейцина, фенилаланина. |
| 259 | В первой подгруппе исключительно гидрофобные аминокислоты (преимущественно валин). Во второй подгруппе разнообразные гидрофобные аминокислоты, но есть несколько последовательностей с тирозином. |
| 260 | В первой подгруппе гораздо больше глицина |
| 346-397 | Этот участок практически полностью отсутствует во второй подгруппе (крупная делеция). |
| 398-430 | Этот участок практически полностью отсутствует в первой подгруппе (крупная делеция). |
5. Таблица со всеми белками из UniProt с доменом семейства Pfam
Белки семейства я искал с помощью Advanced Research. В качестве последней колонки выбрал Taxonomic lineage (SUPERKINGDOM), поскольку уже на уровне доменов жизни есть различия (Viruses и Bacteria).
Ссылка на таблицу в Excel: Pfam_table.xlsx
Кирилл Кузенков, студент второго курса ФББ