Практикум №8
Поиск информации о белке в UniProt
Введение
В данном практикуме я буду описывать белок AAA88455.1 (индентификатор INSDC CDS), парагидроксибензоат гидроксилаза (альтернативное название: 4-гидроксибензоат 3-монооксигеназа). Оценка аннотации: Experimental evidence at protein level.
Белок в качестве простетической группы содержит FAD (Флавинадениндинуклеотид) и, следовательно, представляет из себя флавопротеин. Принадлежит к классу оксидоредуктаз. В частности является одной из самых изученных гидроксилаз. Катализирует реакцию, участвующую в том числе в детоксикации почв, метаболизме (процессы биосинтеза) [1].
4-гидроксибензоат 3-монооксигеназа является гомодимером. С каждым мономером связан флавин, находящийся в активном центре. Структура этого фермента часто служит моделью взаимодействия структуры и реакционной способности других флавинзависимых гидроксилаз [4].
Белок найден в Pseudomonas aeruginosa, грамотрицательной бактерии, знаменитой благодаря своим механизмам устойчивости к антибиотикам и образованию многоклеточных биоплёнок [5].
Функции и механизм катализа
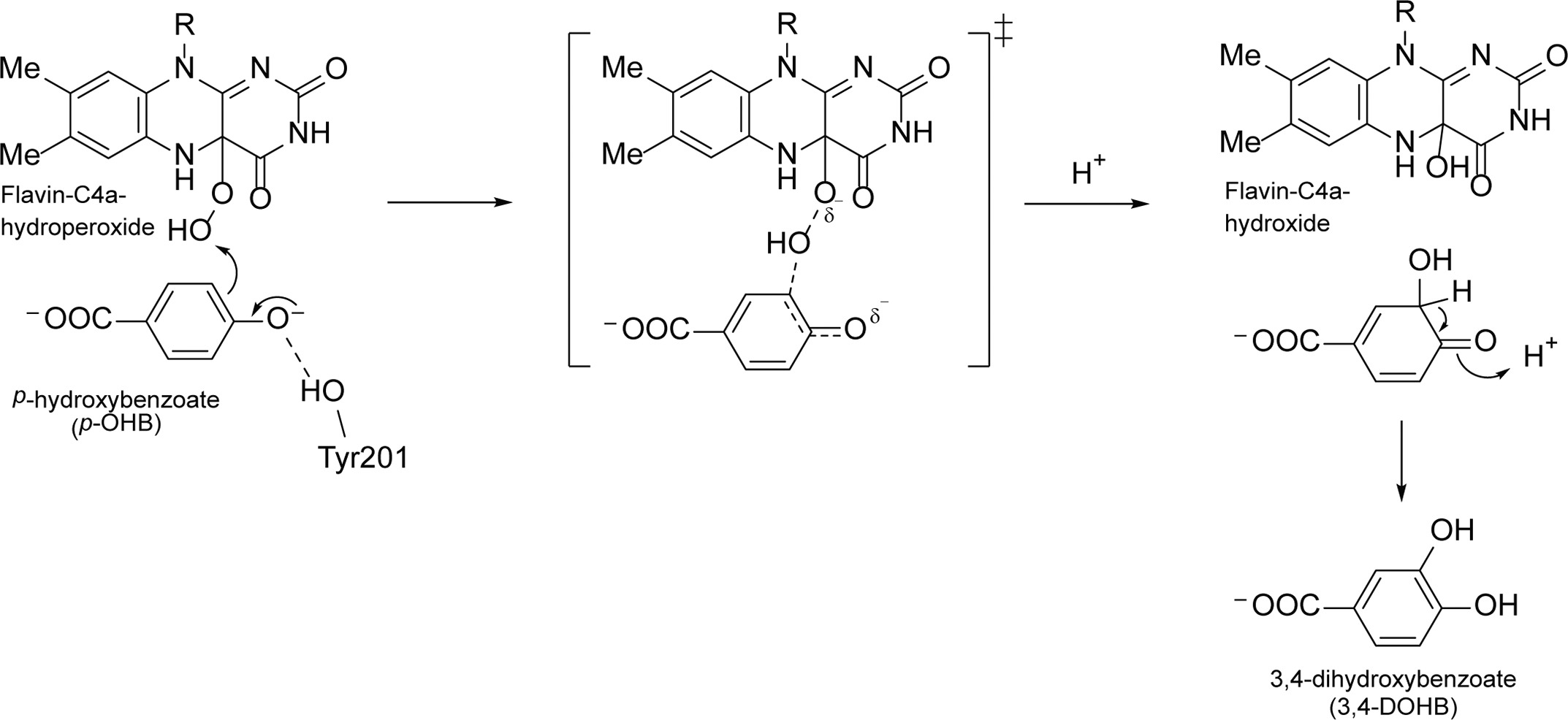
Фермент катализирует реакцию гидроксилирования пара-гидроксибензоата (p-OHB) с образованием 3,4-дигидроксибензоата (3,4-DOHB). Реакция протекает в две стадии: 1) В ответ на связывание пара-гидроксибензоата с ферментом происходит восстановление флавинадениндинуклеотида восстановленным никотинамидадениндинуклеотидфосфатом (NADPH), 2) Окисление восстановленного FAD кислородом с образованием гидропероксида, посредством которого затем происходит встраивание кислорода в p-гидроксибензоат [2].
Полный EC:1.14.13.2 [2].
В процессе катализа происходят перемещения флавина и белка между тремя конформациями: in, out и open. Каждая из конформаций важна для реализации следующих процессов: конформация ''open'' необходима для связывания субстрата и реализации (высвобождения) продукта, ''in'' - непосредственно для процесса гидроксилирования (встраивания кислорода в пара-гидроксибензоат), ''out'' - для восстановления FAD посредством NADPH [6].
Данные о структуре
Было найдено 19 записей о данном белке в PDB (19 идентификаторов в файле protein_info.txt). Все 3D-структуры фермента были сделаны с помощью рентгеноструктурного анализа. Минимальное разрешение - 1.60 Å (PDB identifier 6JU1). Имеется предсказанная с помощью методом молекулярного моделирования структура (Identifier AF-P20586-F1), поддерживаемая экспериментальными данными.
Известно, что субстрат (p-гидроксибензоат) связывается в позиции 212-214 [7].
Кластеры
Информацию о кластерах можно найти в файле protein_clusters.txt.
В кластере UniRef100_P20586 исследуемый белок является Seed & Representative. Все белки кластера найдены в Pseudomonas aeruginosa, но некоторые имеют другую длину последовательности.
В кластере UniRef90_P00438 статус Representative имеет другой белок - P00438 из организма Pseudomonas fluorescens, имеющий такую же длину последовательности, как и исследуемый фермент. При этом большинство других белков, входящих в этот кластер, найдены в Pseudomonas aeruginosa.
В кластере UniRef50_P00438 Representative является тот же белок, что и в кластере UniRef90. Видовой состав здесь самый разнообразный и включает представителей, далёких от рода Pseudomonas.
Прочее
В файле uniprot_queries.txt представлены проведённые мной запросы с использованием Advanced searching.
Можно заметить, что данный фермент встречается совсем не только у рода Pseudomonas, как можно было бы предположить. Найдено 4221 записей (и только одна из них в Swiss-Prot) о ферментах, имеющих тот же EC, что и изучаемый, но представленных в других таксонах. Причём, как говорит один из запросов, у Metazoa и Embryophyta этот белок не встречается.
Известные некоторые мутации, способные изменить поведение фермента. Например, мутация, которая в позиции 45 меняет аденин на глицин, превращает белок в ассиметричный димер. Причём один из мономеров похож (хоть и не идентичен) на таковой у дикого типа, в то время как другой мономер претерпевает значительные изменения, из-за чего этот мономер не может участвовать в гидроксилировании [8].
Или, например, замена аргинина на глутамин в 220 положении снижает сродство фермента к пара-гидроксибензоату [9].
Как видно из запросов, фермент с данным EC может иметь в качестве кофактора не только FAD. Но такие белки по большей части имеют статуы ''Predicted''. А состояние ''Evidence at protein level'' для этого белка достигается лишь четыре раза.
Сравнение протеомов
Для сравнения я выбрал два протеома: UP000002332 и UP000002438. Оба являются референсными.
Они скачаны на kodomo командами, приведёнными в файле download_proteomes.txt
Контрольный протеом (UP000002332) принадлежит бактерии Pseudomonas fluorescens, представляющая гораздо меньшую опасность для человека, чем Pseudomonas aeruginosa (протеом с исследуемым белком - UP000002438), которая обладает множественной лекарственной резистентностью (Высокая устойчивость к противомикробным препаратам) и вызывает ряд широко распространённый внутрибольничных инфекций [5].
P. fluorescens входит в один род с P. aeruginosa, поэтому является довольно близким родственником, хоть и входит в другую ''группу'' внутри рода [10].
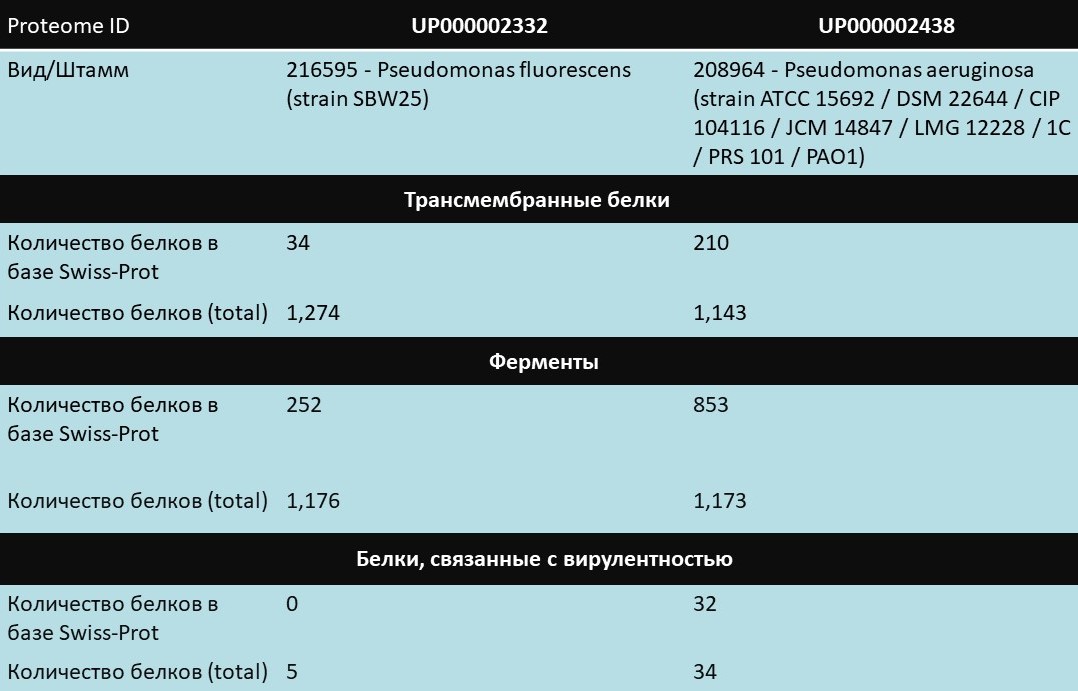
Поскольку P. aeruginosa гораздо опаснее P. fluorescens, можно заранее предполжить, что протеом первой бактерии будет изучен в большей степени. Это предположение подтверждается информацией, представленной ниже. В протеоме UP000002438 соотношение белков в базе Swiss-Prot к общему количеству белков гораздо больше (25,8%), чем в протеоме UP000002332 (6,4%).
В файле proteomes_queries.txt представлены запросы в UniProt с использованием Advanced searching, с помощью которых я оценивал количество трансмембранных белков, ферментов и белков, связанных с вирулентностью.
Используя запросы, я определил соотношение белков:
Трансмембранных белков примерно поровну у обеих бактерий, но белков из базы Swiss-Prot большей у протеома UP000002438, что может говорить о том, что он в лучшей степени аннотирован.
Ферментов также поровну, но, как и в первом случае, количество белков из Swiss-Prot гораздо больше у второго протеома.
Поскольку исследуемая бактерия в большей степени патогенная (и представляет большую угрозу для человека), чем Pseudomonas fluorescens, то и вирулентных белков у неё должно быть больше. Это предположение подтвердилось запросом в UniProt. При этом почти все вирулентные белки протеома UP000002438 находятся в базе Swiss-Prot, что говорит, вероятно, о том, что они представляют высокий интерес для изучения с целью поиска методов борьбы с данными агентами.
Дополняя сравнение, я решил создать список из десяти самых встречаемых KW в данных протеомах. Посредством кода на Python я создал файлы List_KW_UP000002332.txt и List_KW_Up000002438.txt (первый содержит сплошной список KW и впоследствии в большей степени будет обработан средствами bash, второй же в большей степени готов средствами python, но нуждается в сортировке с помощью bash), которые затем скачал на kodomo (через sftp). С помощью команд Top:_proteomes.txt я сделал файлы TopList_KW_UP000002332.txt и TopList_KW_UP000002438.txt, которые содержат тридцать самых встречаемых KW в соответствующих протеомах.
В первую очередь можно сказать о том, что именно для белков из протеома UP000002438 известно большее количество 3D структур (650 упоминаний в качестве KW). Это в очередной раз можно связать с тем, что протеом патогенной и опасной бактерии просто более интересен, поэтому и соответствующие белки стараются изучать более активно.
Конечно, нельзя не заметить наличие плазмиды у Pseudomonas fluorescens (штамм SBW25).
Кроме того, в протеоме UP000002332 заметно больше упоминается в качестве KW внутренний бислой плазмалеммы. Сложно назвать особую причину.
Наличие плазмиды Pseudomonas fluorescens (strain SBW25) заинтересовало меня, поэтому я решил также выявить распределение KW в записях белков, закодированных в данной плазмиде. Я получил список все ключевых слов в данной плазмиде (он продемонстрирован в файле List_KW_UP000002332_pl.txt, полученном с помощью скрипта, аналогичного предыдущему, и последующей сортировки в bash). Меня заинтересовало наличие так называемого ''ANK repeat''. Такие повторы широко распространены у эукариот и в геномах прокариот встречаются редко, а чаще всего попадают посредством горизонтального переноса генов [11].
Как оказалось, плазмида pQBR103 наблюдается в сообществах Pseudomonas fluorescens, колонизирующих сахарную свёклу. Известно, что она повышает конкурентноспособность своего хозяина (устойчивость к ртути, поскольку в файле List_KW_UP000002332_pl.txt пять раз встречается упоминание ''Mercuric resistance''), а часть генов активно экспрессируется лишь в растительной среде (индукция растениями). Анализ последовательности показал большую степень генетической новизны [12].
Можно ли предположить, что плазмида была сформирована посредством горизонтального переноса генов с растениями? Прямого упоминания в статьях не нашёл, но это вполне возможно.
Далее с помощью команд в bash я решил определить первый аминокислотный остаток в каждом из белков данных протеомов. Все белки, как оказалось, начинаются с метионина.
Ссылки на источники
1. Entsch B, Ballou DP, Begley TP (2007-01-01). Wiley Encyclopedia of Chemical Biology. John Wiley & Sons, Inc.
2. Entsch B, Palfey BA, Ballou DP, Massey V. Catalytic function of tyrosine residues in para-hydroxybenzoate hydroxylase as determined by the study of site-directed mutants. J Biol Chem. 1991 Sep 15;266(26):17341-9. PMID: 1910043.
3. Christopher J. Thibodeaux, ... Hung-wen Liu, in Methods in Enzymology, 2019. Chapter Seven - Unraveling flavoenzyme reaction mechanisms using flavin analogues and linear free energy relationships.
4. Montersino S, Tischler D, Gassner GT, van Berkel WJ (September 2011). ''Catalytic and Structural Features of Flavoprotein Hydroxylases and Epoxidases''. Advanced Synthesis & Catalysis. 353 (13): 2301–2319. doi:10.1002/adsc.201100384.
5. Diggle SP, Whiteley M. Microbe Profile: Pseudomonas aeruginosa: opportunistic pathogen and lab rat. Microbiology (Reading). 2020 Jan;166(1):30-33. doi: 10.1099/mic.0.000860. Erratum in: Microbiology (Reading). 2021 Aug;167(8): PMID: 31597590; PMCID: PMC7273324.
6. Gatti DL, Entsch B, Ballou DP, Ludwig ML. pH-dependent structural changes in the active site of p-hydroxybenzoate hydroxylase point to the importance of proton and water movements during catalysis. Biochemistry. 1996 Jan 16;35(2):567-78. doi: 10.1021/bi951344i. PMID: 8555229.
7. Lah MS, Palfey BA, Schreuder HA, Ludwig ML. Crystal structures of mutant Pseudomonas aeruginosa p-hydroxybenzoate hydroxylases: the Tyr201Phe, Tyr385Phe, and Asn300Asp variants. Biochemistry. 1994 Feb 15;33(6):1555-64. doi: 10.1021/bi00172a036. PMID: 8312276.
8. Cole LJ, Gatti DL, Entsch B, Ballou DP. Removal of a methyl group causes global changes in p-hydroxybenzoate hydroxylase. Biochemistry. 2005 Jun 7;44(22):8047-58. doi: 10.1021/bi050108x. PMID: 15924424.
9. Wang J, Ortiz-Maldonado M, Entsch B, Massey V, Ballou D, Gatti DL. Protein and ligand dynamics in 4-hydroxybenzoate hydroxylase. Proc Natl Acad Sci U S A. 2002 Jan 22;99(2):608-13. doi: 10.1073/pnas.022640199. PMID: 11805318; PMCID: PMC117353.
10. Girard L, Lood C, Höfte M, Vandamme P, Rokni-Zadeh H, van Noort V, Lavigne R, De Mot R. The Ever-Expanding Pseudomonas Genus: Description of 43 New Species and Partition of the Pseudomonas putida Group. Microorganisms. 2021 Aug 18;9(8):1766. doi: 10.3390/microorganisms9081766. PMID: 34442845; PMCID: PMC8401041.
11. Bork P. Hundreds of ankyrin-like repeats in functionally diverse proteins: mobile modules that cross phyla horizontally? Proteins. 1993 Dec;17(4):363-74. doi: 10.1002/prot.340170405. PMID: 8108379.
12. Tett A, Spiers AJ, Crossman LC, Ager D, Ciric L, Dow JM, Fry JC, Harris D, Lilley A, Oliver A, Parkhill J, Quail MA, Rainey PB, Saunders NJ, Seeger K, Snyder LA, Squares R, Thomas CM, Turner SL, Zhang XX, Field D, Bailey MJ. Sequence-based analysis of pQBR103; a representative of a unique, transfer-proficient mega plasmid resident in the microbial community of sugar beet. ISME J. 2007 Aug;1(4):331-40. doi: 10.1038/ismej.2007.47. Epub 2007 Jul 5. PMID: 18043644; PMCID: PMC2656933.
Кирилл Кузенков, студент второго курса ФББ