Практикум №3
A- и В- формы ДНК. Структура РНК.
1. Построение моделей ДНК.
С помощью программы fiber пакета 3DNA построил модели A-, B- и Z-формы ДНК. В результате получились следующие файлы:
2. Построение моделей ДНК.
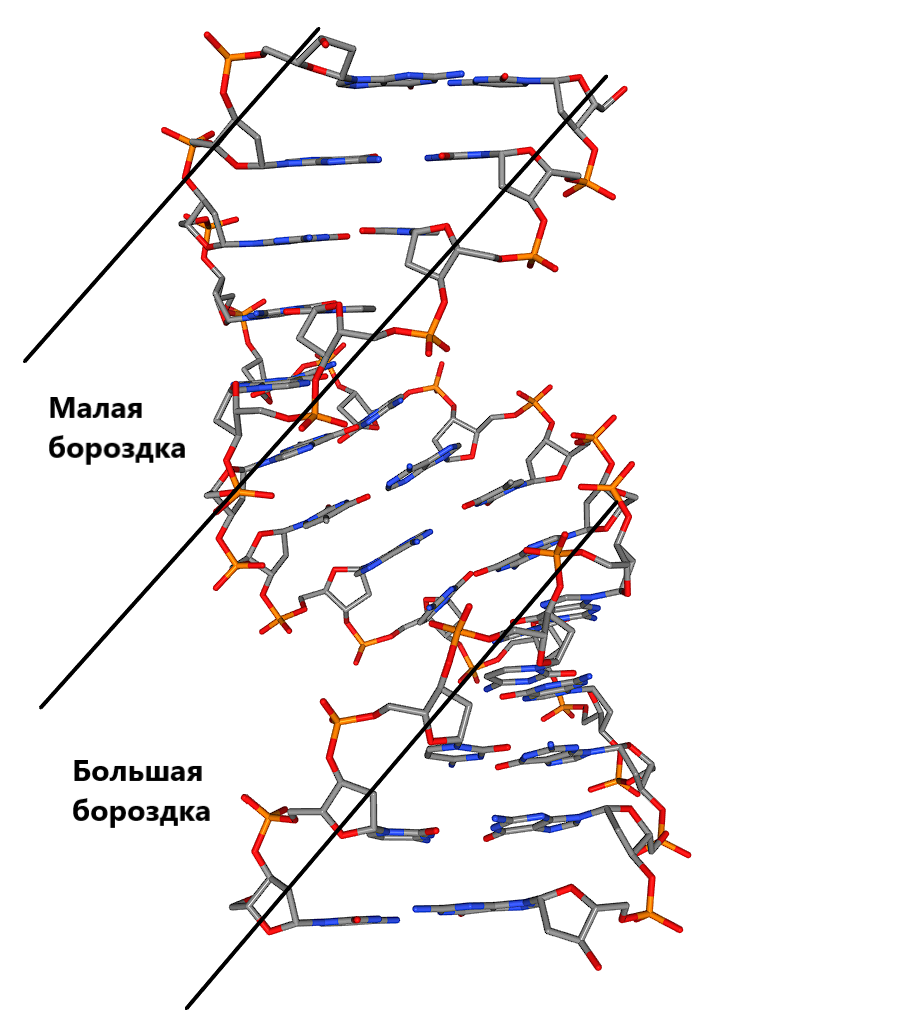
Выбрав A-форму ДНК, я решил рассмотреть сгенерированную модель и её экспериментальную структуру 3v9d с помощью NGLview. Я определил положение большой и малой бороздок в экспериментальной структуре, изобразив их на рисунке №1.
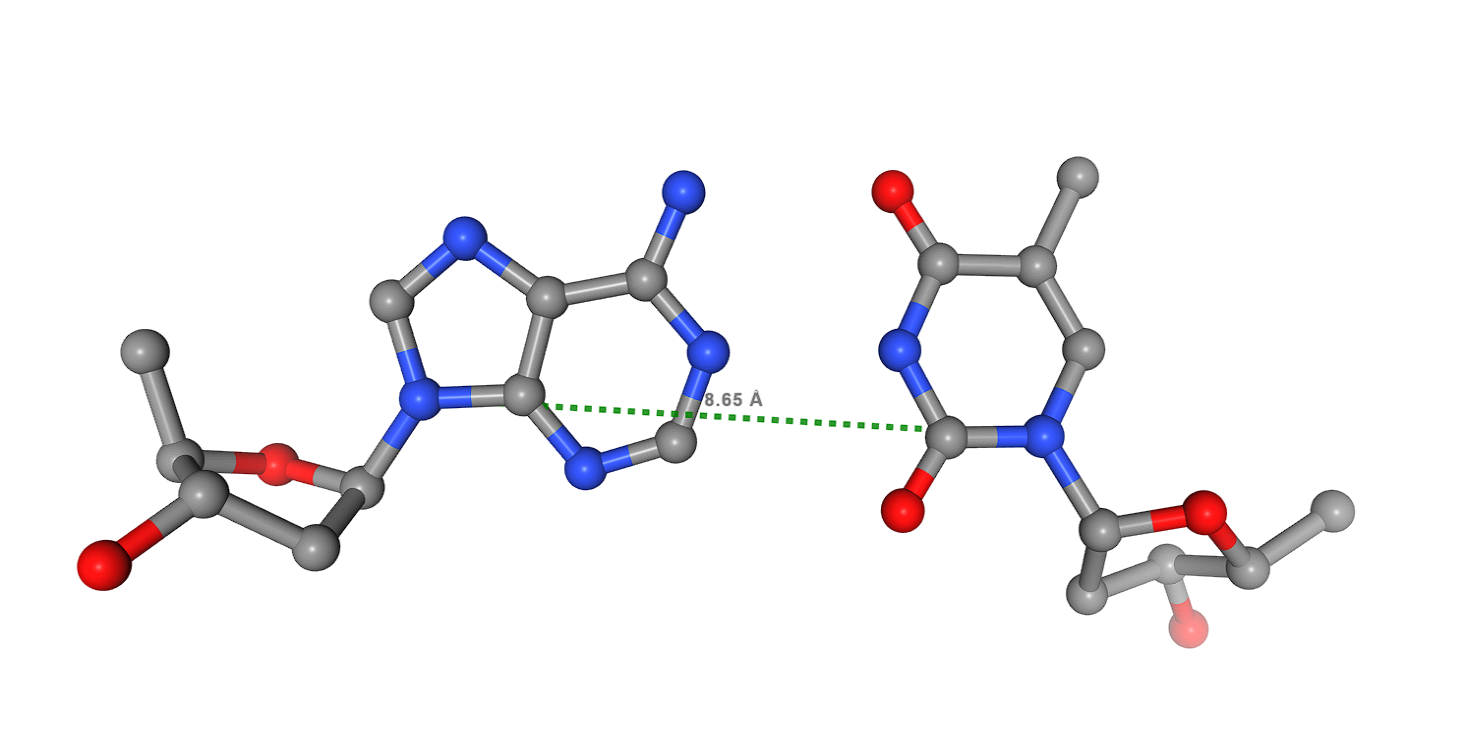
Далее я выбрал 7-ой тимин в цепи А и отобразил его на следующих рисунках c комплементарным ему аденином цепи B. Также я визуализировал аденин в паре с тиминином, проведя дополнительно линию, соединяющую азоты, которые формируют N-гликозидные связи. Это было сделано для того, чтобы легче определить, какие из атомов тимина ''смотрят'' в большую бороздку, а какие в малую.
Исходя из вышепредставленных данных, можно сделать вывод, что у тимина в малую бороздку смотрят атомы (отмечены синим на рисунке №3): [DT]7:A.N1, [DT]7:A.C2, [DT]7:A.O2. В большую (отмечены красным на рисунке №3): [DT]7:A.N3, [DT]7:A.C4, DT]7:A.O4, [DT]7:A.C5, [DT]7:A.C6, [DT]7:A.C7.
Ниже приведена таблица с характеристиками основных форм ДНК. Измерение проводилось с использованием NGLview.
| A-форма | B-форма | Z-форма | |
| Тип спирали (правая или левая) | правая | правая | левая |
| Шаг спирали (Å) | 28.03 | 33.75 | 43.5 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки | 7.98 ([DT]15:A.P-[DC]28:B.P) | 17.21 ([DG]25:B.P-[DC]12:A.P) | 7.20([DC]12:A.P-[DC]26:B.P) |
| Ширина малой бороздки | 16.81 ([DG]9:A.P-[DA]26:B.P) | 11.69 ([DT]31:B.P-[DA]14:A.P) | 11.58 ([DG]11:A.P-[DG]35:B.P) |
3. Работа с тРНК.
Далее в этом практикуме я определил некоторые параметры структуры 1j1u. Я загрузил файл 1j1u.pdb на kodomo и с помощью команды remediator --old 1j1u.pdb > 1j1u_old.pdb перевёл его в ''старый'' формат. Затем я ввёл команду find_pair -t 1j1u_old.pdb stdout | analyze, результатом которой стало несколько файлов, необходимых для дальнейшей работы.
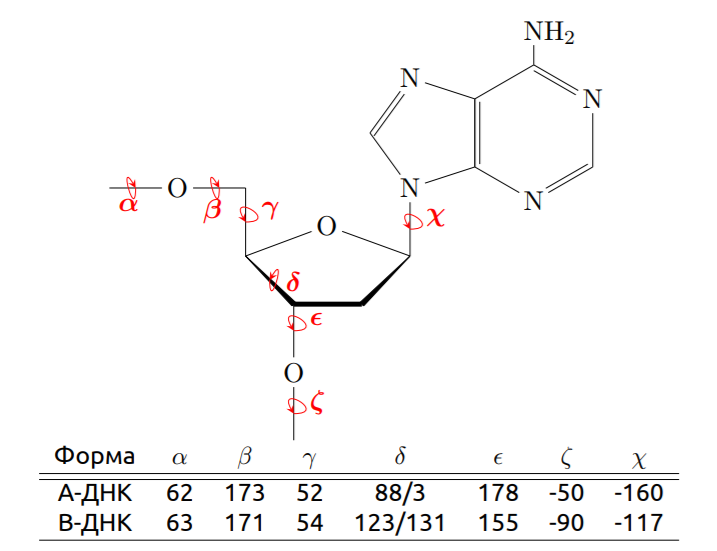
1.1. Из полученного файла 1j1u_old.out я получил информацию о торсионных углах нуклеотидов. Я посчитал средние всех углов, получив следующий результат:
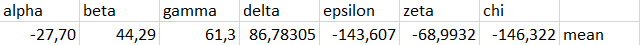
Исходя из справочных данных и полученных средних по всем значениям торсионных углов, можно сделать вывод, что тяжи данной структуры более всего схожи с A-формой ДНК. Более всего это заметно по углу delta и chi. Другие углы довольно сильно отличаются от соответствующих углов в ДНК.
2.2. Затем я рассмотрел раздел ''RMSD of the bases'' в вышеупомянутом файле и определил координаты стеблей.

а) Координаты стеблей: 501...507 - 573...567, 550...554 - 566...562, 537...545 - 534...527, 510...513 - 526...523
b)Неканонические пары: 552:U-*---G:564, 555:U-**--A:559,
556:U-**+-G:519, 537:A-**--U:534,
539:A-**--C:533, 545:A-**--G:527,
514:A-**--U:508, 515:G-**+-C:549.
c)Стабилизирующие водородные связи: 555:U-**--A:559, 556:U-**+-G:519.
Кирилл Кузенков, студент второго курса ФББ
-Т(7).png)