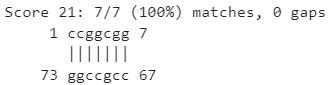Практикум №4
Комплексы ДНК-белок.
1. Программа einverted EMBOSS и предсказание вторичной структуры РНК с помощью ViennaRNA. Сравнение предсказаний с реальной структурой.
С помощью программы einverted EMBOSS я получил предсказания инвертированных участков в нуклеотидной последовательности исследуемой тРНК(1j1u).
Далее представлены команды с некоторыми параметрами и соответствующие им предсказания:
a) einverted -sequence rna.seq -gap 12 -threshold 0 -match 3 -mismatch -4 -outfile outfile -outseq seqout
Очевидно, что это предсказание слабо слабо соответствует данной тРНК, поскольку, исходя из предсказания, в ней имеется всего один инвертированный участок. Это не соответствует действительности. Следовательно, стандартные параметры программы einverted и понижание порога не помогли.
b) einverted -sequence rna.seq -gap 0 -threshold 0 -match 2 -mismatch -0 -outfile outfile0020 -outseq seqout0020
С помощью цикла я подобрал параметры, которые в большей степени соответствуют реальной структуре данной тРНК. Полученное предсказание видно на рисунке №2.
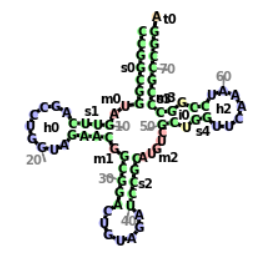
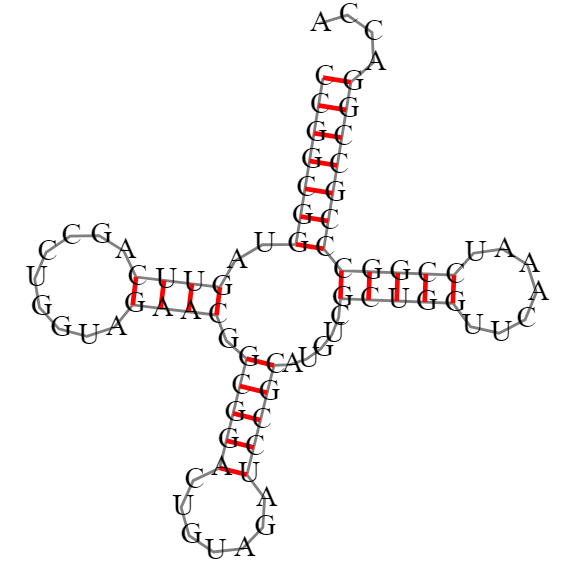
Далее я предсказал вторичную структуру тРНК по алгоритму Зукера с помощью ViennaRNA.
После получения данных я сравнил реальную с предсказанными структурами. В итоге получилась следующая таблица:
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5'-1-7-3' 5'-67-73-3' Всего 7 канонических пар оснований. |
Верно предсказано 6 из 7 пар. | Верно предсказаны все пары. |
| D-стебель | 5'-10-14-3' 5'-23-26-3' Всего 4 канонических пар оснований. |
Плохое предсказание. | Верно предсказаны все пары. |
| T-стебель | 5'-50-54-3' 5'-62-66-3' Всего 4 канонических пар оснований, 1 - неканоническая. |
Плохое предсказание | Верно предсказаны все пары |
| Антикодоновый стебель | 5'-37-45-3' 5'-27-34-3' Всего 5 канонических пар оснований, 3 - неканонических. |
Плохое предсказание. | Плохое предсказание |
| Общее число канонических пар нуклеотидов | 20 | 25, из них 6 предсказаны верно. | 21, из них 16 предсказаны верно. |
2. Поиск ДНК-белковых контактов в заданной структуре.
Мне была задана структура ''1ozj''.
Далее я написал скрипты, определяющие следующие наборы атомов: 1) set1 - множество атомов кислорода 2'-дезоксирибозы, 2) set2 - множество атомов кислорода в остатке фосфорной кислоты, 3) set3 - множество атомов азота в азотистых основаниях.
Следующий скрипт отображает последовательные стадии в JMol.
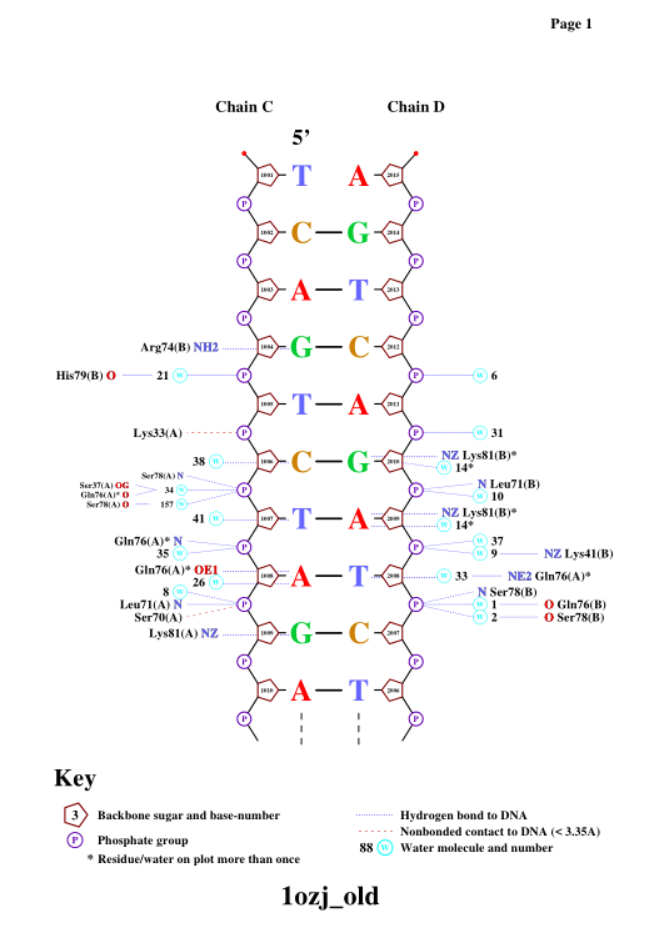
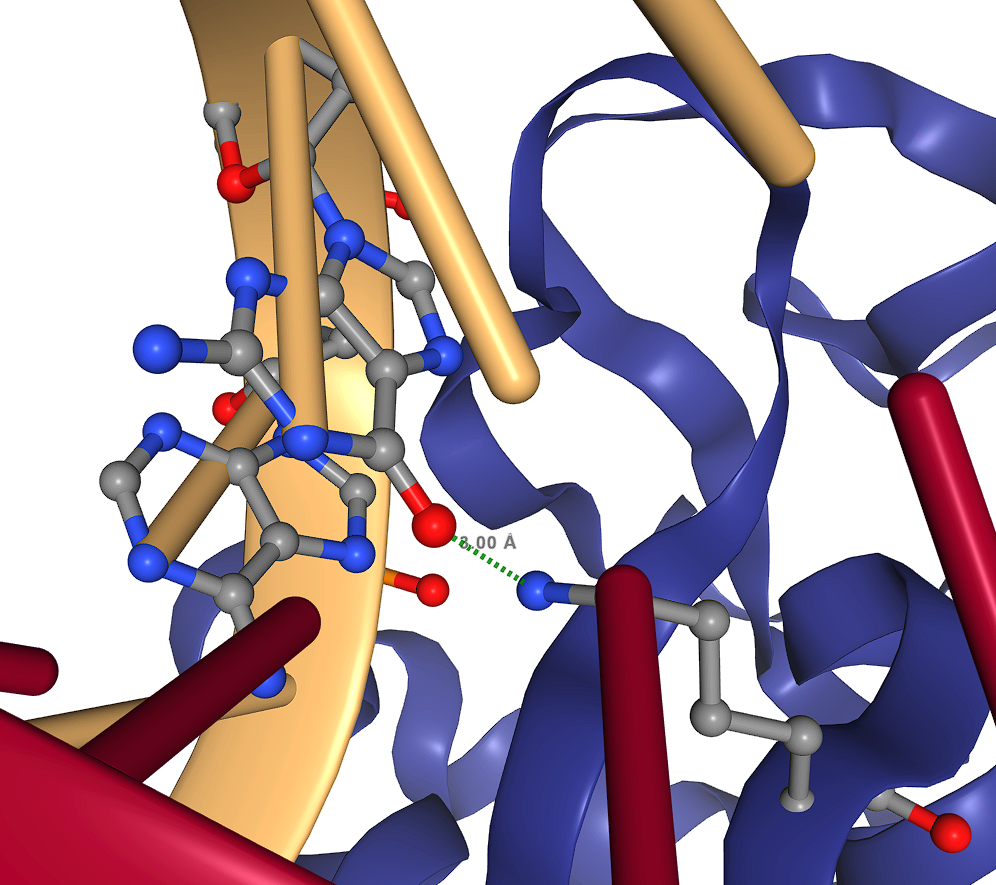
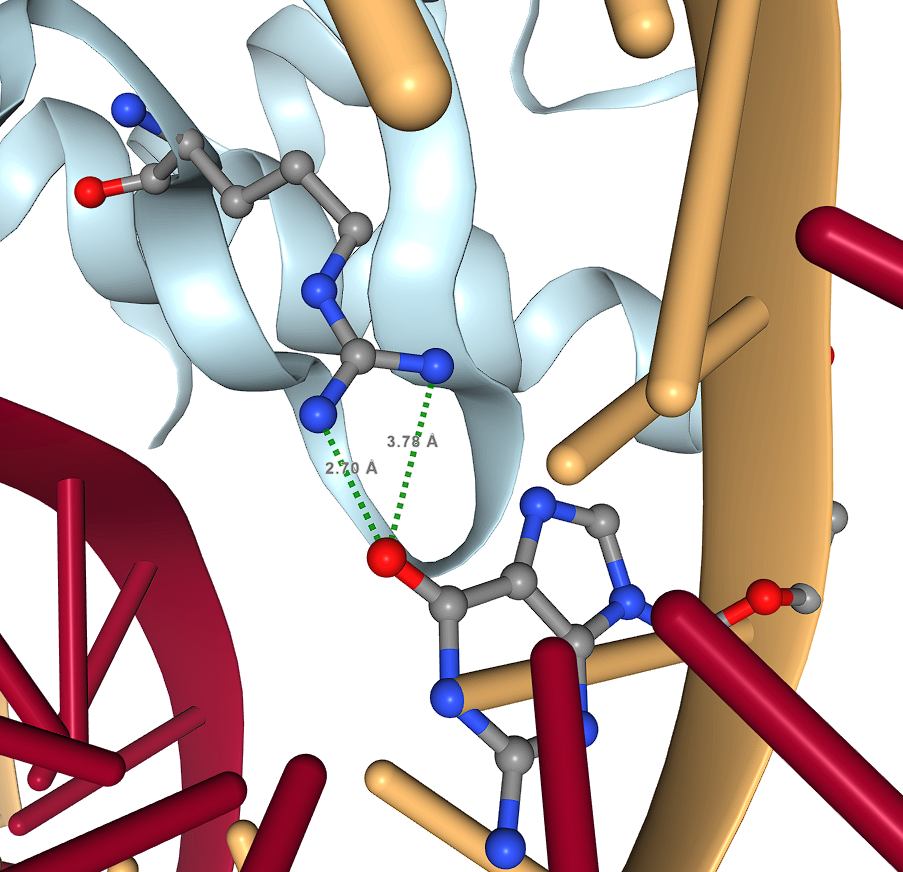
В итоге была получена таблица контактов ДНК-белок:
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 5 | 30 | 40 |
| остатками фосфорной кислоты | 1 | 20 | 40 |
| остатками азотистых оснований со стороны большой бороздки | 10 | 10 | 25 |
| остатками азотистых оснований со стороны малой бороздки | 4 | 3 | 8 |
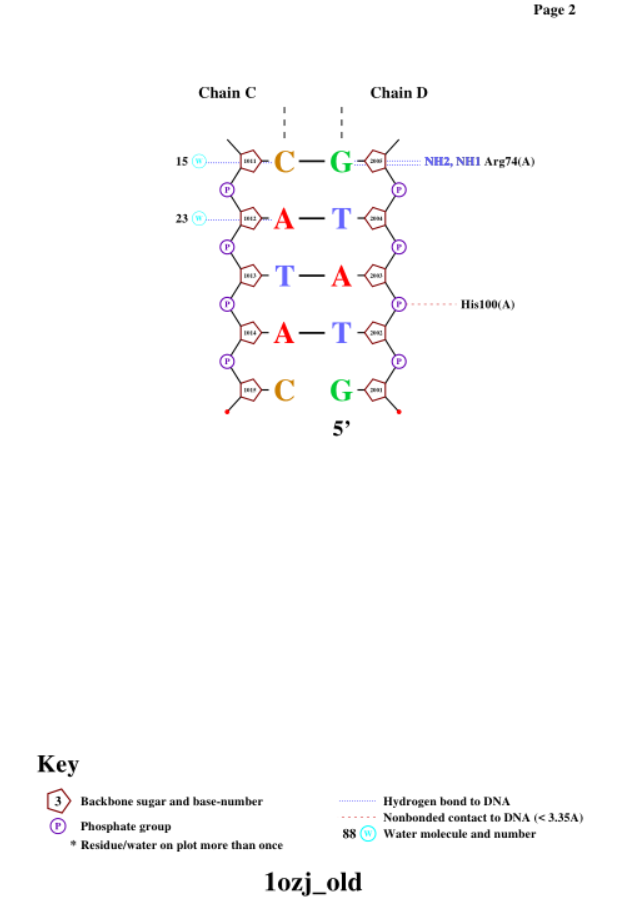
С помощью программы nucplot я получил схему взаимодействий ДНК-белок.
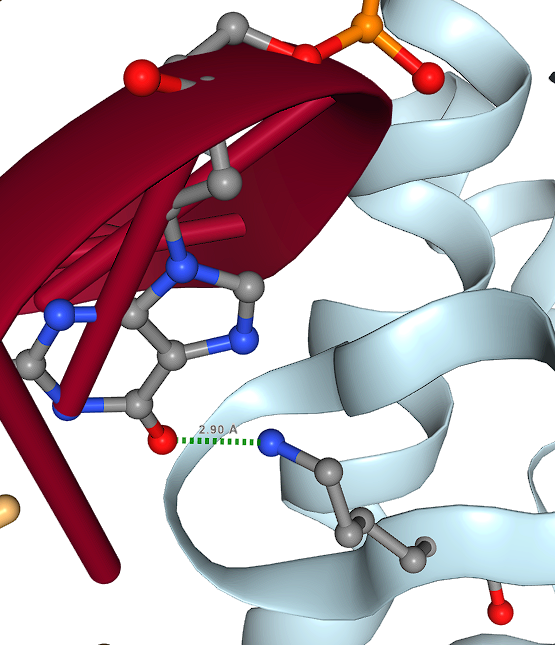
Аминокислотный остаток, формирующий наибольшее количество контактов на схеме - Lys81. Самый важный для распознавания ДНК - Arg74, поскольку он формирует целых две связи с гуанином цепи D.
The end...
Кирилл Кузенков, студент второго курса ФББ