Практикум №8
Нуклеотидный BLAST.
Задание №1: Выбор короткого контига и различные варианты BLAST.
С помощью таблицы локальных особенностей генома я нашёл относительно короткий контиг, на которым имеется CDS. Не удивительно, что он является unplaced genomic scaffold, поскольку именно они оказались самыми короткими. Тем не менее, из него я взял кусок complement(join(6423..6996,7705..7760), поскольку именно он соответствовал найденному CDS в контиге (вырезаны только экзоны). Ниже перечисляю некоторые характеристики фрагмента:
1. NW_026060255.1 (RefSeq-Accn контига). Из этого контига взят фрагмент complement(join(6423..6996,7705..7760).
2. Длина - 630 bp
Далее привожу схему расположения генов на данном контиге.
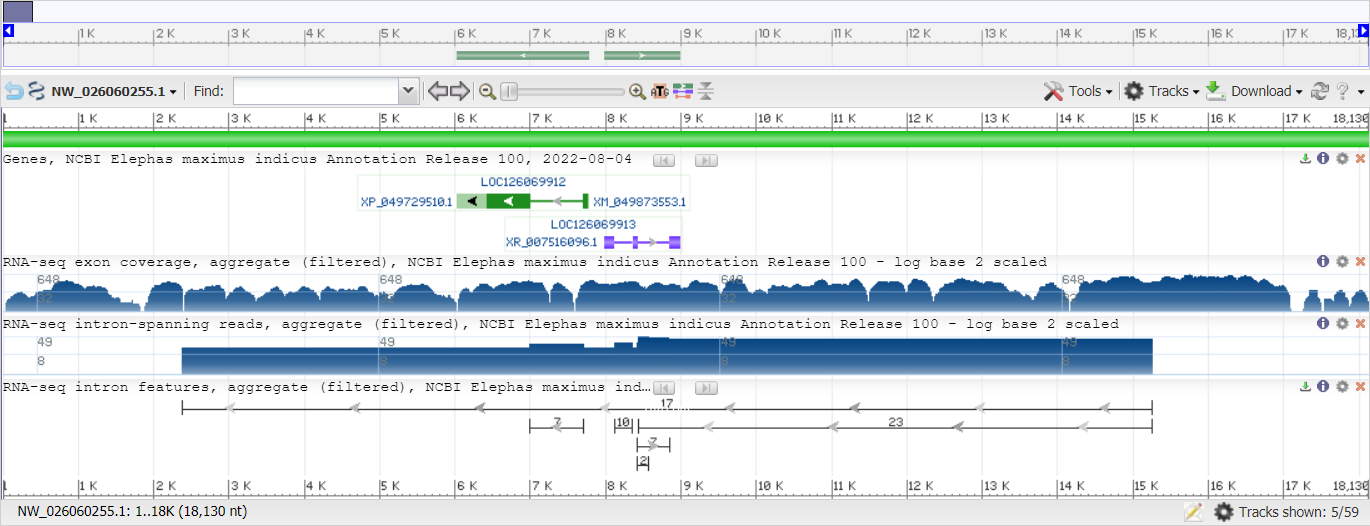
На картинке, демонстрирующей локализацию генов, тёмно-зелёными прямоугольниками показаны экзоны гена, кодирующего некоторый неохарактеризованный белок. Светло-зелёный прямоугольник соответствует нетранслируемой области mRNA этого гена. Ген находится на ''-'' цепи ДНК.
Напротив, ген, соответствующий некодирующей РНК, располагается на ''+'' цепи ДНК.
Соответственно, на контиге найдено два гена. Один из них кодирует белковый продукт и содержит два экзона.
Повторюсь, что мной отобран фрагмент, составленный именно из этих двух экзонов (на ''-'' цепи ДНК).
Ссылка на последовательность фрагмента контига в FASTA-формате
Далее я запустил несколько разновидностей алгоритма BLAST для данной посследовательности, выбрав для исключения таксон Afrotheria (таксон, включающий в себя хоботных и ближайших родственников: даманов, трубкозубов, прыгунчиковых и так далее) (taxid:7711). Я решил взять таксон поменьше, чтобы не было результатов без находок:
1. blastn: Найдены 577 находок. Почти все они ''PREDICTED''. Встречаются и отдельные хромосомы из других геномных сборок (например, из сборки Lutra lutra, то есть речной выдры).
2. megablast: Найдена всего 3 находки: две из Equus asinus (Домашний осёл) и одна из Ceratotherium simum simum (Южный белый носорог). Это также неохарактеризованные белки.
3. blastx: Найдено 1246 находки. Практически все находки - неохарактеризованные/гипотетические белки. Но также находится сходство с FAM170B (как у человека, так и у других представителей). Как оказалось FAMB170 - акросомальный белок, играющий важную роль в оплодотворении млекопитающих. В частности, показано, что его ингибирование приводит к утере фертильности мышей [1]. Забавно, что данные неописанный белок имеет некое сходство с FAMB170.
4. tblastx: К сожалению, tblastx отказывается работать, выводя информацию о том, что я перегружаю сервера NCBI...
Blastn - алгоритм BLAST, который принимает на вход нуклеотидную последовательность и ищет схожие последовательности из некоторой нуклеотидной базы данных. Незаменим, когда необходимо найти возможные гомологи некой (найденной где-то, например) нуклеотидной последовательности (как ДНК, так и РНК). Megablast - подтип blastn, который применяется для поиска очень сходных последовательностей. Может применяться, например, для поиска ошибок секвенирования. Blastx принимает на вход нуклеотидную последовательность, транслирует её в белок и ищет в белковой базе данных схожие белки. Если у вас есть экзонная (непосредственно кодирующая) последовательность ДНК, то можно сразу найти возможные гомологи соответствующего белка (как, например, в моём случае: я кодирующая часть гена неохарактеризованного белка дал на вход blastx и попробовал найти схожие белки, прийдя к неожиданному белку FAM170B). Алгоритм tblastx берёт нуклеотидный запрос, транслирует его в белок и ищет схожие в нуклеотидной базе данных, транслированной в шести рамках считывания. Таким образом лучше искать схожие белки по эукариотическому гену (с интронами).
Задание №2: Гомологи бактериальных рибосомальных РНК в геноме эукариота.
Первым делом я скачал геном своего организма и проиндексировал его с помощью команды makeblastdb. Ниже приведена соответствующая команда:
makeblastdb -in GCF_024166365.1_mEleMax1_primary_haplotype_genomic.fna -dbtype nucl
Далее я скачать файл с последовательностями рРНК Escherichia coli и провёл локальный поиск BLAST отдельно с 16sRNA и 23sRNA. Ниже приведены команды:
blastn -task blastn -query 16s_RNA.fasta -db GCF_024166365.1_mEleMax1_primary_haplotype_genomic.fna -evalue 0.05 -word_size 11 -out blast_16S.out -outfmt 7
blastn -task blastn -query 23s_RNA.fasta -db GCF_024166365.1_mEleMax1_primary_haplotype_genomic.fna -out blast_23S.out -outfmt 7
1. Выводы соответствующих команд: blast_16S.out, blast_23S.out.
Видно, что круг найденных последовательностей не слишком разнообразен: в первом случае запрос был выровнен с рядом участков в 19 (NC_064837.1) и 11 (NC_064829.1) хромосомах слона, представляющих из себя участки кластеров генов 18S-рРНК. При этом довольно удивило то, что все выравнивания оказались чрезвычайно короткими и соответствовали (примерно) начальным фрагментам 18S-рРНК слона. Кажется, сходство между 16S-рРНК и 18S-рРНК не столь значительно, как мне казалось. Последние две строки вывода выровнялись с неаннотированными участками 15 (NC_064832.1) и 4 (NC_064823.1) хромосомам.
В случае с 23S-рРНК картина оказалась схожей: точной так же были найдены участки сходства на 19 и 11 хромосомах, которые соответствовали уже 28S-рРНК. Остальные участки сходства приходились на 1, 15, 5 хромосомы, на X и даже на Y хромосомы (перечислены не все), однако все эти участки оказались неаннотированными.
2. Я решил использовать blastn, поскольку на input мы подаём нуклеотидную последовательность РНК (а не белка (!): тогда бы мы использовали blastx или tblastx...) с целью найти гомологичные последовательности в нуклеотидной базе данных (сформированной из fasta-формата генома выбранного эукариота). Я использовал значение параметра word size по умолчанию (эксперимент c word size, равным 4, не удался, поскольку время работы blastn значительно увеличилось), но решил поставить порог на E-value в 0.05, чтобы в качестве результатов не попадались совсем ''странные'' случаи.
3. Найдено около 16 гомологов 16s-rRNA, которые являются участками генов 18s-rRNA, а также около 70 гомологов 23s-rRNA, являющиеся участками генов 28s-rRNA (остальные участки оказались неаннотированными). Как известно, 18s-rRNA - это рибосомальная РНК, входящая в состав малой субъединицы (40S) эукариотической рибосомы (80S). В то время как 28S-rRNA - это рибосомальная РНК, которая входит в состав большой субъединицы эукариотической рибосомы. Не удивительно, что именно они оказались находками при работе blastn, поскольку известно, что 16s-rRNA гомологична 18s-rRNA, а 23s-rRNA гомологична 28s-rRNA. В то же время меня удивило то, что степень их сходства оказалась не столь высокой, как я себе представлял...
4. У каких-то участков аннотация была, у каких-то не было. Подробнее описано в первом пункте.
Ссылки.
1. Li Y, Lin S, Luo M, Guo H, Chen J, Ma Q, Gu Y, Jiang Z, Gui Y. FAM170B, a novel acrosomal protein involved in fertilization in mice. Mol Reprod Dev. 2015 Oct;82(10):787-96. doi: 10.1002/mrd.22523. Epub 2015 Sep 30. PMID: 26179146.
Кирилл Кузенков, студент второго курса ФББ